Поиск мотивов среди гомологов белка YojM_BACSU
На сервере kodomo программой ememe был осуществлён поиск мотивов среди гомологов белка YojM_BACSU, взятых из репрезентативной выборки с практикума 8. Результат работы программы представлен на отдельной странице.
В связи с ограничением поиска в 3 мотива, программой ememe и было найдено 3 мотива. Информация о них содержится в таблице 1.
| № | Число последова- тельностей в нём | Длина мотива | E-value | LOGO |
|---|---|---|---|---|
| 1 | 56 (все) | 21 | 4,4*10-496 | 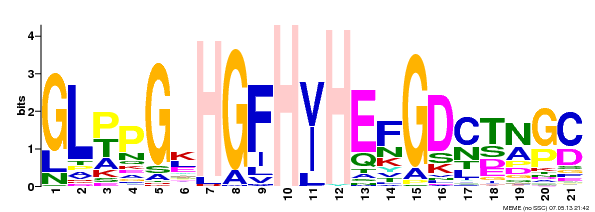
|
| 2 | 56 (все) | 21 | 8.0*10-477 | 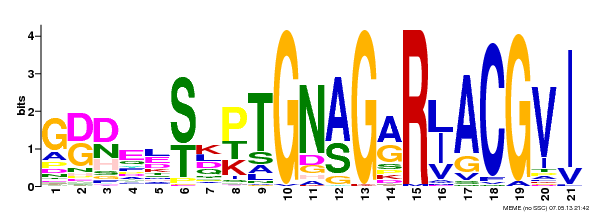
|
| 3 | 47 | 22 | 1.1*10-441 | 
|
Сравнение блоков, найденных MEME, c полным выравниванием, выданным muscle
В целом, выравнивания MEME и Muscle различаюся между собой (см. проект JalView  ). Подробнее на конкретных мотивах:
). Подробнее на конкретных мотивах:
1. Сравнение первого мотива и глобального выравнивания продемонстрировано на рисунке 1.
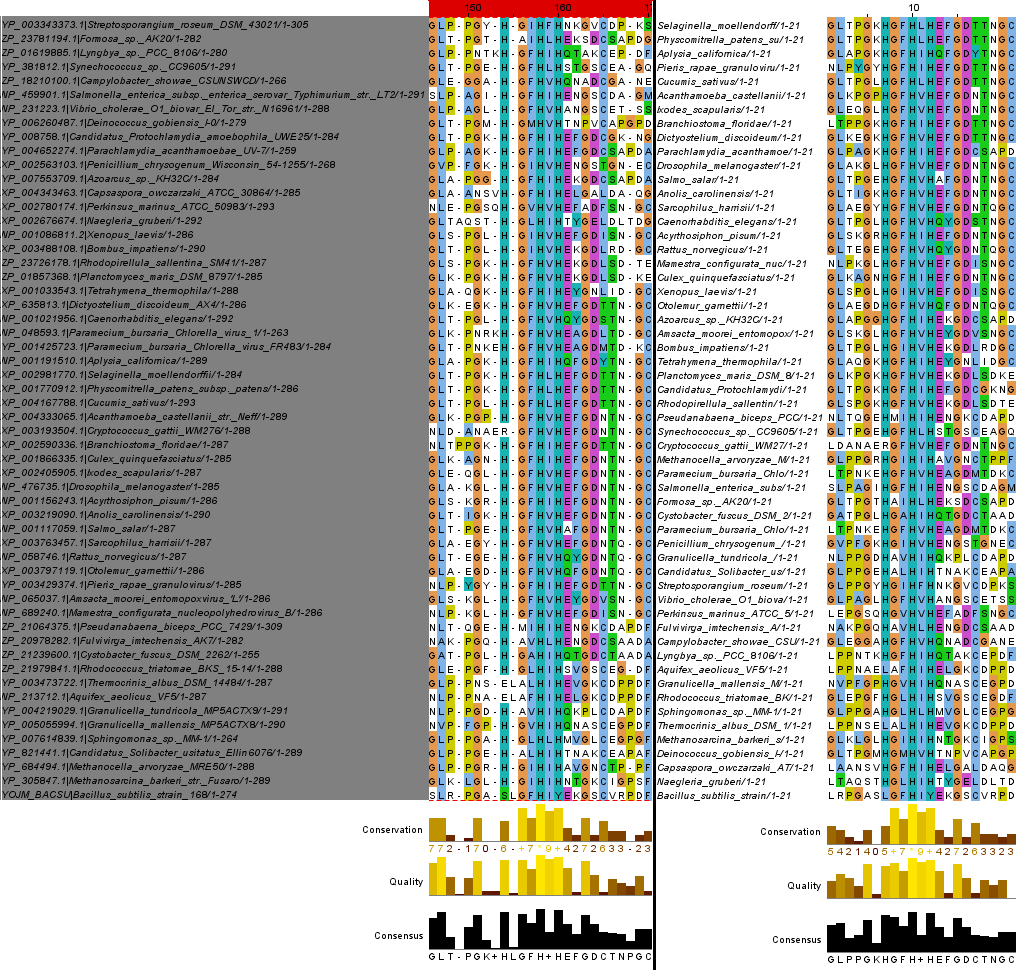 Рисунок 1. Сравнение глобального (слева) и локального (справа) выравниваний (мотив №1)
Рисунок 1. Сравнение глобального (слева) и локального (справа) выравниваний (мотив №1)2. Сравнение второго блока и общего выравнивания осуществлялось с помощью рисунка 2.
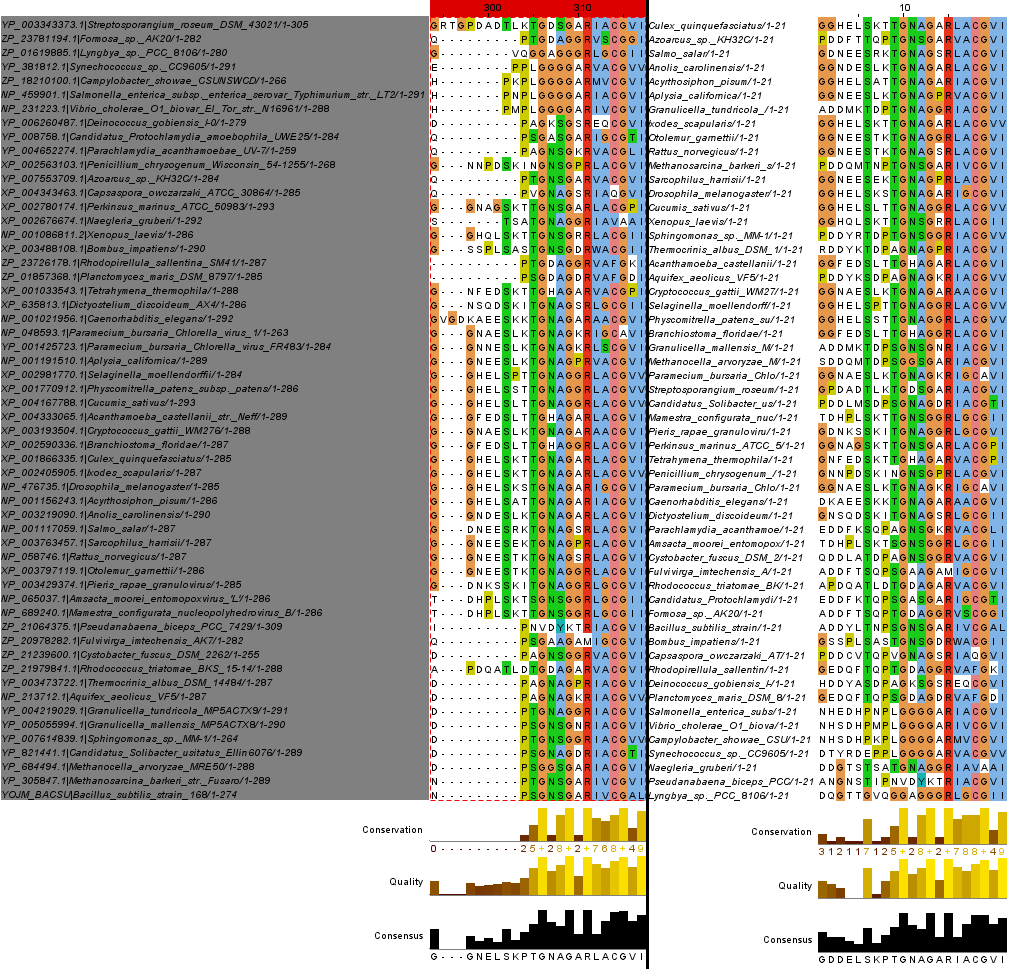 Рисунок 2. Сравнение глобального (слева) и локального (справа) выравниваний (мотив №2)
Рисунок 2. Сравнение глобального (слева) и локального (справа) выравниваний (мотив №2)3. Сравнение третьего мотива и глобального выравнивания произведено посредством рисунка 3.
 Рисунок 3. Сравнение глобального (слева) и локального (справа) выравниваний (мотив №3)
Рисунок 3. Сравнение глобального (слева) и локального (справа) выравниваний (мотив №3)Поиск найденных мотивов в других последовательностях
На сервере kodomo программой emast был проведён поиск мотивов, найденных MEME, в последовательностях выравнивания домена Pfam PF00080 белка YojM_BACSU (см. Описание домена SOD_Cu (PF00080)); параметры устанавливались по умолчанию. С результатом её работы можно ознакомиться на отдельной странице.
Краткое описание результатов:
- Всего последовательностей во входном файле - 127
- Встречаемость мотивов в последовательностях указана в таблице 2
- Все мотивы нашлись в 87 последовательностях
| Номер мотива | Последовательность | Количество последовательностей, в которых содержится |
|---|---|---|
| 1 | GLPPGYHGFHIHEFGDCTNPC | 119 (93.7%) |
| 2 | GDDYESQPTGNAGARWACGVI | 109 (85.8%) |
| 3 | FNPFNKKHGYPWDEERHWGDLG | 92 (72.4%) |
Выравнивание домена PF00080 полностью соответсвует найденным ранее мотивам (визуально). Единственным замечанием может быть лишь то, что доля аргинина в третьем мотиве уменьшается с ростом количества последовательностей (и не окрашивается в JalView в выравнивании Pfam). Ознакомиться с выравниваниями можно с помощью проекта JalView  .
.