Построение дерева по нуклеотидным последовательностям
Построение филогенетического дерева проводилось по последовательностям РНК малой субъединицы рибосомы (16S rRNA). Для этого были найдены записи EMBL геномов бактерий из таблицы. Затем из каждой записи вырезаны последовательности 16S rRNA (одна для каждой бактерии), которые были записаны в fasta-файл (см. табл. 1). После этого полученные последовательности были выровнены с помощью muscle на сервере EBI. Выравнивание представлено в проекте Jalview и fasta.
| Название бактерии | Мнемоника | AC записи EMBL | Координаты гена | Направление цепи |
|---|---|---|---|---|
| Bacillus anthracis | BACAN | AE016879 | 9335..10841 | direct |
| Bacillus subtilis | BACSU | AL009126 | 30279..31832 | direct |
| Clostridium tetani | CLOTE | AE015927 | 8715..10223 | complement |
| Geobacillus kaustophilus | GEOKA | BA000043 | 10421..11973 | direct |
| Lactobacillus acidophilus | LACAC | CP000033 | 59255..60826 | direct |
| Listeria monocytogenes | LISMO | AL591980 | 96266..97811 | complement |
| Staphylococcus epidermidis | STAES | AE015929 | 1598006..1599559 | complement |
| Streptococcus pneumoniae | STRPN | CP000936 | 15372..16805 | direct |
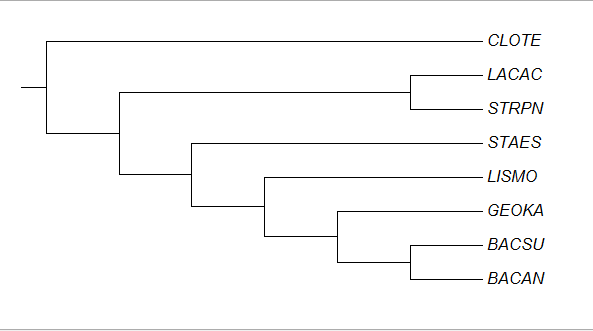
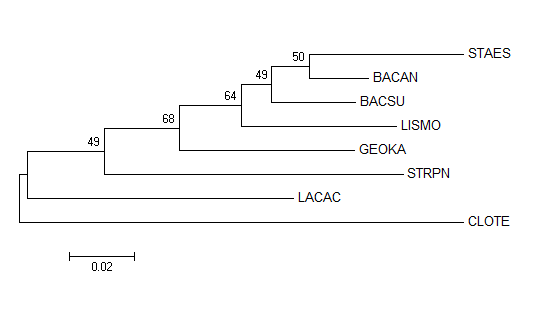
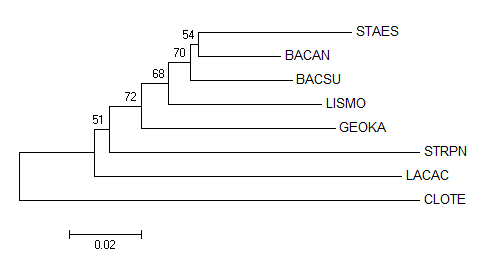
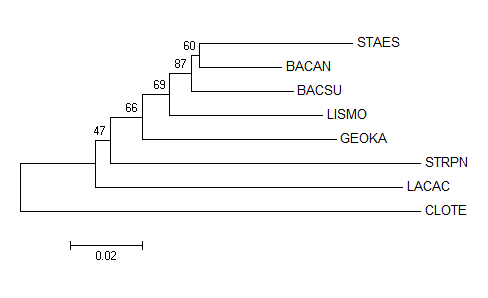
Выравнивание последовательностей 16S rRNA было импортировано в программу MEGA, в которой были реконструированы филогенетические деревья методами максимального правдоподобия, минимальной эволюции и Neighbor-Joining. Деревья приведены в таблице 2.
| Истинное дерево | Maximum Likelihood |
|---|---|
| 
|
| Neighbor-Joining | Minimum Evolution |
| 
|
Как видно из таблицы выше все метода привели к одной и той же топологии дерева, но не совсем правильной. Из достоинств дерева следует отметить то, что чётко разделены классы клострилий и бацилл. Подробное сравнение дерева с истинным представлено в таблице 3.
| Ветвь | Правильное дерево | Реконструкция |
|---|---|---|
| {LACAC, STRPN} vs {others} | + | - |
| {CLOTE, LACAC, STRPN} vs {others} | + | + |
| {BACAN, BACSU} vs {others} | + | - |
| {BACAN, BACSU, GEOKA} vs {others} | + | - |
| {BACAN, BACSU, GEOKA, LISMO} vs {others} | + | - |
| {STAES, BACAN} vs {others} | - | + |
| {STAES, BACAN, BACSU} vs {others} | - | + |
| {STAES, BACAN, BACSU, LISMO} vs {others} | - | + |
| {CLOTE, LACAC} vs {others} | - | + |
| Верных ветвей | 5 | 1 |
| Ошибок | 0 | 4 |
В отличие от реконструкции по белкам, результатом проведённой работы является всего 1 топология дерева, и то неправильная. Анализ по белкам, видимо, способен давать более точную филогению.
Построение и анализ дерева, содержащего паралоги
CLPX_BacSu - это АТФ связывающая субъединица Clpx АТФ-зависимой протеазы Clp бактерии Bacillus subtilis; относится к шаперонам семейства ClpX. Чтобы найти гомологов этого белка был произведён поиск бласт по записям банка UniProt бактерий из таблицы с помощью файла proteo.fasta:
makeblastdb -in proteo.fasta -dbtype prot
blastp -query clpx_bacsu.fasta -db proteo.fasta -out blastp7.out -evalue 0.001 -outfmt 7
После этого были выбраны только белки из рассматриваемых бактерий (получилось 35 шт), их ID представлены в файле с ID. Далее с помощью python-скрипта из proteo.fasta были получены все последовательности белков c полученными ID и записаны в файл с последовательностями. Они затем были выровнены программой muscle на сервере EBI; выравнивание доступно в fasta и в проекте Jalview. Полученное выравнивание было импортировано в MEGA, с помощью которой построено филогенетическое дерево методом Neighbor Joining (см. рис. 1).

Рис.1 Филогенетическое дерево гомологов белка CLPX_BACSU.
Также для просмотра доступно дерево, полученное с помощью bootstrap-анализа. Если считать дерево правильным, на нём можно указать паралоги (гомологичные белки из одного организма, образуются удвоением гена):
- B0AWL5_BACAN, C2NBK7_BACAN, B1SHF4_BACAN
- Q891B9_CLOTE, Q899H3_CLOTE
- Q899V4_CLOTE, Q8990L5_CLOTE
а также ортологи (гомологичные белки из разных организмов, образуются в результате видообразования):
- Группа белков CLPX
- Группа белков HSLU
С помощью полученного дерева легко проследить эволюционные события. Так, в месте, обозначенном красным ромбом произошла дупликация гена, в результате чего образовались белки CLPX и HSLU. А затем проиходил процесс разделения путей эволюции белков в результате видообразования, что привело к образованию ортологов, выделенных синем.