Отчёт по практикуму 9
Поиск в UniProt белков заданной архитектуры
В первом задании требуется выбрать два домена Pfam и найти в UniProt составленные из них белки.
Выбранные домены — PF00999, PF00359 (приведены AC). Вот данные о доменах:
- PF00999:
- ID — Na_H_Exchanger;
- название — Sodium/hydrogen exchanger family (домен семейства натриево-водородных обменников);
- число последовательностей среди бактерий — 23900 (Pfam).
- PF00359:
- ID — PTS_EIIA_2;
- название — Phosphoenolpyruvate-dependent sugar phosphotransferase system (фосфоенолпируватзависимая сахарфосфотрансфераза);
- число последовательностей среди бактерий — 12741 (Pfam);
Поиск по UniProt был проведён со следующим запросом:
taxonomy:"Bacteria [2]" database:(type:pfam pf00999) database:(type:pfam pf00359)
Pfam приводит 75 белков, составленных только из данных двух доменов. Здесь же нашлось 403 белка, из которых 307 не содержали посторонних доменов. Обработанные результаты можно скачать. Это таблица в формате xlsx, на втором листе которой построена гистограмма длин белков с исследуемой доменной организацией. Белки с посторонними доменами выделены золотистым цветом, а названия, содержащие Fragment, — красным.
Гистограмма длин белков приведена на рис. 1. Видно, что большинство белков имеют длину в промежутке 500..799 а. о. (267 шт., или 87 %).
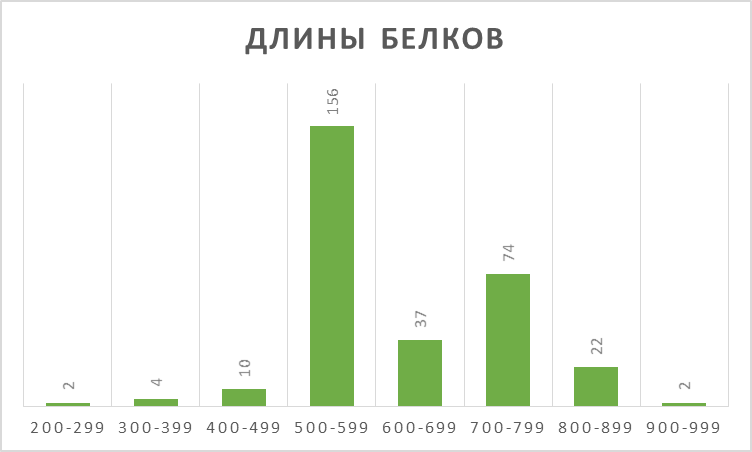
Гистограмма длин белков из UniProt, имеющих в составе только домены PF00999 и PF00359.
Также была создана выборка из 56 последовательностей характерной длины (см. поле Selected в таблице). Последовательности отобранных белков можно скачать в формате fasta.
Построение HMM-профиля по выбранной архитектуре и проверка его работы
Полученную выше выборку белков характерной длины можно использовать для построения HMM-профиля, а им впоследствии искать новые белки с такой архитектурой.
Загрузим последовательности белков в JalView и выровняем программой MUSCLE. После этого обработаем выравнивание в соответствии с рекомендациями из презентации: уберём малоконсервативные участки с концов, избавимся от последовательностей с явными вставками и делециями. Итоговое выравнивание можно скачать. Там остался 41 белок.
Построим и калибруем профиль. Команды, которые для этого потребуются:
- hmm2build arch.hmm align_cut.fasta
- hmm2calibrate arch.hmm
Доступен и файл arch.hmm, получившийся на этом этапе. Для дальнейшей проверки профиля скачаем последовательности всех белков с доменом PF00359 в составе из UniProt. Выбран этот домен, так как белков с ним меньше: всего 73023 против 138903 для PF00999. При задании команды поиска не был специально задан порог на E-value, но потом берём результаты только до 0,01. Вот сама команда:
- hmm2search arch.hmm all_pf00359.fasta.gz
Результаты поиска с E-value не выше 0,01 импортируем в MS Excel и обрабатываем (таблицу можно скачать, см. лист hmm_found; ступеньки Score и кривая ROC там же, вынесены на отдельный лист charts).
В результате вычисления F1 можно заключить, что оптимальным по соотношению чувствительность-точность (PPV) порогом был бы вес -42,9 (берём все находки с таким или большим весом). В таблице 1 указаны характеристики получившегося теста.
| Вердикт | С нашей архитектурой | С иной архитектурой |
|---|---|---|
| Подтверждено | 279 | 11 |
| Отвергнуто | 28 | 2129 |
Чувствительность составила 90,9 %, а специфичность — 99,5 %.
Интересно, что порог, выбранный по F1, попал перед началом более пологого участка на диаграмме score.