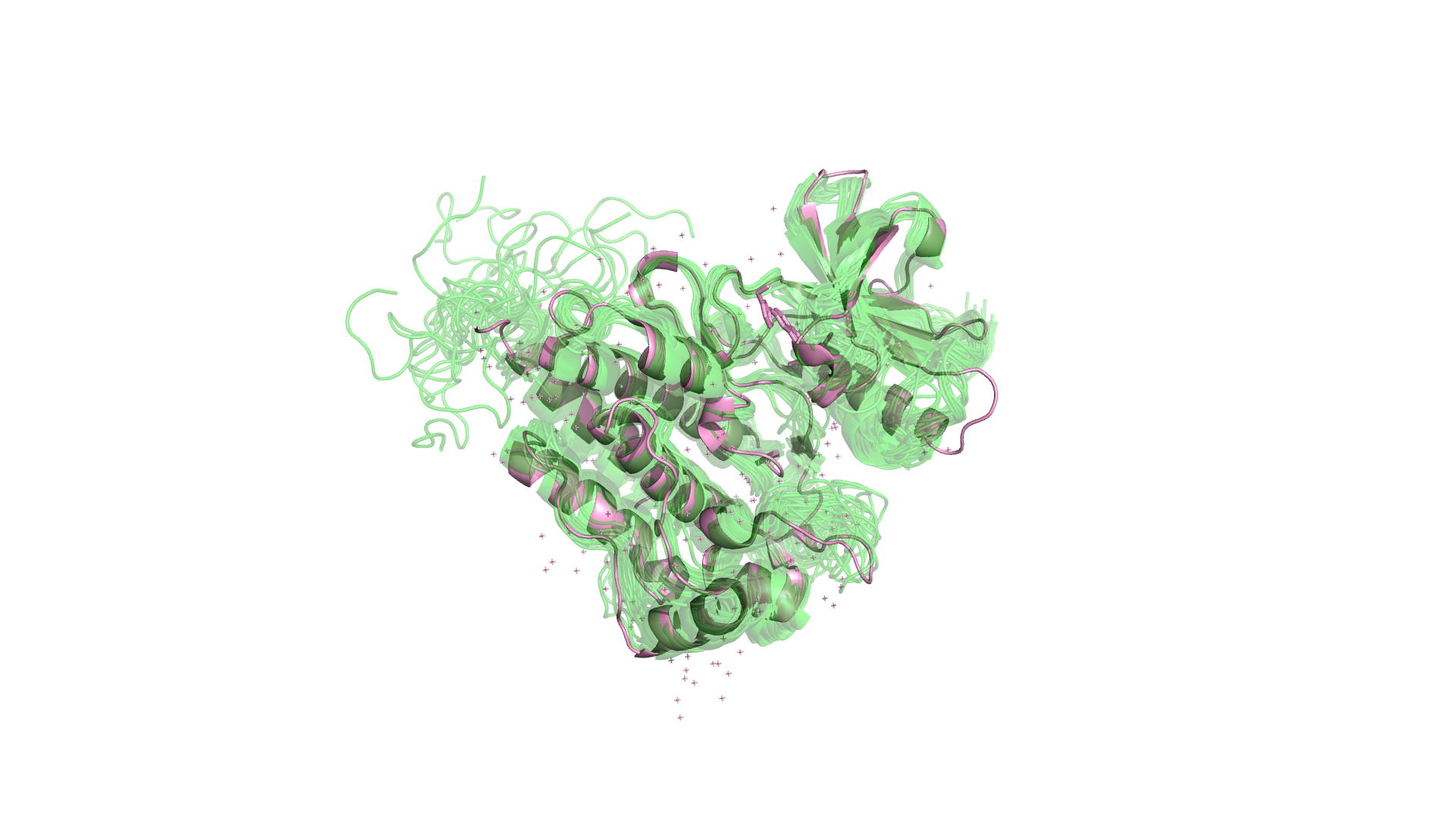
Рисунок 1.1 Сравнение 6XR7(зеленый) и 2G2H(розовый) на уровне структур.
PDBID:6XR7,2G2H
В данном практикуме сравниваются структуры одного и того же белка, полученные с помощью ЯМР и РСА. Структура 6XR7 получена с помощью ЯМР и включает 20 моделей. Структура 2G2H получена с помощью РСА и имеет разрешение 2.00 Å. Сравним структуры, полученные разными методами.

Рисунок 1.1 Сравнение 6XR7(зеленый) и 2G2H(розовый) на уровне структур.
Хочется отметить, что расположение крупных элемнтов(альфа-спиралей) в структурах совпадает. Однако, вода присутствует только в структуре, полученной с помощью РСА, поскольку в ЯМР используется дейтерированный растворитель. Также в полученной с помощью ЯМР структуре отсутствует молекула низкомолекулярного лиганда, однако это может быть связанно не только с особенностями методов. Некоторые фрагменты петель в структуре, полученной с помощью РСА, отображаются в виде необычных коротких бета-листов и альфа-спиралей, отсутствующих в ЯМР-структуре. Наиболее значительные отличия в двух структурах наблюдаются на их концах и в петлях, где подвижность остатков максимальна. Более того, в ЯМР структуре на концах содержатся остатки(4 с одного конца и 11 с другого), положение которых, видимо, не смогли установить в РСА-структуре. Теперь рассмотрим некоторые различия структур на уровне остатков.
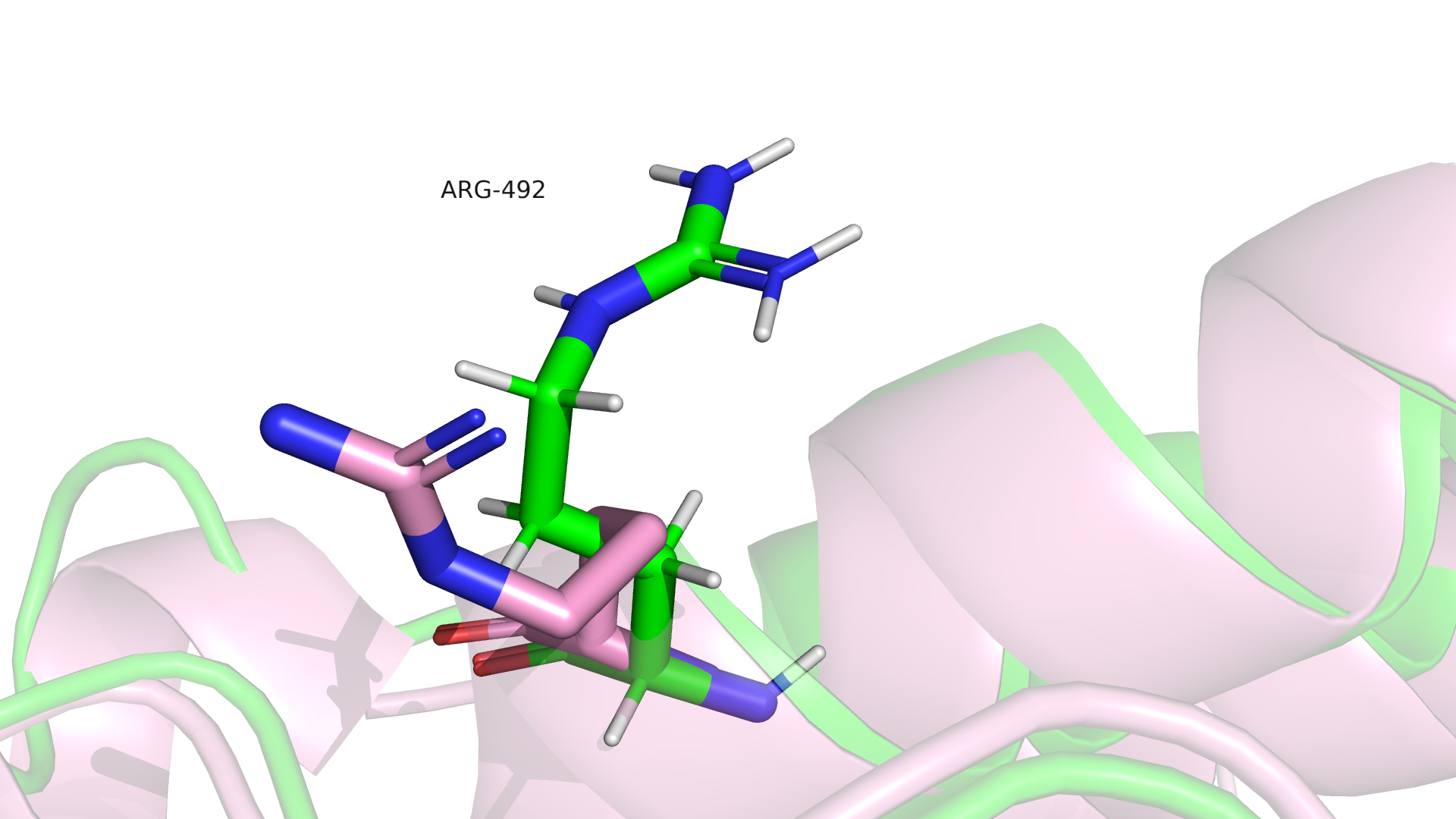
Рисунок 1.2 Сравнение 6XR7(зеленый) и 2G2H(розовый) на уровне остатков.
Заметно, что протоны присутствуют на структуре 6XR7, полученной с помощью ЯМР. Разрешения РСА-стурктуры в 2.00 Å не хватает, чтобы увидеть отдельные протоны. Так же можно отметить, что ориентация радикала Arg492 в структурах значительно отличается.
PDB структура, получаемая с помощью ЯМР состоит из набора моделей, которые по мнению авторов более всего похожи на настоящую структуру. Вариация отдельных элементов структуры может говорить о их высокой подвижности. Вычислим RMSF для ЯМР структуры и средние B-факторы струкры, полученной с помощью РСА. Построив график их зависмости можно попытаться сделать вывод о том, насколько корректно рассматривать ансамбль моделей ЯМР-структуры, как отражение подвижности белка.

Рисунок 2. Scatter plot зависимости B-фактора от RMSF.
Рассмотрим график. В целом видно увелечение B-фактора с увеличением RMSF. Однако, большое число точек имеет высокий B-фактор и относительно низкое значение RMSF. Как мне кажется, это может быть связано с качеством РСА структуры. В целом, ансамбль моделей ЯМР-структуры, пусть и с некоторыми неточностями, но отображает подвижность белка.
В данном задании были выбраны три водородные связи, детали которых далее представлены в Таблице 1 и на рисунках 3.1-3.6.
Таблица 1. Параметры водородных связей.

В случае водородной связи между атомами остова в ядре белка можно обратить внимание на то, что она сохраняется во всех моделях структуры из ЯМР. Это можно связать с тем, что альфа-спираль является устойчивой вторичной структурой, подвижность остова в ней ограничена. Сохранение водородной связи представлено на рисунке 3.2.
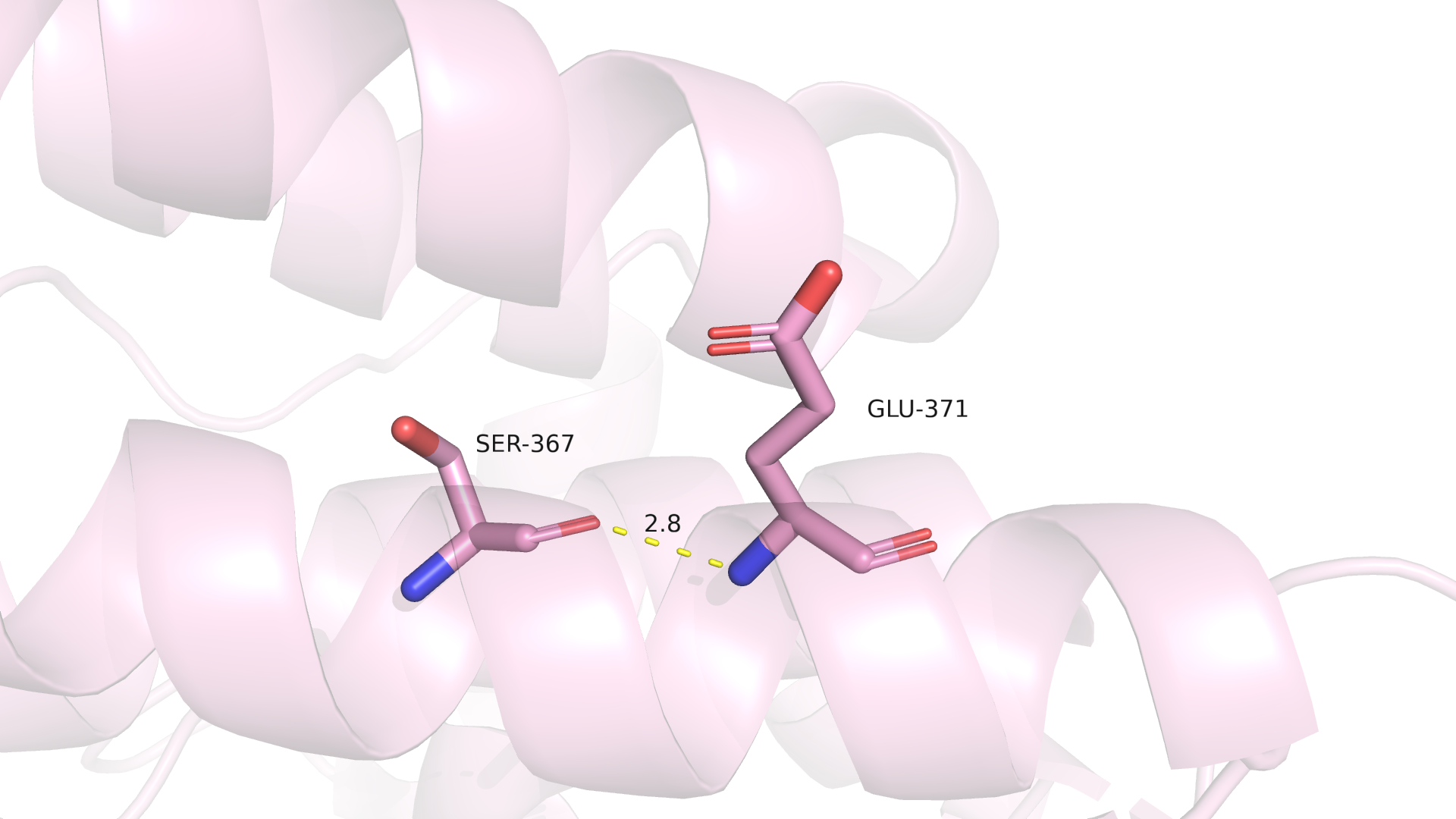
Рисунок 3.1 Связь атомов остова Ser367-Glu371 в РСА-структуре.

Рисунок 3.2 Связь атомов остова Ser367-Glu371 в ЯМР-структуре.
Теперь рассмотрим связь между атомами боковых цепей в ядре белка. Только 10% моделей ЯМР структуры сохраняют данную связь. Это можно связать с тем, что боковая цепь гораздо более подвижна даже в ядре белка. На рисунке 3.4 приведен пример утраты связи(гистидин развернулcя в другую сторону).

Рисунок 3.3 Связь атомов боковой цепи Ser367-His509 в РСА-структуре.
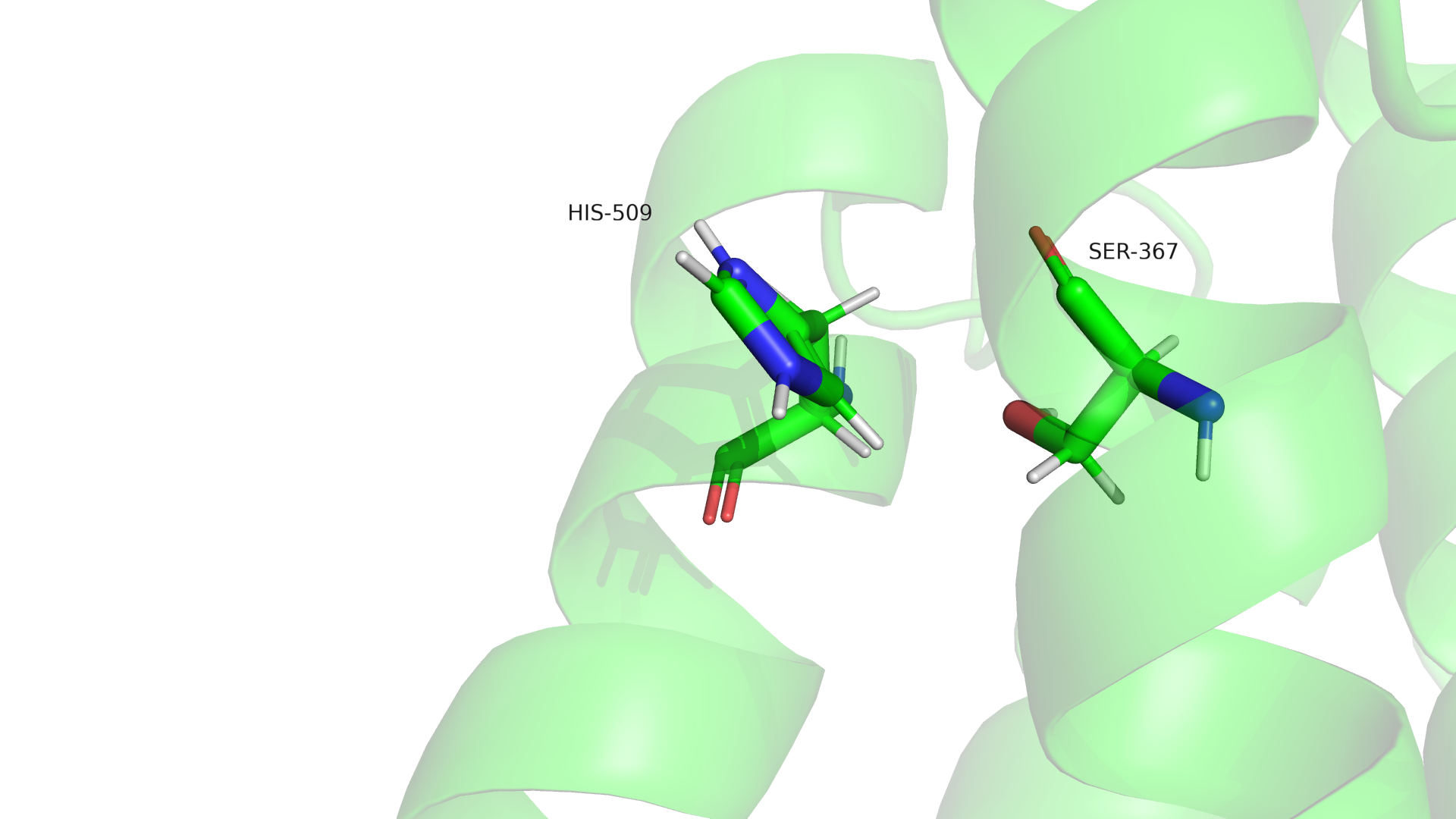
Рисунок 3.4 Утрата связи Ser367-His509 в ЯМР-структуре.
В петле, выходящей на поверхность глобулы, связь между атомами остова сохраняется во всех моделях ЯМР структуры. Как мне кажется, это скорее исключение из правила. Ожидаемой была бы высокая подвижность этой петли и утрата связи в части моделей ЯМР структуры. Однака, конкретно эта петля оказалась достаточно стабильной. Также, как мне кажется, на результат повлияло и то, что мы рассматривали связь между атомами остова, которые, как правило, менее подвижны, чем атомы боковой цепи.
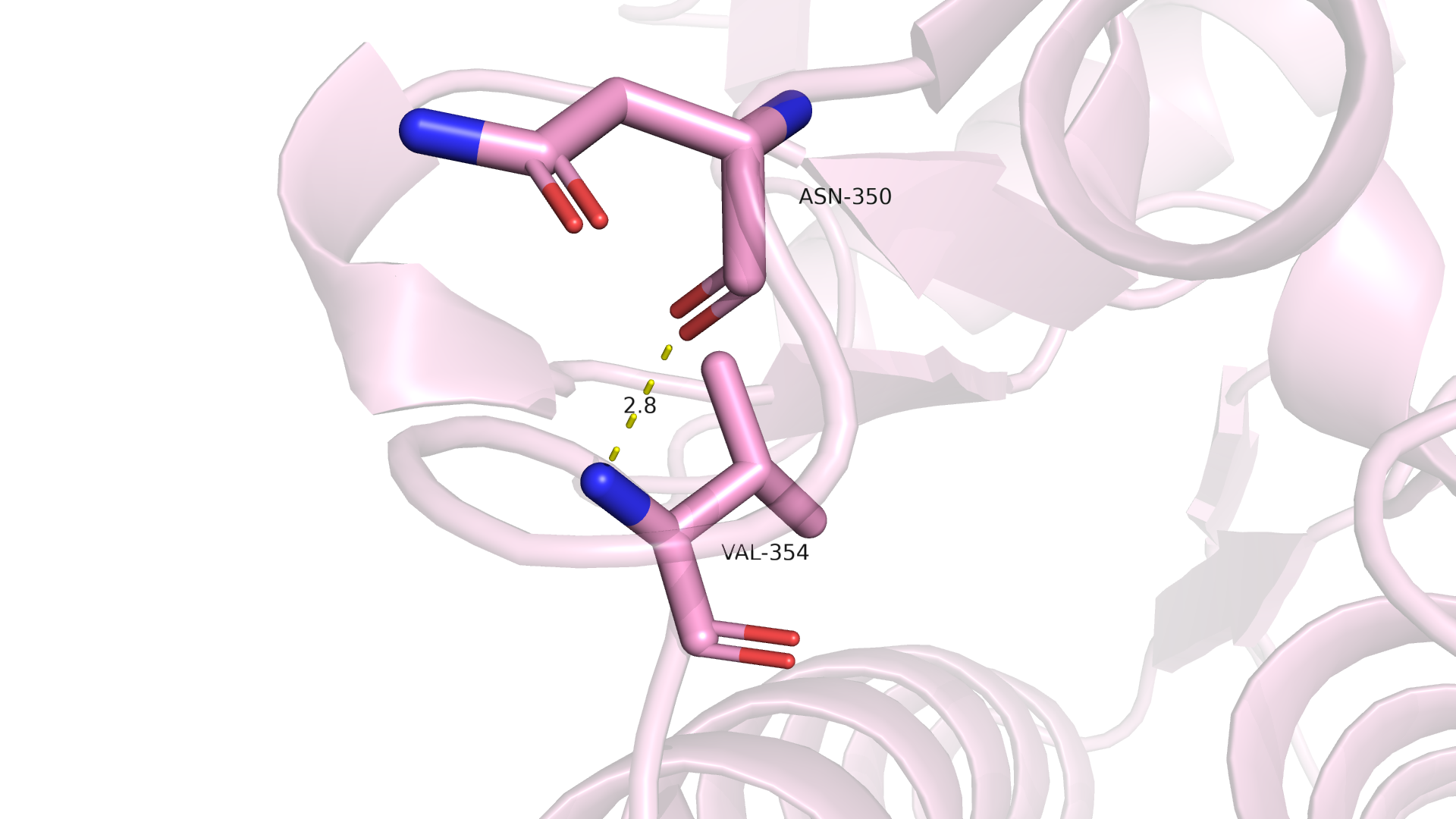
Рисунок 3.5 Связь атомов остова Asn350-Val354 в РСА-структуре.
© Simon Konnov 2017-2020