Навигация по сайту: |
Сборка генома de novoПодготовка чтенийОтрезаем адаптеры: java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 ../init/SRR4240359.fastq adapters_removed.fastq ILLUMINACLIP:../adapters/adapters.fasta:2:7:7 Результат: осталось 13502036 (99,59%) ридов, 55902 (0,41%) отфильтровались. Избавляемся от коротких и плохих чтений: java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 ../remove_adapters/adapters_removed.fastq trimmed.fastq TRAILING:20 MINLEN:30 Результат: выжило 12549379 (92,94%) ридов, удалено 952657 (7,06%). Контиги, построенные с помощью velvetgЧтобы получить "типичное" покрытие нужно сначала избавиться от артефактов: для k=25 9389 контигов из 10168 имеют длину менее 100bp и не очень информативны, для k=29 аналогичный показатель составил 1818 контигов из 2028. В качестве покрытия был взять показатель short1_cov.
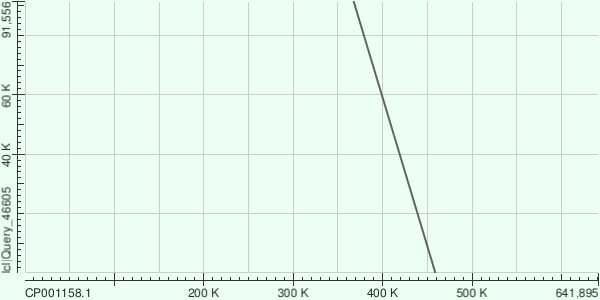
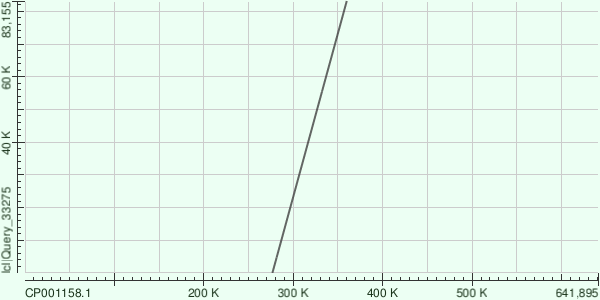
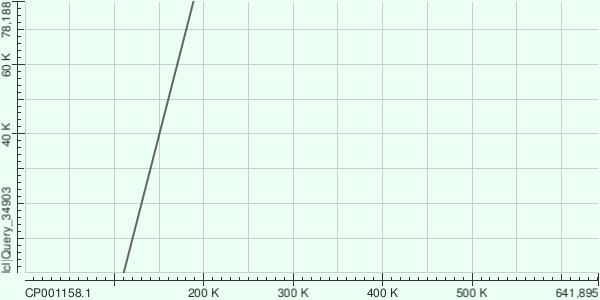
В качестве типичного покрытия наверное было бы разумным взять медианное покрытие. Относительно него все три самых длинных контига являются аномально покрытыми: ~90 для k=25 и ~54 для k=29. Сравнивая результаты для разных k, можно сделать вывод, что velvetg очень чувствителен к выбору длины слова: в одном случае получилось много коротких контигов с чуть лучшим покрытием(k=25), в другом получились большие контиги с чуть более слабым покрытием(k=29). Анализ трех самых длинных контигов для k=29Последовательности контигов можно найти на kodomo по адресу /nfs/srv/databases/ngs/spush/pr14/ Как я случайно сделал задание не для того геномаНе увидев сразу на какой геном нужно накладывать контиг, я начал искать сборку генома организма, указанного в проекте секвенирования(SRR4240359). Перешел по "scientific name" организма. На странице организма (ссылка) ->Portal->Assembly->Chromosomes. Получил AC CP001158.1. На него blast-ом положил наши контиги и получил очень красивые картинки, в которых контиги легли линейно(без повторов):
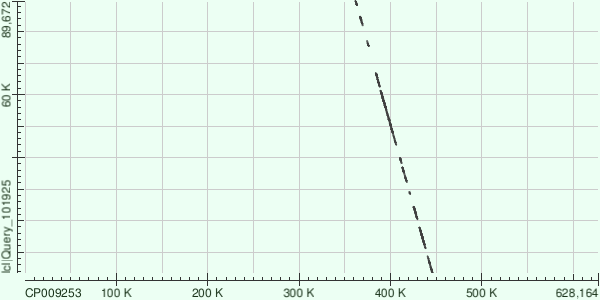
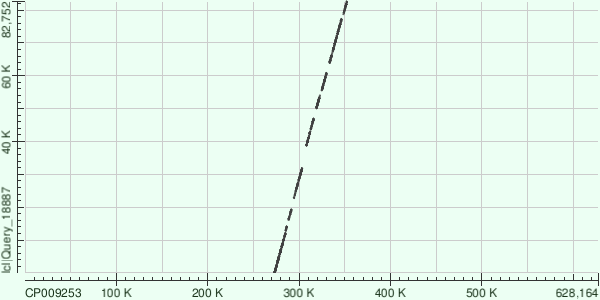
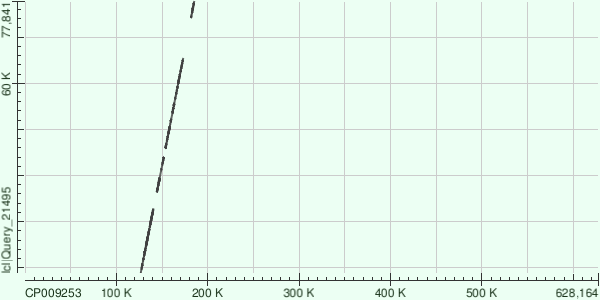
Как я сделал то, что требовалось в заданииГеном CP001158.1, найденный мной, и CP009253, предложенный нам в задании, являются геномами разных штаммов одной бактерии и, возможно, отличаются не сильно. Несмотря на то что контиг выравнялся с геномом в виде отдельных фрагментов, образовавшаяся картина очень напоминает полученную выше. Контиги все так же ложатся без повторов, линейно. Ниже привожу некоторую общую статистику по всем выравненным участкам, полученную из hit table.
Полученные карты локального сходства свидетельствуют о том, что у штамма с геномом CP009253 есть множественные индели, отличающие его от штамма с геномом CP001158.1. |