
1. Глобальное парное выравнивание гомологичных белков
В ходе работы было проведено выравнивание в программе needle с параметрами по умолчанию трёх пар белков Escherichia coli K12 и Bacillus subtilis (strain 168) с одинаковой мнемоникой функции. Для выбора пар на сайте Uniprot были найдены и скачаны списки идентификаторов, оканчивающихся на _ECOLI и на _BASCU соответсвенно, всех аннотированных записей. Далее при помощи инструментов Google Sheets были сопоставлены пары с одинаковыми мнемониками функции. Из них были выбраны три, характеристики выравниваний которых представлены в Таблице 1. Для осуществления выравнивания в терминаде использовалась команда needle sw:*_ecoli sw:*_bacsu name.needle -auto, где * заменяет начало ID.
| Protein Name | ID 1 | ID 2 | Score | Identity, % | Similarity, % | Gaps | Indels |
|---|---|---|---|---|---|---|---|
| Quinolinate synthase A | NADA_ECOLI | NADA_BACSU | 319.0 | 23.5 | 42.1 | 26.9 | 12 |
| Nicotinate-nucleotide pyrophosphorylase [carboxylating] | NADB_ecoli | NADB_BACSU | 761.0 | 35.6 | 51.5 | 10.4 | 8 |
| L-aspartate oxidase | NADC_ECOLI | NADC_BACSU | 489.5 | 35.1 | 51.1 | 12.8 | 6 |
Стоит отметить, что у Bacillus subtilis (strain 168) рекомендуемое название Probable nicotinate-nucleotide pyrophosphorylase [carboxylating].
2. Локальное парное выравнивание гомологичных белков
Выполнение этой части практикума отличается от первой лишь использованием программы water (команда water sw:*_ecoli sw:*_bacsu name.water -auto) и наличием двух дополнительных характеристик в Таблице 2.
| Protein Name | ID 1 | ID 2 | Score | Identity, % | Similarity, % | Gaps | Indels | Coverage 1 | Coverage 2 |
|---|---|---|---|---|---|---|---|---|---|
| Quinolinate synthase A | NADA_ECOLI | NADA_BACSU | 329.0 | 25.8 | 46.2 | 20.9 | 8 | 89.6% | 94.6% |
| Nicotinate-nucleotide pyrophosphorylase [carboxylating] | NADB_ecoli | NADB_BACSU | 775.5 | 38.5 | 55.7 | 6.1 | 8 | 94.4% | 91.0% |
| L-aspartate oxidase | NADC_ECOLI | NADC_BACSU | 489.5 | 35.1 | 51.1 | 12.8 | 4 | 88.2% | 88.9% |
Как уже отмечалось, у Bacillus subtilis (strain 168) рекомендуемое название (Probable nicotinate-nucleotide pyrophosphorylase) [carboxylating]) отличается от такового у E. coli.
3. Результат применения программ выравнивания к неродственным белкам
Для иллюстрирования относительности характеристик выравниваний как показателя гомологичности было проведено парное глобальное и локальное выравнивание для последовательностей с ID AGAA_ECOLI (Putative N-acetylgalactosamine-6-phosphate deacetylase) и COTJA_BACSU (Protein CotJA), результаты приведены в Таблице 3. Первый белок является ферментом, в то время как второй участвует в образовании покровов при формировании споры. Такое сильное функциональное различие свидетельствует о стремящейся к нулю вероятности гомологии.
| Program | ID 1 | ID 2 | Score | Identity, % | Similarity, % | Gaps | Indels | Coverage 1 | Coverage 2 | |
|---|---|---|---|---|---|---|---|---|---|---|
| needle | AGAA_ECOLI | COTJA_BACSU | 10.5 | 2.1 | 3.0 | 94.0 | 1 | |||
| water | 22.0 | 26.4 | 63.6 | 0.0 | 0 | 6.6 | 13.4 | |||
Как видно из Таблицы 3, небольшое количество гэпов в локальном и глобальном и инделей в локальном выравниваниях не может являться достаточным для утверждения, что последовательности являются гомологичными. Для полноценного анализа необходимо учитывать функции белков, структурное соответствие сходных участков и покрытие. Расхождение функций и низкое покрытие указывают, что гомологичность последовательностей маловероятна.
4. Множественное выравнивание белков и импорт в Jalview
В этой части работы было проведено множественное выравнивание для семи (из 245 находок) последовательностей с мнемоникой NADA (Quinolinate synthase A):
- NADA_ECOLI, Escherichia coli K12
- NADA_ARATH, Arabidopsis thaliana (Mouse-ear cress)
- NADA_YERPA, Yersinia pestis bv. Antiqua (strain Antiqua)
- NADA_ORYSJ, Oryza sativa subsp. japonica (Rice)
- NADA_BACSU, Bacillus subtilis (strain 168)
- NADA_NEIMB, Neisseria meningitidis serogroup B (strain MC58)
- NADA_VIBCH, Vibrio cholerae serotype O1 (strain ATCC 39315 / El Tor Inaba N16961)

На Изображении 1 видно, что у 2 и 4 последовательностей, т.е. выделенных из хлоропластов Резуховидки Таля и Риса посевного, имеются длинные почти равные последовательности крайне низкой консервативности, заканчивающийся в пределах 200-250 аминокислотных остатков, далее последовательности продолжат оставаться между собой ближе, чем с остальными, что заметно в участках с длинными инделями. Например, в 590-635 столбцах выравнивания.
В целом последовательности выровнялись хуже предполагаемого, но достаточно хорошо для выявления гомологичности, что смогут лучше проиллюстрировать парные локальные выравнивания. Наиболее консервативные участки короткие (307-370 столбцы), но необходимо учитывать время появления хлоропластов. Прошёл миллиард лет, и расхождение белков из хлоропластов с остальными лишь в несколько остатков может говорить о высокой консервативности (331-339 столбцы).
5. Выравнивание белка с его гомологом
В этом пункте было проведено выравнивание исследуемого в 3 и 8 практикумах белка с его гомологом. Выравнивание можно найти в файле jalview_1.fasta, вывод программы Jalview представлен на Изображении 2 и в проекте jalview_1.jvp. Окрашивание проведено на основании матрицы BLOSUM62 по идентичности 50%.
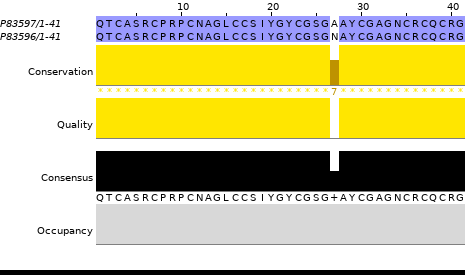
Как видно из выравнивания, последовательности определённо гомологичны, при этом крайне консервативны, о чём свидетельствует факт, что расхождение имеется только в замене одной аминокислоты. Ни инсерций, ни делеций не наблюдается.