Реконструкция филогении по нуклеотидным последовательностям
Построение дерева по нуклеотидным последовательностям
В начале из базы полных геномов NCBI были получены последовательности РНК малой субъединицы рибосомы (16S rRNA) для бактерий из предыдущего практикума. Последовательности были взяты из файлов с раширением .frn и записаны в данный файл.
С помощью Muscle в программе JalView последовательности были выравнены, после чего на основе этого выравнивания в программе Mega5.1 по алгоритму Neighbor-Joining было построено дерево, которое представлено на рисунке 1а. Под буквой б) изображено дерево, построенное тем же методом, но по белку (фактору высвобождения пептидной цепи 1) этих бактерий, которое полностью совпадает с правильным деревом.
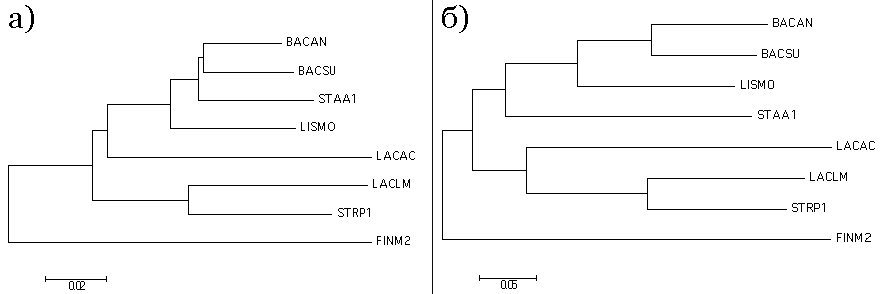
Рисунок 1. Графическое изображение деревьев.
а) дерево, построенное на основе выравнивания нуклеотидных последовательностей; б) дерево, построенное по выравниванию белков из предыдущего практикума.
Рисунок получен с помощью программы Mega5.1. Деревья построены по алгоритму Neighbour-Joining в программе Mega5.1.
Из рисунка 1 видно, что несовпадают следующие нетривиальные ветви:
- {BACAN,BACSU,STAA1} против {LISMO,LACAC,LACLM,STRP1,FINM2};
- {BACAN,BACSU,STAA1,LISMO,LACAC} против {LACLM,STRP1,FINM2}.
Получается, что LACAC из порядка Lactobacillales теперь с бактериями из порядка Bacillales, что довольно странно.
Таким образом, можно сделать вывод, что реконструкция по нуклеотидным последовательностям менее качественная по сравнению с реконструкцией по белкам. Это вероятней всего связано с тем, что сами выравнивания нуклеотидных последовательностей менее достоверны, чем аминокислотных.
Построение и анализ дерева, содержащего паралоги
Далее с помощью программы blastp (порог на E-value 0,001) был проведён поиск гомологов белка CLPX_BACSU в данных бактериях. Использованная команда представлена ниже:
blastp -query clpx_bacsu.fasta -db my_bacs.fasta -evalue 0.001 -out homo.txt
- my_bacs.fasta — файл с протеомами отобранных бактерий
- homo.txt — файл, полученный на выходе.
В результате было найдено 36 гомологов (их последовательности содержатся в данном файле). Потом эти белки были выравнены по алгоритму Muscle в программе JalView, а по выравниванию было реконструировано дерево (алгоритм Neighbor-Joining), изображённое на рисунке 2.
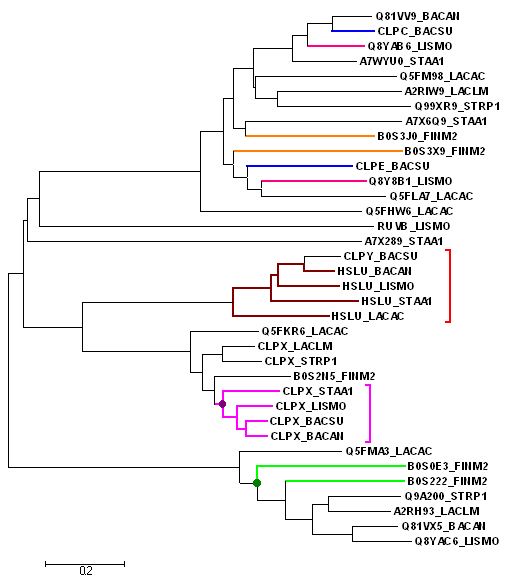
Рисунок 2. Дерево, построенное по последовательностям гомологов белка CLPX_BACSU.
Рисунок получен с помощью программы Mega5.1. Дерево построено по алгоритму Neighbour-Joining в программе Mega5.1.
На рисунке 2 зелёным, розовым, синим, оранжевым цветом выделены примеры паралогов, фиолетовым и бордовым цветом — примеры ортологов (они также выделены скобкой соответствующего цвета). Зелёным кружочком обозначено разделение путей эволюции белков в результате дупликации гена, фиолетовым — в результате видообразования.