Практикум 8. BLAST
Часть 1
Для определения функций последовательности, полученной в практикуме 6, был использован BLASTn с настройками по умолчанию.
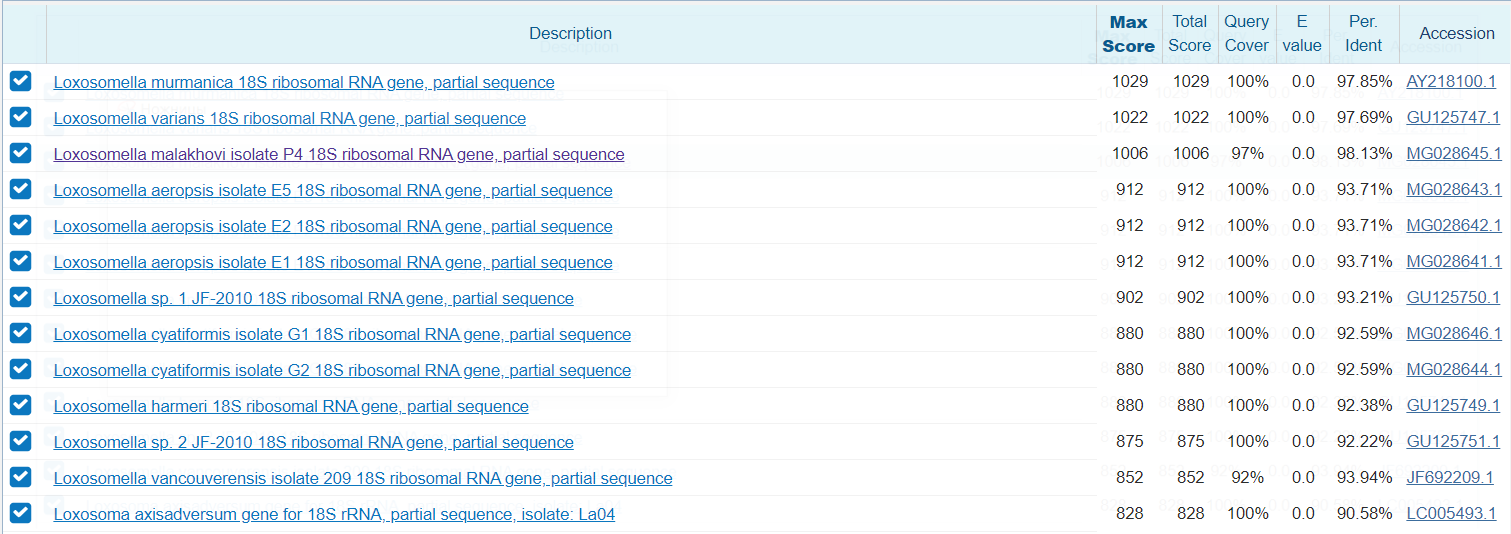

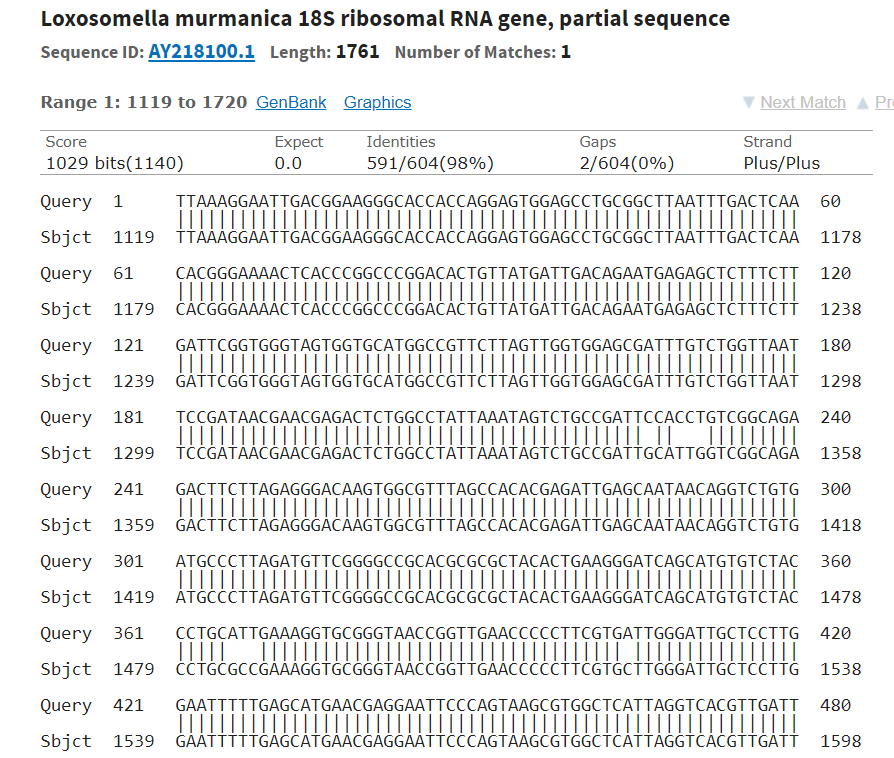
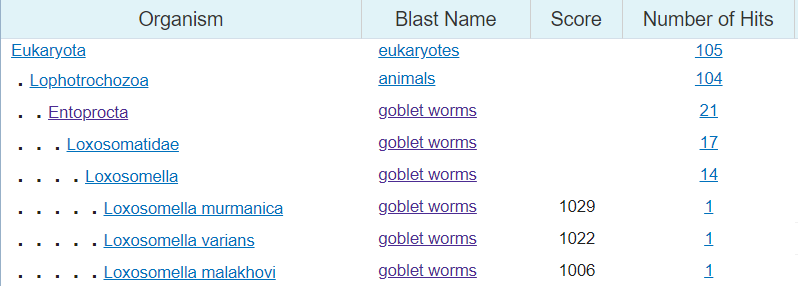
Часть 2. Сравнение алгоритмов
Для последовательности были проведены три сеанса бластования с разными параметрами. Megablast и blastn были проведены с настройками по умолчанию. Чувствительный бласт - Результаты приведены на сводной картинке

При изменении параметров бласта результат выдачи сильно не меняется. Резко изменяются значения E-value при запуске blastn с пользовательскими настройками. Дело в том, что 18S рибосомальная РНК является очень консервативным геном и похожа у разных организмов.
| Megablast | Blastn default | Blastn custom |
|---|---|---|
| Max(250) | Max(250) | Max(250) |
CDS вируса
Для работы был выбран CDS вируса из прошлого практикума.
>lcl|NC_042114.1_cds_YP_009623587.1_5 [locus_tag=FDJ39_gp05] [db_xref=GeneID:40100393] [protein=DNA polymerase] [protein_id=YP_009623587.1] [location=2240..3946] [gbkey=CDS] GTGACTTGCTATTATGCAGGAGACTTTGAAACGACTACAAACGAGGAAGAAACAGAGGTATGGCTATCTT GCTTTGCGAAAGTTATTGACTATGACAAGCTAGACACATTCAAGGTAAACACTAGCTTAGAGGATTTTTT AAAATCGCTCTATCTTGACCTAGACAAGACTTATACCGAGACGGGAGAAGATGAGTTTATCATATTCTTT CACAATCTCAAGTTTGACGGCTCTTTCTTGTTATCTTTCTTTCTGAATAATGATATAGAATGTACTTACT TTATAAATGATATGGGGGTTTGGTATTCTATTACGCTTGAGTTTCCAGACTTTACGCTGACTTTTAGAGA TAGTCTGAAAATTCTGAATTTTTCAATCGCTACGATGGCAGGACTTTTCAAAATGCCTATAGCAAAAGGA ACAACACCCTTGCTAAAACATAAGCCAGATGAAATTAAGCCAGAATGGATTGATTATATTCATGTAGACG TTGCAATTCTTGCTCGTGGTATCTTTGCTATGTATTACGAGGAAAATTTTTCTAAGTACACATCAGCAAG TGAAGCGCTGACAGAGTTTAAACGGATTTTCAGGAAGTCTAAACGAAAATTCAGAGACTTTTTCCCAATC TTGGACGAAAAGGTGGATGATTTTTGTCGTAAAGCCTATCGTGGGGGCTGGACGTTTGCCAATCCTAAAA CGCAGGGGCGCACGCTTAAACAGTTGATAGACATCTATGACATTAACAGTATGTACCCAGCGACCATGTT ACAGAACGCTTTACCTATTGGAATACCGAAGCGATACAAAGGCAAGCCCAAAGAGATAAAGGAAGACCAC TATTATATCTACCACATTAAAGCCGATTTTGACTTAAAACGTGGCTACCTCCCAACTATCCAGATTAAGA AAAAACTGGACGCTTTAAGAATCGGAGTCCGCACTAGCGACTATGTGACTACATCGAAGAACGAGGTTAT AGATTTATATTTGACTAATTTTGACCTTGATTTATTCTTAAAGCACTATGACGCAACTATCATGTATGTT GAAACACTTGAATTTCAGACAGAATCAGGCTTGTTTGATGATTATATCACAACTTACAGATACAAGAAAG AAAACGCACAAAGCCCAGCAGAAAAACAAAAGGCTAAGATTATGCTAAATAGCTTATACGGGAAGTTTGG CGCTAAAATCATATCTGTTAAGAAACTAGCCTATCTGGACGATAAAGGTATATTACGCTTTAAAAATGAC GATGAAGAAGAAGTACAGCCCGTTTACGCACCTGTTGCCCTTTTCGTAACATCTATTGCCCGTCACTTTA TTATATCGAACGCACAAGAAAACTACGATAATTTCTTATATGCAGATACAGACAGCTTGCACTTGTTCCA TTCTGACAGCCTTGTCCTTGACATTGACCCGTCAGAGTTCGGCAAGTGGGCGCATGAGGGGAGAGCCGTT AAGGCAAAATACTTACGCTCAAAACTCTACATCGAAGAGTTGATACAAGAAGACGGGACAACACACCTAG ACGTCAAGGGCGCAGGTATGACCCCAGAGATTAAAGAAAAAATCACTTTTGAAAACTTTGTTATCGGGGC AACTTTTGAGGGTAAAAGGGCAAGTAAGCAGATTAAAGGAGGTACGCTAATTTATGAAACAACCTTTAAA ATCAGGGAAACAGACTATCTCGTATGA
Эта последовательность была отбластована три раза. Данные о выдачах приведены на картинке

Для повышения чувствительности была уменьшена длина слова. Другие настройки бласта не работали за приемлимое время.
megablast показал лишь гены самых близких родственников вируса - другие вирусы.
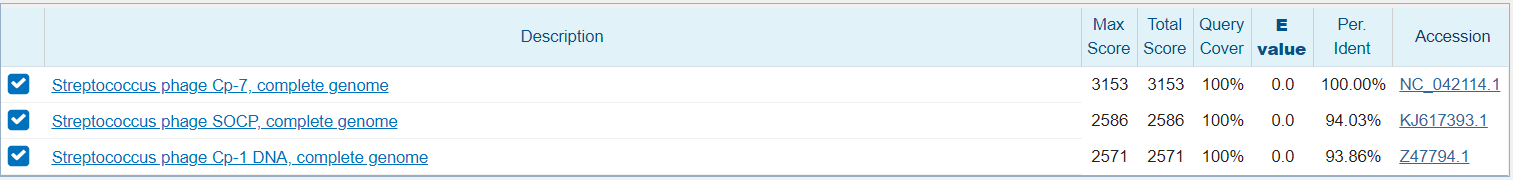
blastn выдал уже значительно больше вариантов. Примечательно, что все они имеют хорошо узнающийся и консервативный участок на конце. Также среди них был еще один вирус(но уже не бактериальный, а мимивирус). Три новые находки имели E-value меньще 0,5, что мы будем считать значимым.
В третьем случае добавилось еще несколько последовательностей, но по сравнению с разницей megablast - blastn скачок не такой значительный. Но появилась еще одна с E-value 0.38.
| Megablast | Blastn default | Blastn custom |
|---|---|---|
| 3 | 57 | 65 |
Гомологи трех белков в неаннотированном геноме
Для выполнения этого задания были выбраны TBP - TATA связывающий белок, бета4A-цепь тубулина и гистон H3.2. Их последовательностти были скачены из Uniprot. Все белки человеческие.
seqret sw:tbp_human tbp.fastaseqret sw:tbb4a_human tbb4a.fasta
seqret sw:h32_human h32.fasta
makeblastdb -in X5.fasta -dbtype nucl
tblastn -query tbp.fasta -db X5.fasta -db_gencode 6 > tbp.txt
tblastn -query tbb4a.fasta -db X5.fasta -db_gencode 6 > tbb4a.txt
tblastn -query h32.fasta -db X5.fasta -db_gencode 6 > h32.txt
| tbp_human | tbb4a_human | h32_human | |
|---|---|---|---|
| Количество находок |
2 | 6 | 14 |
| Лучшая находка |
unplaced-986 | unplaced-665 | scaffold-104 |
| E-value лучшей |
2e-95 | 0.0 | 2e-78 |
| Score лучшей |
314 | 781 | 256 |
| Вывод | Скорее всего гомологична. Наложилось более половины белка с процентом схожести 92%. | Определенно гомологична. При полном перекрытии процент схожести 92% | Определенно гомологична. При полном перекрытии процент схожести 96% |
TBP - транскрипционный фактор, играет очень важную роль в регуляции транскрипции у эукариот. Непосредственно помогает РНК-полимеразе сесть. Тубулин - белок микротрубочек. Характерен для всех эукариот. Гистон 3.2 - один из коровых гистонов, формирует нуклеосому. Все они обеспечивают клеточные механизмы, характерные только для эукариот(за редким исключением).
Поиск белков в контиге
Для поиска гена был взят произвольный контиг из сборки ASM274050v1 Aspergillus niger. Поиск осуществлялся в БД RefSeq и при стандартных настройках бласта.
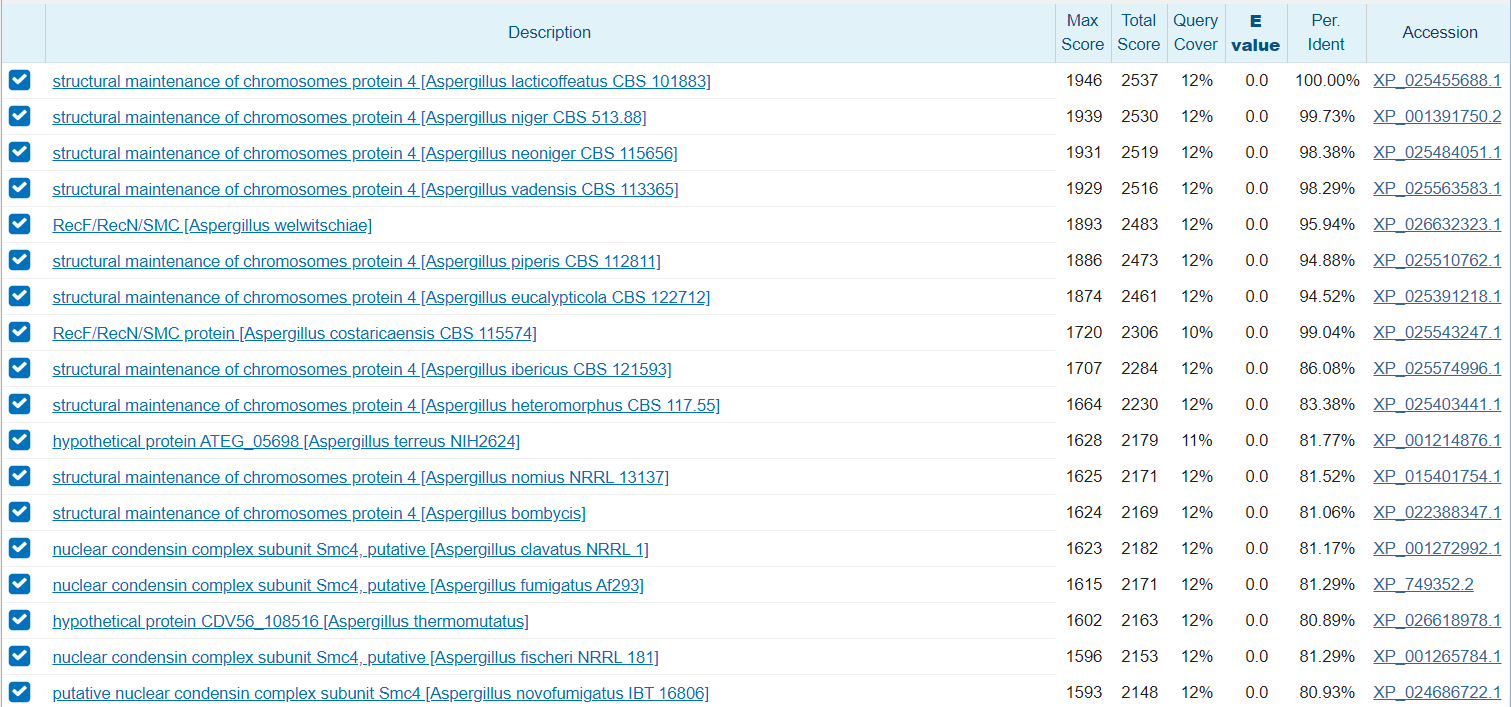
В данный контиг по видимому попал белок структурной поддержки хромосом номер четыре. Этот белок участвует в конденсации хромосом.
