| Белок для исследования | Белок-прототип |
|
SWISS_Prot ID — PIP28_ARATH
SWISS-Prot AC — Q9ZVX8 |
Последовательность здесь |
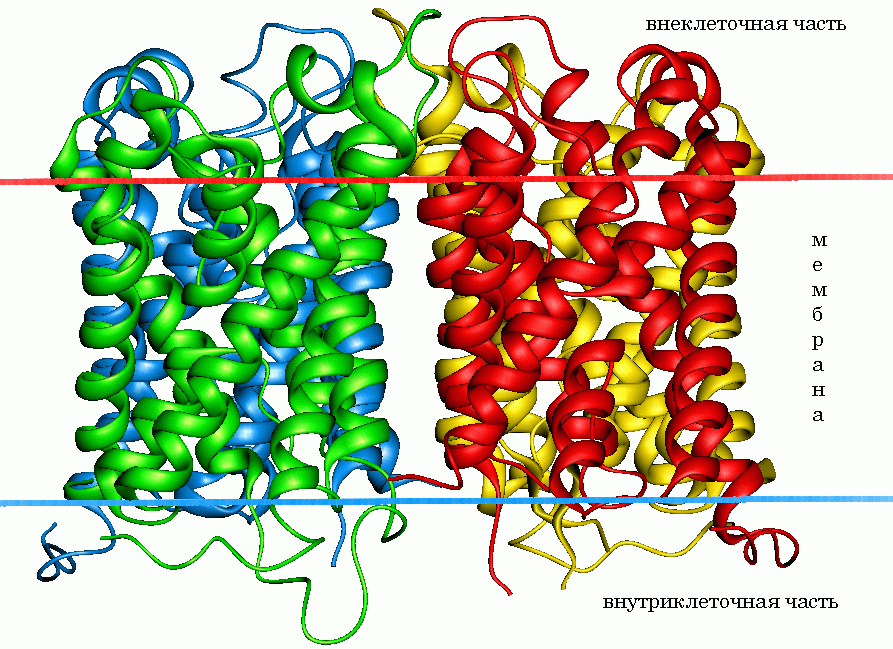
| PDB ID | 2b5f |
Данный мне PDB ID — тетрамер.
Все 4 субъединицы совпадают на 100%.
Но с белком PIP28_ARATH процент идентичности — 78.9%. Выравнивание сохранено в GeneDoc.
Выравнивание исследуемого белка с заданным прототипом:
Последовательность для исследуемого белка получена с помощью команды для UNIX.
seqret sw:Q9ZVX8
Далее выравнивание было получено с помощью программы needle:
needle pip28_arath.fasta prototip.fasta pip28-prot.aln
Это выравнивание сохраненно в GeneDoc
Score: 1131.0
Идентичность: 77.0%
Предсказание топологии почти вручную:
Для белка PIP28_ARATH (изучаемый белок) был построен профиль гидрофобности аминокислотной последовательности.
Данные для построения профиля получены с помощью программы pepwindow пакета EMBOSS:
pepwindow -graph data -length 19 pip28_arath.fasta
Размер скользящего окна равен 19 аминокислотным остаткам.
Трансмембранные сегменты в последовательности размечены с помощью программы GeneDoc по данным, полученным из pepwindow.
За гидрофобные сегменты считались те, значение гидрофобности которых больше 1,8.
Вид предсказания можно посмотреть здесь.
Комментарии:
На этом этапе для определения внеклтеточных и внутриклеточных/цитоплазматических участков были использованы данные ExPASy.
При этом были обнаружены 2 трансмембранных участка, которые не были идентифицированы по признаку "значение гидрофобности больше 1,8".
Учитывая профиль гидрофобности, некоторые аминокислотные остатки (с наибольшим значением гидрофобности) были также причислены к трансмембранным.
На основании всех полученных данных была сделана окончательная версия границ трансмембранных участков (строка hands).
Предсказание топологии с помощью наиболее популярной программы:
Tопология заданного белка предсказана с помощью сервера TMHMM (опции по умолчанию).
Полученное предсказание можно посмотреть здесь.
К таблице "Топология белка PIP28_ARATH" добавлена соотвествующая колонка. Также изменения внесены и в файл GeneDoc.
Комментарии:
Программа TMHMM выдает данные как о границах трансмембранных участков, так и о местоположении вне- и внутриклеточных петель.
Выделение трансмембранных сегментов в 3D-структуре прототипа:
По идентификатору PDB (2b5f) найдено описание трансмембранных сегментов в БД OPM (Orientations of Proteins in Membranes database).

Данные можно посмотреть здесь.
К таблице "Топология белка PIP28_ARATH" добавлена соотвествующая колонка. Также изменения внесены и в файл GeneDoc.
Комментарии:
Этот метод основывается не на последовательности самого белка (PIP28_ARATH), а на его гомологе, имеющем PDB структуру в БД OPM.
Поэтому результаты заведомо неточные: ОРМ нашел 2 "лишних" трансмембранных участка: 102-111 и 223-232.
Но в целом границы неплохо совпадают.
Сравнение предсказанных топологий и положения белка в мембране:
Таблица сравнения находится здесь.
Комментарии:
| На картинке изображена субъединица А структуры 2b5f.pdb. Она получена путем выполнения следующих команд: background white wireframe off backbone 70 color structure restrict *A or hetero select hetero cpk 100 color cpkТак же показаны границы мембраны: внешняя граница (красные шарики) и внутренняя граница (синие шарики). Внутренняя граница обращена в цитоплазму. Cпиральные участки покрашены к красный цвет.
Итак, вне клетки расположены следующие фрагменты:
В цитоплазме находятся:
|
В 3 случаях неспиральная часть заходит в мембрану (95-101, 196-198, 219-222 атомы).
Из этих фрагментов нас интересуют не все, а только концы спиралей:
94 атом в цитоплазме,
все остальные находятся на внешней стороне мембраны:
59-63,
74,
138,
142-147,
161-163,
215-217,
233,
236-241.
|
|
Кроме того, толщина мембраны около 20 а.о., а эти фрагменты в 2 раза меньше.
|