Сравнение выравниваний
Поиск гомологов белка B3PEE6 в SwissProt
При запуске BLAST были использованы следующие параметры:
1. Query subrange. С помощью этого параметра можно применить BLAST не ко всей последовательности,
а только к ее участку (координаты: от 1 до длины последовательности). Я выставил по умолчанию: от начала до
конца.
2. Database. Выбор базы данных. Я выбрал UniProtKB/Swiss-Prot(swissprot).
3. Organism. Ограничить поиск только белками некоторых видов (или, наоборот, исключить некоторые виды -
exclude). Кроме того, можно исключить белки с теми или иными идентификаторами RefSeq. Я запускал BLAST
по всем организмам.
4. Algorithm. Выбор алгоритма; я использовал blastp (по умолчанию). Другие варианты: PSI-BLAST (Position-Specific Iterative
BLAST), PHI-BLAST (Pattern Hit Initiated BLAST), DELTA-BLAST (Domain Enhanced Lookup Time Accelerated BLAST).
Так или иначе, все эти алгоритмы улучшают поиск гомологов, так как учитывают важность отдельных консервативных
блоков.
5. Max target sequences. Максимальное число находок, выставил 20000.
6. Short queries. Надо ли выставлять параметры (см. ниже) для коротких входных последовательностей
автоматически? В данном случае не имеет значения, так как входная последовательность длинная.
7. Expect threshold. Верхний порог E-value. Чем больше, тем менее "хорошие" находки будут включены
в выдачу.
8. Word size. Длина слов, по которым идет индексирование в алгоритме BLAST. Я выставил 3 - чем меньше
длина слова, тем лучше работает алгоритм (но и дольше).
9. Max matches in a query range. Ограничить число последовательностей, выровненных по одному участку.
По умолчанию 0. Этот параметр следует менять, если множество совпадений с одним участком мешают blast
найти последовательности, похожие по другим участкам.
10. Matrix. Матрица замен. По умолчанию BLOSUM62. Доступны различные матрицы PAM и BLOSUM.
11. Compositional adjustments. Корректировка матрицы замен для борьбы с участками малой сложности,
в которых определенные аминокислоты передпредставлены. Существуют разные методы, по умолчанию -
Conditional compositional score matrix adjustment.
12. Filter и Mask. Можно не учитывать участки малой сложности (filter low-complexity regions). Либо же не учитывать их только
при создании индексов (lookup table), по которым BLAST будет производить поиске (mask for lookup table only). Кроме того, можно
маскировать строчные буквы в последовательности. По умолчанию все эти параметры отключены.
Выдача BLAST доступна в Таблице.
Для построения множественного выравнивания были выбраны 10 последовательностей из различных организмов и с
различными Similarity и E-value. Вы можете скачать выравнивание в формате FASTA.
Последовательности с относительно высоким E-value действительно оказались не очень близки белку B3PEE6, поэтому были удалены,
как и некоторые с высоким E-value.
Вы можете скачать полученное выравнивание оставшихся четырех белков.
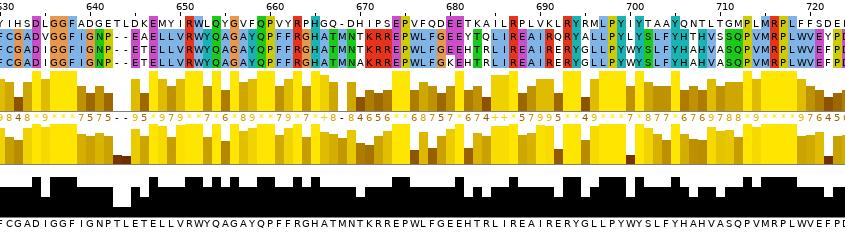
Однако, что любопытно, из оставшихся белков самым выбивающимся оказался сам B3PEE6 (см. Рис. 1). Это связано
с тем, что мой белок - олигосахарид-4-альфа-D-глюкозилтрансфераза из генома бактерии Cellvibrio japonicus
Ueda107 - специфичный фермент данной бактерии, обладающей способностью разлагать клеточную стенку растений и
имеющей достаточно уникальный метаболизм. Поэтому, хотя данная альфа-глюкозидаза и похожа на множество других
глюкозидаз, между собой прочие белки похожи заметно больше. Тем не менее, гомологичные участки между всеми четырьмя
последовательностями также существуют. Например, с 651 по 660 позицию выравнивания (см. Рис. 1.).
Карта сходства белков E6SLJ6 и E4ZIZ7
Карта сходства получена для белков E6SLJ6 и E4ZIZ7. Первый катализирует превращение 7,8-дигидронеоптерина в 6-гидроксиметил-7,8-дигидроптерин. Функция второго достоверно не известна, но, на основании его схожести с другими дигидронеоптерин альдолазами, можно предположить аналогичную функцию. Длина E6SLJ6 - 445 а.о., длина E4ZIZ7 - 270 а.о.
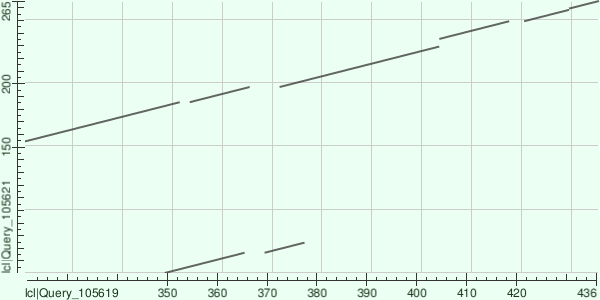
Из карты сходства можно заключить, что в белке E4ZIZ7 произошла дупликация участка. Кроме того, схожи между собой только концы белков (с 150 по 265 а.о. в E4ZIZ7, с 320 по 436 а.о. в E6SLJ6). Можно предположить, что E4ZIZ7 не является функциональным белком, или же выполняет другую функцию. Впрочем, не стоит судить об этом по карте сходства. Она просто показывает наглядно крупные изменения: в нашем случае, дупликацию.
Параметры BLAST
1. Выбор другой матрицы. Я запустил BLAST по своему белку со всеми теми же параметрами, что и в первом задании, но выбрал матрицу замен BLOSUM90. Изменился также параметр Gap costs: по умолчанию штраф за открытие гэпа стал 10, а не 11. Белки в выдаче совпали с моим первым запуском. Однако Score во всех случаях увеличился, а E-value для некоторых находок уменьшилось, а для некоторых увеличилось. В принципе, в этом нет ничего удивительного, ведь в blosum90 по сравнению с blosum62 все числа меньше, а числа на диагонали больше (замены "на себя" дороже, а остальные дешевле). Кроме того, уменьшился штраф за гэпы. Поэтому нам просто повезло, что Score увеличился: большие бонусы за совпадения и меньшие штрафы за гэпы "перевесили" больший штраф за несовпадение. Изменение E-value тоже имеет объяснение: при пересчете Score в Bit Score мы учитываем, что матрица замен другая.