Занятие 10: Эволюционные домены (часть 2)
< Часть перваяПостроение филогенетического дерева
Для построения филогенетического дерева сначала отредактируем имена последовательностей так, чтобы в названии каждой из них была отражена доменная архитектура и сравниваемые таксоны. Так как для сравнения были выбраны двухдоменная и трёхдоменная архитектуры, то разумно закодировать соответствующие доменные архитектуры цифрами 2 и 3. Соответствующие выбранные таксоны были обозначены следующим образом:
- для доменной архитектуры 1 (Cyclin_N, Cyclin_C):
- Ma – Mammalia,
- Am – Amphibia,
- Ac – Actinopterygii;
- для доменной архитектуры 2 (F-box, Cyclin_N, Cyclin_C):
- Eu – Eutheria,
- Ba – Batrachia,
- Me – Metatheria,
- Di – Dinosauria.
Для того, чтобы переименовать последовательности в файле с выравниванием, были использованы файл с информацией о таксономии белков, в которых присутствует домен Cyclin_N, и файл с информацией об архитектуре последовательностей (были получены на предыдущем занятии) и написан и применён скрипт rename_seqs.py.
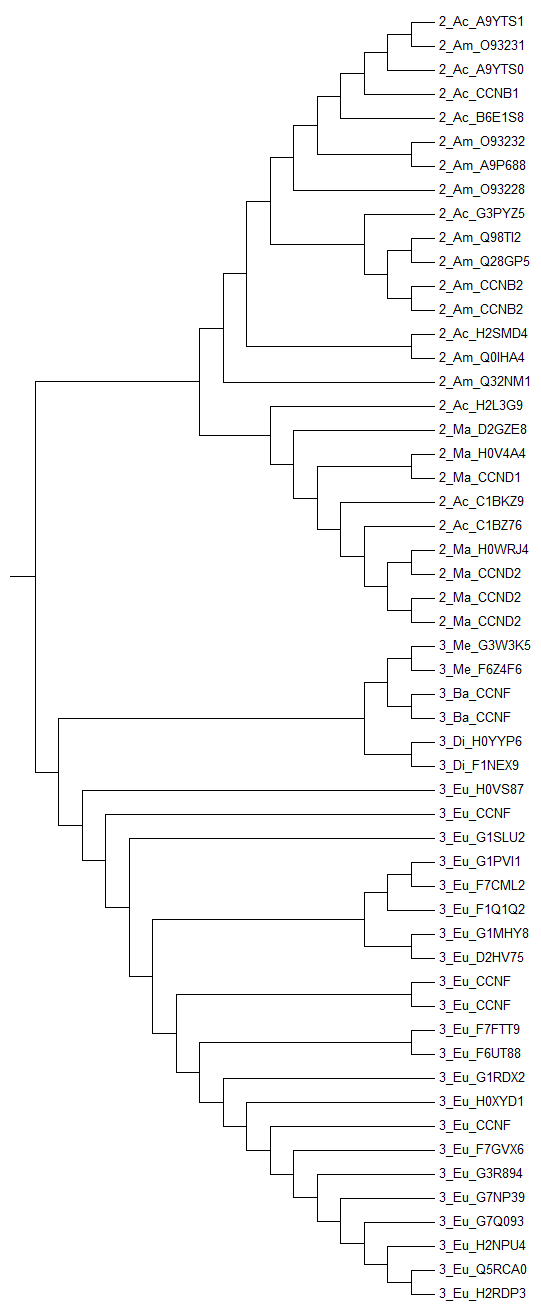
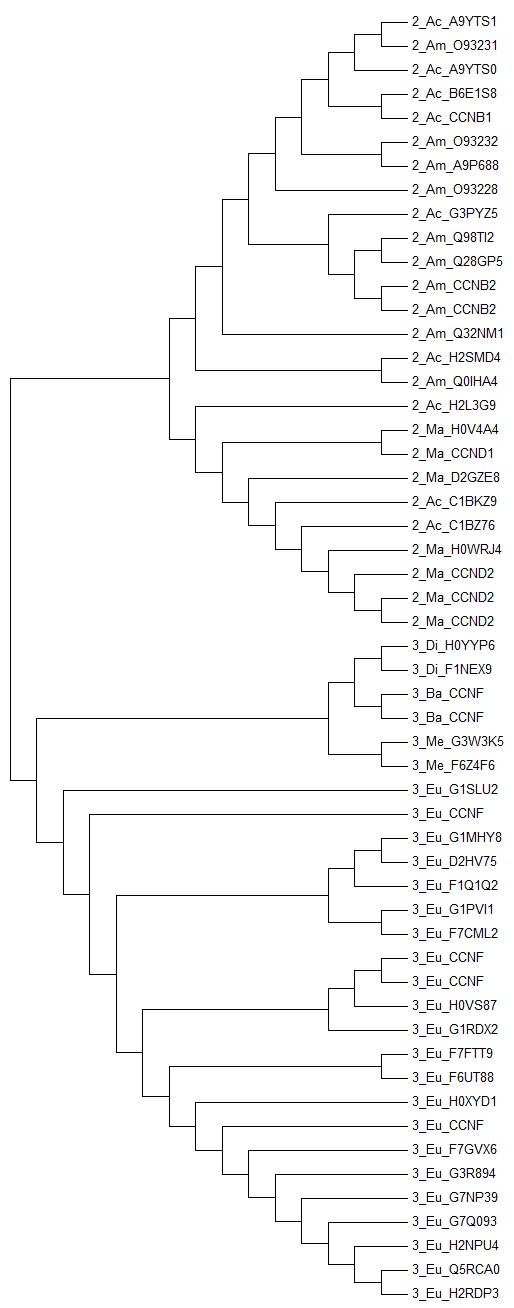
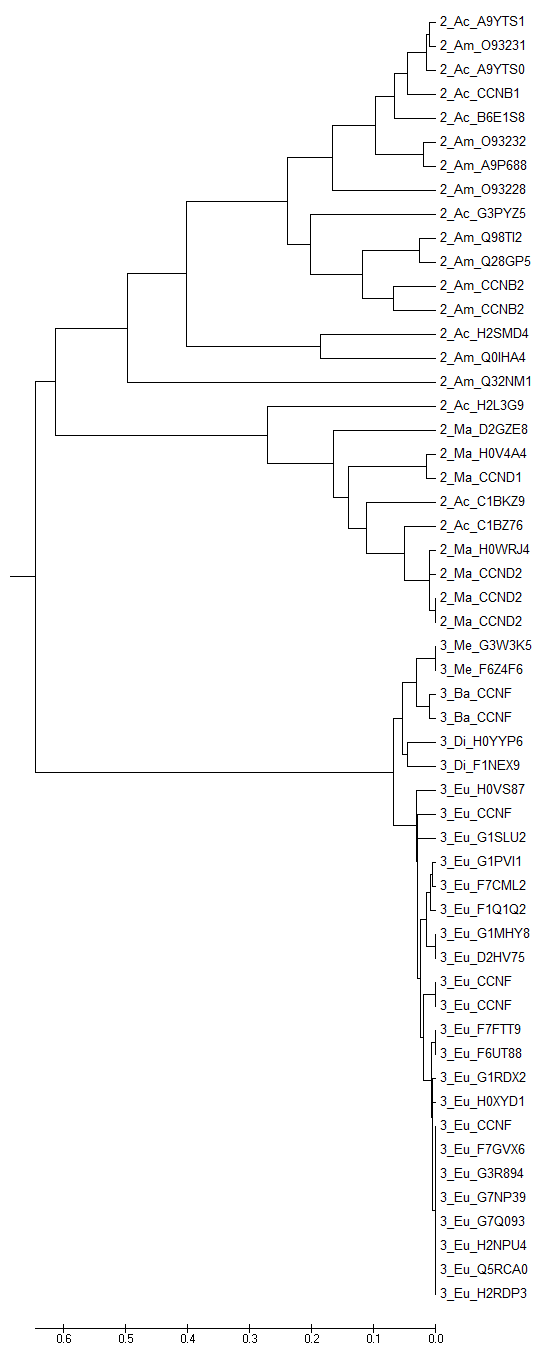
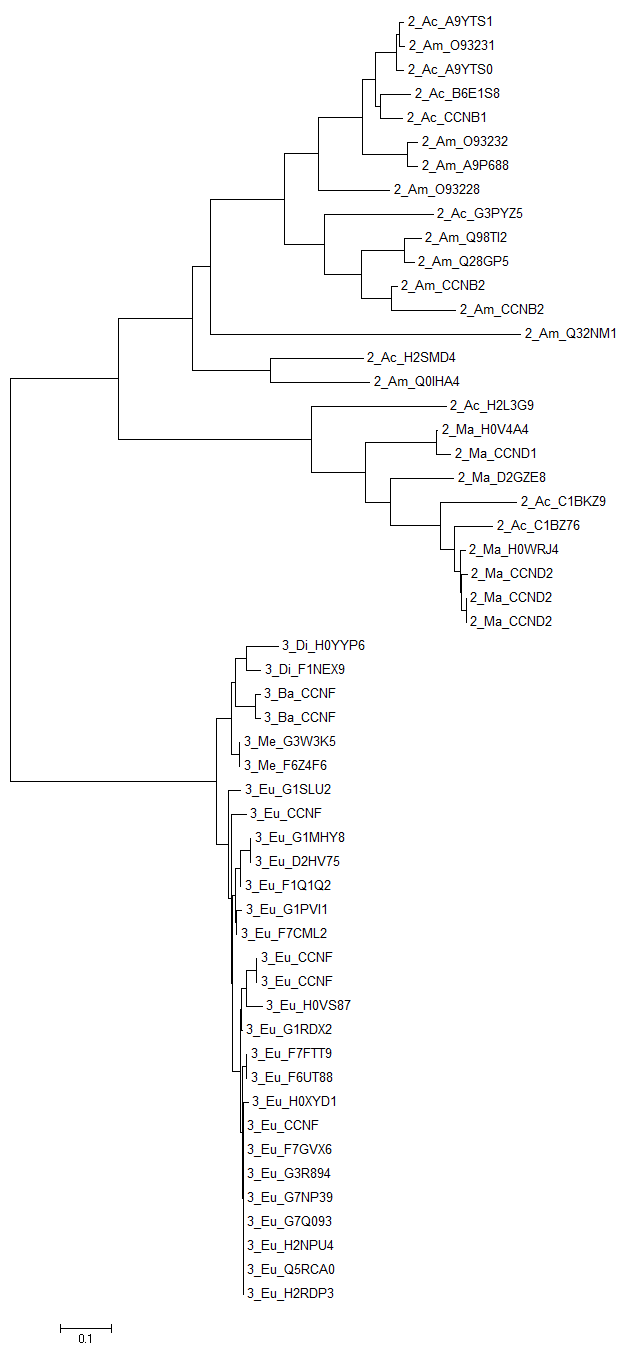
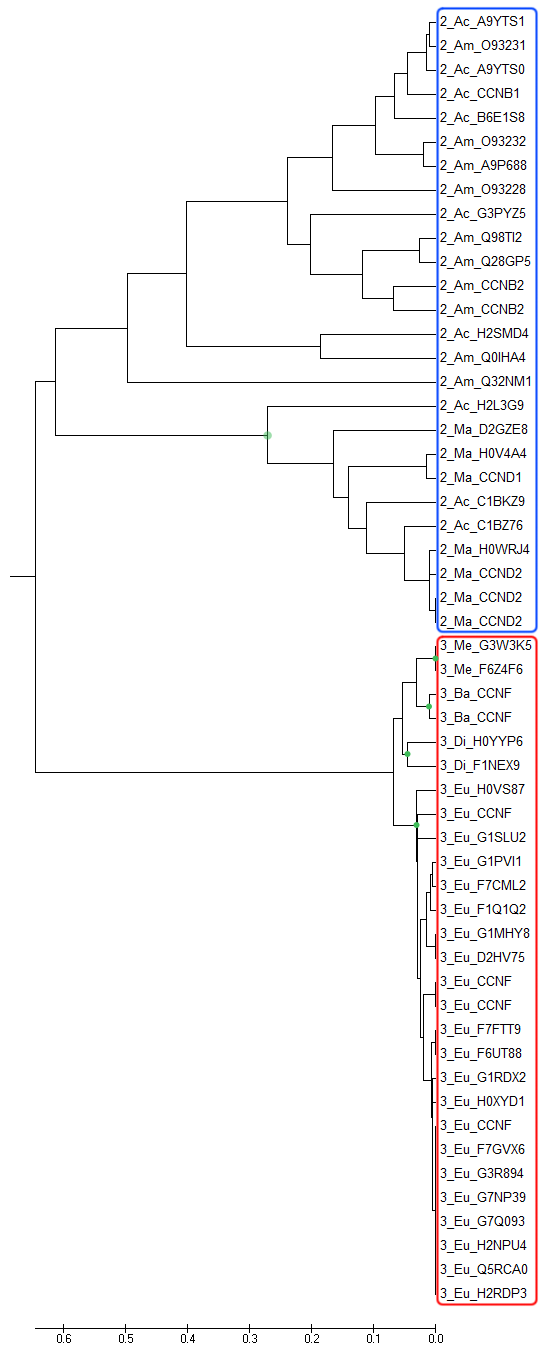
Построения дерева на основе полученного выравнивания было выполнено методами UPGMA (предполагает молекулярные часы, строит укоренённое дерево) и Neighbor-Joining (не использует гипотезу о молекулярных часах и строит неукоренённое дерево), чтобы затем иметь возможность сравнить построенные деревья. Полученные с помощью программы MEGA изображения построенных деревьев (и отдельно топологии этих деревьев) приведены ниже.
Скобочные формулы для деревьев (формат Newick): UPGMA (топология), Neighbor-Joining (топология).
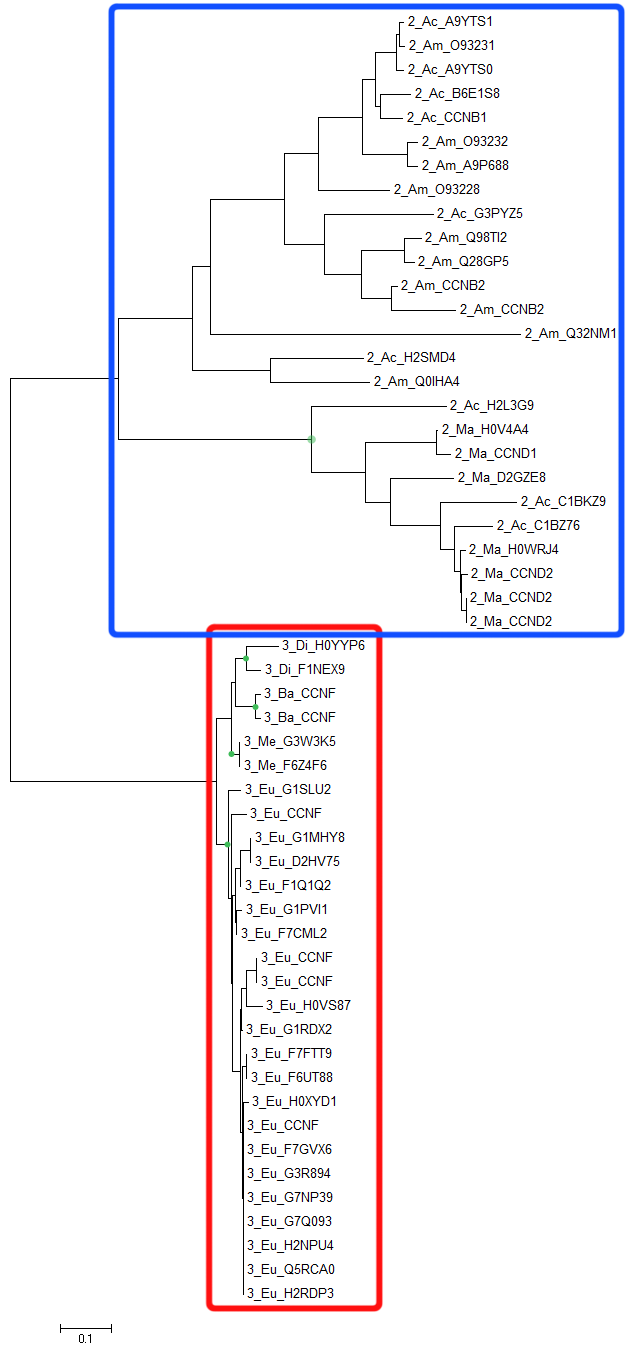
Для наглядности демонстрации результата можно использовать выделение цветом клад дерева по доменной архитектуре. Серия изображений ниже (они аналогичны приведённым выше деревьям, но содержат дополнительную информацию в виде выделения цветом отдельных элементов дерева) демонстрирует, что доменные архитектуры чётко разграничены в эволюции и их разделение произошло «давно» (в эволюционном смысле). Дерево, построенное методом UPGMA, практически полностью (за исключением некоторых последовательностей) совпадает с деревом, реконструированным по методу Neighbor-Joining и укоренённым таким образом, чтобы две клады соответствовали доменным архитектурам (вообще, такое укоренение не всегда возможно, если рассматривать всё множество возможных топологий для рассматриваемых последовательностей). Разделение на выбранные подтаксоны для последовательностей с рассматриваемой трёхдоменной архитектурой чётко прослеживается в построенных деревьях (точки зелёного цвета приведены в качестве ориентира), чего нельзя сказать о последовательностях с двухдоменной архитектурой: выделяется лишь ветвь, ведущая к млекопитающим (Ma; полупрозрачная точка зелёного цвета приведена в качестве ориентира), однако и в этой кладе встречаются последовательности из лучепёрых рыб (Ac).
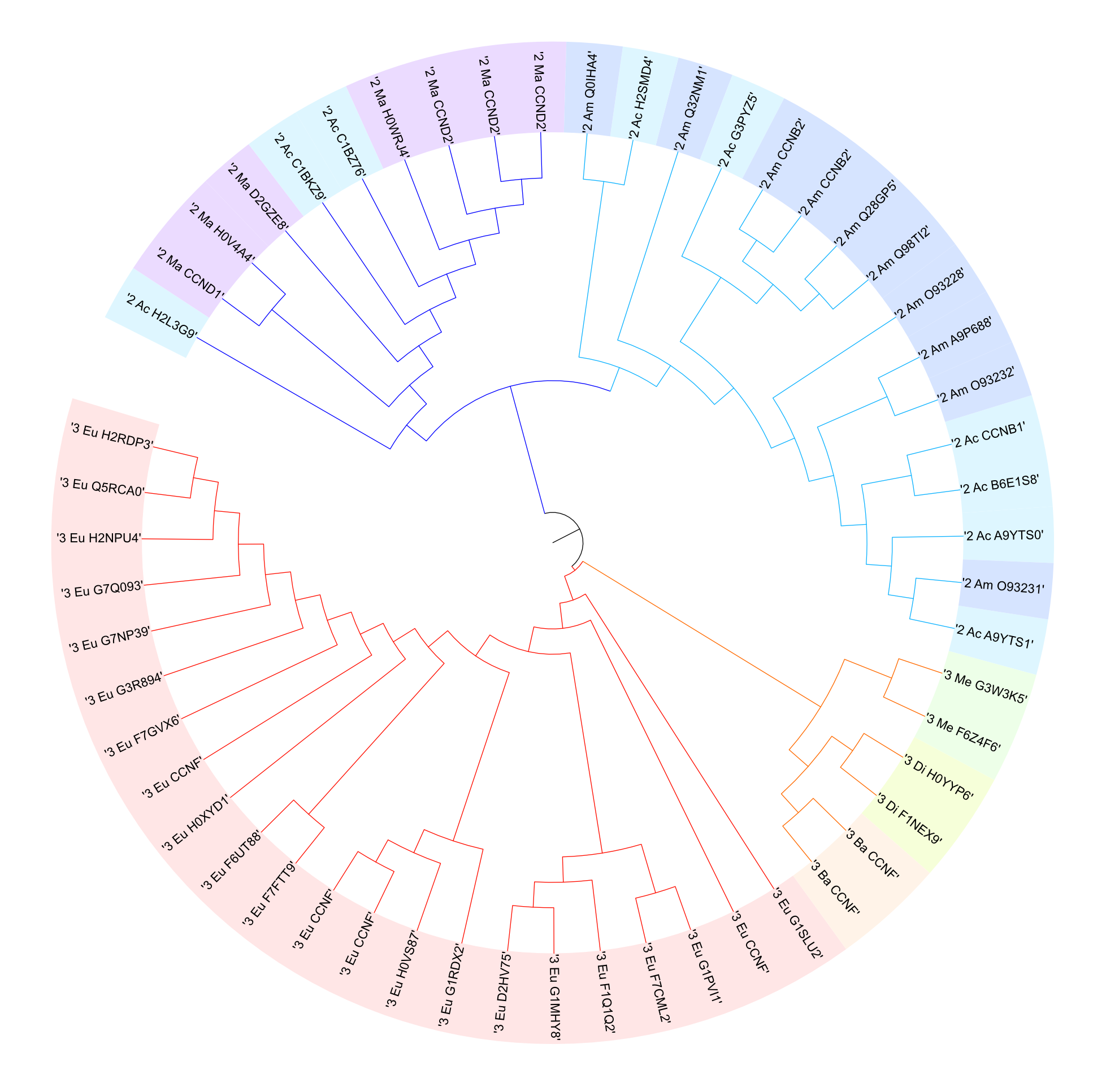
Изображения, полученные с помощью сервиса для визуализации филогенетических деревьев iTOL для рассматриваемого набора последовательностей (было использовано дерево, построенное по методу Neighbor-Joining), представлены ниже:
Для оценки достоверности полученных деревьев был использован бутстрэп-анализ, на основании которого можно получить информацию о поддержке ветвей. Изображение для консенсусного дерева, реконструированного по методу UPGMA с использованием Bootstrap (число реплик – 100), приведено ниже. Как видно, деление дерева на 2 клады по доменной архитектуре присутствовало в 100% деревьев при анализе Bootstrap. Также высокую поддержку имеют ветви, ведущие к подтаксонам для трёхдоменной архитектуры. Клада, к которой относятся последовательности всех млекопитающих из числа рассматриваемых (среди последовательностей с двухдоменной архитектурой), присутствовала в 100% случаев.
Для того, чтобы более точно описать эволюцию доменной архитектуры белков, содержащих домен Cyclin_N, было решено произвести новый выбор таксонов:
- для доменной архитектуры 1 (Cyclin_N, Cyclin_C):
- A – Alveolata,
- V – Viridiplantae,
- M – Metazoa,
- F – Fungi;
- для доменной архитектуры 2 (F-box, Cyclin_N, Cyclin_C):
- M – Metazoa.
Вновь был произведён отбор последовательностей и построение дерева. На основании приведённого ниже изображения дерева, реконструированного по методу UPGMA с использованием 100 реплик Bootstrap, можно сделать предположение, что выделение двух рассматриваемых доменных архитектур произошло ещё до разделения эукариот на царства (в частности, до выделения Metazoa).
Часть третья >
Ссылки
- Файл cyclin_n_alignment.fa.
- Файл cyclin_n_taxonomy.txt.
- Файл cyclin_n_xls.txt.
- Файл cyclin_n_upgma.nwk.
- Файл cyclin_n_upgma_topology.nwk.
- Файл cyclin_n_nj.nwk.
- Файл cyclin_n_nj_topology.nwk.
- Файл cyclin_n_big_taxa_renamed.fa.
- Файл rename_seqs.py.