Занятие 8: Особенности мембранных белков
Описание трансмембранных белков с известной 3D структурой
Для трёх трансмембранных бета-баррелей и трех трансмембранных альфа-спиральных белков с помощью базы данных OPM был определён ряд параметров, приведённых таблице ниже.
| PDB код | Тип | Мембрана | Толщина гидрофобной части мембраны, Å | Медиана числа остатков в одном трансмембранном участке |
| 2GFP | Спираль | Внутренняя мембрана Escherichia coli | 31.6 ± 1.5 | 21 |
| 2UUH | Спираль | Мембрана эндоплазматического ретикулума Homo sapiens | 29.4 ± 1.1 | 18.5 |
| 4B4A | Спираль | Внутренняя мембрана Aquifex aeolicus | 30.4 ± 1.2 | 22 |
| 1QJP | Баррель | Внешняя мембрана Escherichia coli | 25.4 ± 1.5 | 9 |
| 3PIK | Баррель | Внешняя мембрана Escherichia coli | 24.3 ± 1.2 | 10 |
| 2JK4 | Баррель | Внешняя мембрана митохондрий Homo sapiens | 23.4 ± 2.3 | 7 |
Отбор гомологов
Для белка 1M56 (цепь A) с помощью PSI-BLAST был произведён поиск гомологов. Белок 1M56 принадлежит протеобактерии Rhodobacter sphaeroides. При поиске гомологов были исключены белки из филума Proteobacteria. После четырёх итераций PSI-BLAST (порог на E-value 1e-5, максимальное количество хитов 1000) было отобрано 12 хитов из разных таксономических групп. Затем названия последовательностей были отредактированы с сохранением указания на сам белок и соответствующий ему организм.
Анализ структуры выданного белка
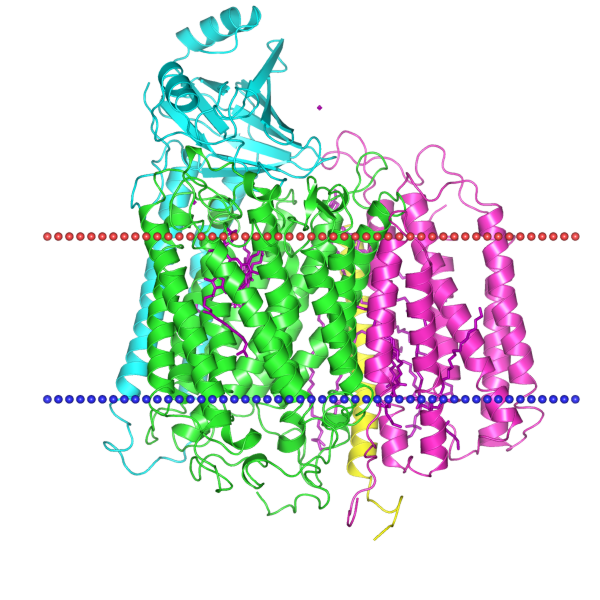
Информация о белке 1M56 (цепь A), полученная с помощью баз данных OPM и TCDB, представлена в таблице ниже.
| PDB ID | Организм | Тип мембраны | TC-код | Наклон спиралей к нормали | Количество трансмембранных спиралей | Название белка |
| 1M56 | Rhodobacter sphaeroides | Внутренняя мембрана | 3.D.4.6.2 | 3 ± 0°
Отдельно для четырёх субъединиц: 4°, 12°, 6°, 19°. |
22 (4 субъединицы) | Бактериальная цитохром c-оксидаза |
Описание каждого из полей TC-кода для белка 1M56:
- 3.* – первичные активные транспортёры.
- 3.D.* – транспортёры, осуществляющие свою работу за счёт окислительно-восстановительных реакций.
- 3.D.4.* – Суперсемейство транспортирующих протоны цитохромовых оксидаз (COX).
- 3.D.4.6.2 – 4 компонента системы цитохром c-оксидазы (субъединицы I – IV, в том числе субъединица I – белок P33517).
Анализ множественного выравнивания трансмембранных белков
С помощью программы MUSCLE было построено множественное выравнивание отобранных гомологов. Затем результат был загружен в программу JalView. К выравниванию были добавлены аннотации TM_REAL и TM_PREDICTED. TM_REAL содержит указания на участки выравнивания отвечающие трансмембранным спиралям в белке со структурой 1M56:A, которая была прикреплена к последовательности белка COX1_RHOSH (исходного) в выравнивании в программе JalView. TM_PREDICTED обозначает предсказанные с помощью TMHMM участки трансмембранных спиралей в гомологе COX1_HUMAN.
После применения цветовой схемы, позволяющей визуально различать гидрофобные и гидрофильные остатки (Hydrophobicity: гидрофобные остатки выделены красным цветом), вместе с зависимостью интенсивности цвета от консервативности позиции (By Conservation, было использовано значение 20% в качестве порога) было получено следующее изображение:
Часть белка, ориентированная в n-сторону мембраны, расположена вверху. Участки, относящиеся к трансмембранным спиралям, консервативны (хотя встречаются небольшие фрагменты участков неконсервативных остатков), при этом наиболее часто в спиралях встречаются остатки лейцина, изолейцина, валина, аланина, фениалаланина, метионина, глицина и пролина (как видно, трансмембранные участки действительно состоят в основном из гидрофобных остатков). Среди участков между спиралями встречаются как консервативные участки, так и неконсервативные (относительно порога в 20% для цветового отображения консервативности).
Стоит отметить, что в трансмембранных спиралях также нередко встречаются остатки серина, аспарагина, тирозина (полярны), и даже гистидина (полярен и положительно заряжен) и глутаминовой кислоты (полярен и заряжен отрицательно). Эти остатки как правило консервативны (в особенности консервативны остатки гистидина), поэтому закономерно предположить, что их наличие связано с функциональной ролью белка.
Выравнивание в формате .jar (проект JalView).
Если сравнивать результаты программы TMHMM и реальную структурную информацию по совпадению на выравнивании, то можно отметить, что все трансмембранные спирали были предсказаны. Присутствуют участки полного совпадения предсказаний и структурной информации, в большинстве же случаев трансмембранные участки смещены относительно друг друга в среднем на 3 позиции. Согласно создателям программы, TMHMM способна корректно предсказать 97-98% трансмембранных спиралей [источник]. В данном случае программа показала неплохой результат, верно предсказав 92% трансмембранных участков.
Клетка размером с дыню
Проведём мысленный эксперимент: увеличим клетку до размера дыни. Если рассматривать клетку Escherichia coli, то линейные размеры её составляют примерно 0,5 μm × 2 μm (микрометров) [источник], а толщину мембраны можно считать равной 30 Å [источник]. Используя для расчёта «длину» клетки и размер дыни, равный 25см [источник], мы можем сделать заключение о том, что линейные размеры клетки при её увеличении до размера дыни следует увеличить в 1,25×105 раз. Тогда мембрана будет иметь толщину 0,0375 сантиметров (375 мкм). Для сравнения, толщина листа бумаги A4 примерно равна 105 мкм [источник]. Таким образом, мембрану клетки, имеющей размеры дыни, можно сравнить со стопкой из 4 листов офисной бумаги.
Правило positive inside
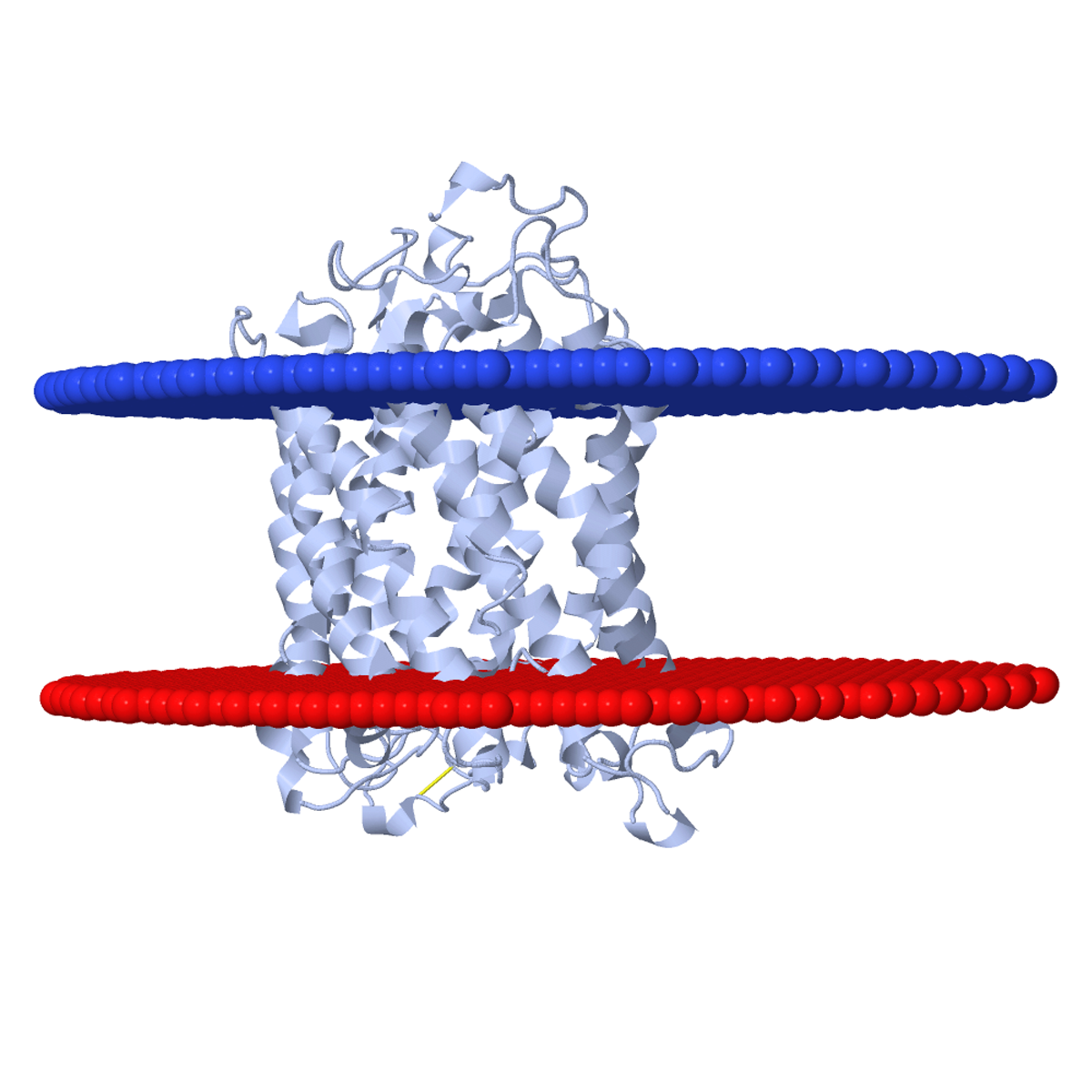
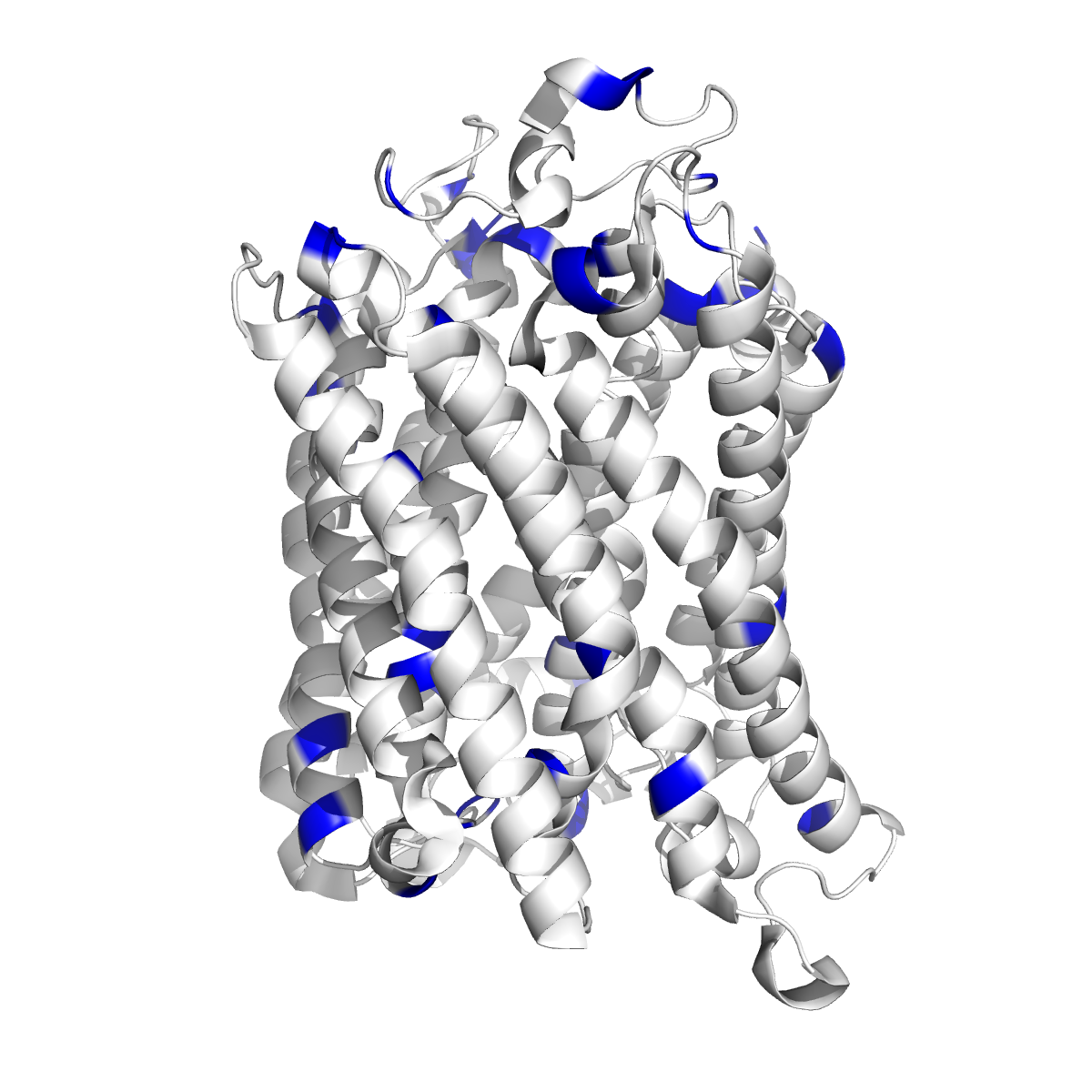
Для белка 1M56 (цепь A) можно выяснить, выполняется ли правило positive inside для трансмембранных белков, состоящее в том, что часть белка, расположенная с n-стороны от мембраны, содержит больше положительно заряженных остатков. Если рассмотреть остатки лизина, аргинина и гистидина в трансмембранных участках данного белка (это условие можно усилить консервативностью в гомологах) и определить с какой стороны мембраны число положительно заряженных остатков больше, то можно наблюдать чёткое следование расположения остатков этому правилу, что в очередной раз демонстрирует его универсальность в применении к трансмембранным белкам.
Для визуальной демонстрации удобнее всего воспользоваться трёхмерной структурной белка. На изображениях ниже приведена структура цепи A белка 1M56. Синий и красный слои на первом изображении обозначают n- и p-стороны мембраны соответственно. На втором изображении в структуре белка, расположенного аналогичным образом (n-стороне соответствует верх изображения), синим цветом выделены остатки лизина, аргинина и гистидина. Видно, что с n-стороны мембраны положительно заряженных остатков больше, чем с p-стороны.
Предсказание бета-баррели по последовательности
В качестве трансмембранной баррели с известной 3D структурой был использован белок VDAC-1 канала 2JK4, который расположен во внешней мембране митохондрий человека. В качестве гомолога был использован найденный белок B3MJX1_DROAN из Drosophila ananassae. В качестве сервисом для предсказания были использованы BOCTOPUS и PRED-TMBB. В целом с предсказанием лучше справился первый сервис, предсказание второго не содержит некоторых трансмембранных участков. Один из участков был пропущен двумя использованными сервисами для предсказания.
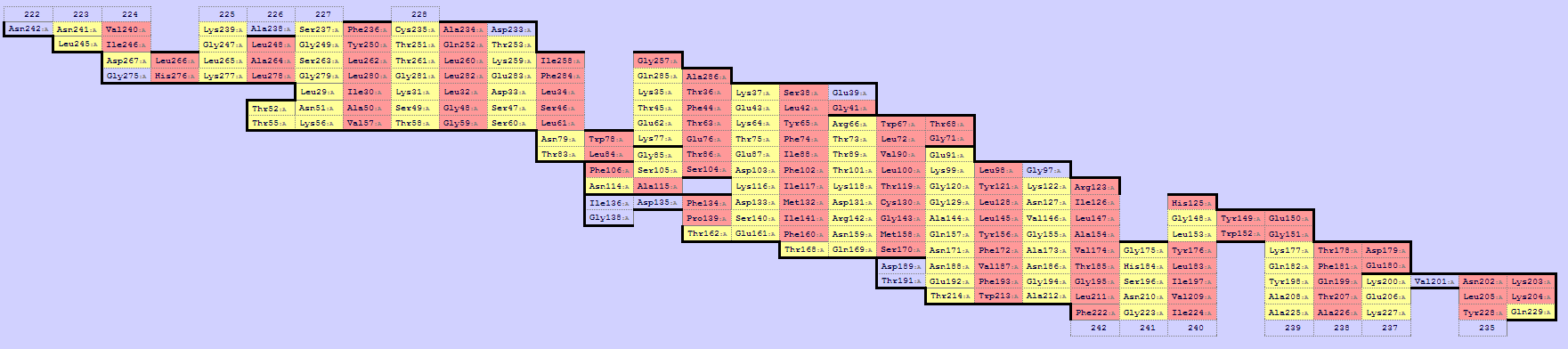
Гидрофобность остатков бета-баррели, обращённых в мембрану
С помощью сервиса SheeP можно определить, какие остатки белка 2JK4 "смотрят" наружу от бета-листа. На составленной карте, приведённой на изображении ниже, красным выделены остатки, боковые цепи которых "смотрят" наружу баррели, жёлтым – внутрь.
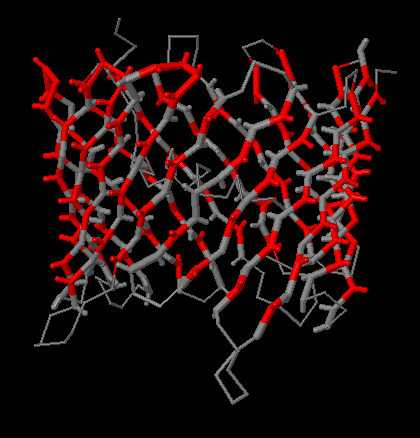
Изображение 3D структуры, на котором смотрящие наружу боковые цепи указаны красным цветом, приведено ниже.
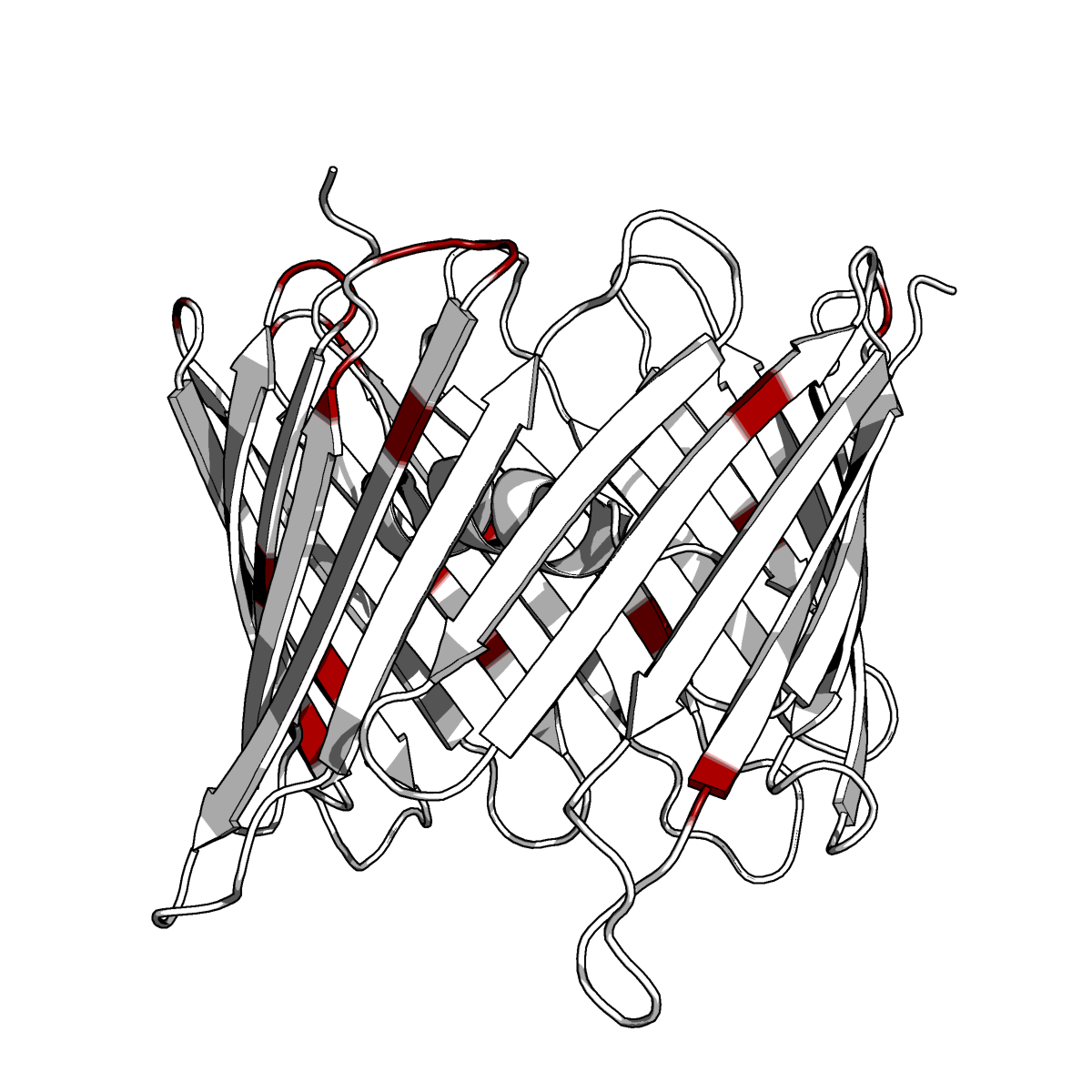
Как видно, большинство (74%) обращённых наружу остатков гидрофобны. Однако встречаются и полярные (гистидин, глутамин, аргинин, лизин, глутаминовая кислота, аспарагин, серин). Как правило, они расположены ближе к одной из сторон мембраны, входя в состав "перегибов" бета-листов.
На изображении ниже такие остатки (полярные остатки, обращённые наружу по результатам анализа структуры с помощью сервиса SheeP) обозначены на структуре рубиновым цветом:
Ссылки
- Файл homologous.txt.
- Файл homologs.txt.
- Файл homologs_with_names.txt.
- Файл homologs_alignment.fasta.
- Файл homologs_jalview_project.jar.
- Файл beta-barrels.fasta.
- Файл beta-barrels_alignment.fasta.
- Файл beta-barrels.jar.