Under construction
2.1.a Как проверять ограничения на домен и доменную архитектуру
Используйте подсказки к практикуму 13 второго семестра. Должно же что-то остаться в вашей голове после 1го курса?:)
Лучше смотреть число белков с помощью запроса к Uniprot, так как скачивать таблицы 1 и 2 и последовательности придётся из него.
Предварительно стоит посмотреть доменные архитектуры в Pfam, так картинки красивые и описания доменов.
Возможно ваши результаты по практикуму 13 облегчат выбор.
2.1.b Составление таблиц 2 и 1
Помнится, вы выполняли подобное задание в практикуме 13.
Задаёте условия на то, какой домен Pfam должен содержать белок; для таблицы 1 - какой еще домен Pfam должен содержать белок.
Если потребуется - условие на то, из какого таксона взят белок
Сохраняете таблицу заказав нужные колонки в ней.
Одна трудность имеется: в Uniprot нельзя указать в какой последовательности идут домены в белке:
Белок с доменами A > B и другой с доменами B > A попадут в один и тот же запрос. (При сохранении в формате GFF информация для различения есть, но формат GFF не просто разбирать)
Такая ситуация встречается (см. в презентации), но редко. Проще всего обнаружить её, построив выравнивание (п. 2.2.a) и удалив из него неправильные последовательности.
Еще бывает проблема с числом заказанных одинаковых доменов в последовательности; запрос состоит в том, что есть хотя бы один такой домен.
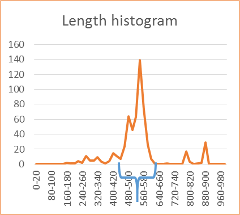
2.1.c Как строить гистограмму длин белков
Гистограммы - см. семестр 1. А теперь вы обязаны понимать что это такое по статистике.
Как определить характерный размер белка из семейства
Основная мода выделена. Пики правее основной моде содержат белки, которые длиннее на 200 а.к. белков из основной моды. 200 а.к. достаточно для формирования дополнительного домена, который, возможно, неизвестен Pfam.
Белки левее моды - вероятные фрагменты полных последовательностей (т.е. ошибки аннотации генов или псевдогены) или содержат крупные делеции, меняющие свойства белка.
Мода в этом примере включает два пика. Намёк но то, что две подгруппы. Но только намек, может случайность.
2.1.d Представительная выборка
В таблице 1 с белками с нужной архитектурой отметьте белки с характерной длиной. В представительную выборку отберите по несколько белков из каждого таксона, например, семейства. Общее правило описать невозможно - зависит от конкретной ситуации. В отчете опишите из каких отделов и семейств выбраны белки. Это будет важно при проверке профиля и обсуждении результата.
2.2.a Скачать последовательности выборки
- Лучше скриптом из Uniprot, зная, что fasta файл с последовательностью белка лежит на html странице Uniprot с адресом:
https://www.uniprot.org/uniprot/P63015.fasta
Здесь P63015 - код доступа (AC) последовательности
можно скачать и таким запросом, составленным в Excel или как-то еще См образец запроса в Excel
Длина строки запроса в Uniprot ограничена. Но 60-100 AC помещается
Вместо запроса, составленного в Excel, крайне рекомендую использовать форму Retrieve/ID mapping, это вполне себе её прямое назначение (одно из двух). Эту же форму можно использовать и из скрипта, будет быстрее, чем по одной записи скачивать. Чуть подробнее тут.
— ИР
2.2.b Ревизия выравнивания состоит в следующем:
Найти самый N-концевой консервативный блок слайды с примерами
- Начало в поз 45 на слайде.
- Удалите все колонки до него (поз. 1-44)
- удалите очевидные фрагменты, те что без N-концевого конс. блока (посл-ти 11 и 13 на слайде; посл. 7 не знаю оставлять или нет
- Найдите последний C-концевой консервативный блок (слайд 2). (На слайде нумерация поз. сбита,т.к. вырезал этот фрагмент для рисунка). Для решительных последняя консервативная позиция имеет номер 19. Для осторожных последняя конс. поз. 49. Я осторожный. При этом я удалил бы 16-ю последовательность, дающую длинную вставку. "консервативные" позиции начиная с 98й считаю ошибкой выравнивателя, т.к. их мало, они отделены от остльных вариабельными по длине участками. А их самих не хватит на структурный элемент белка.
- Удалите все колонки после последней колонки C-концевого консервативного блока. Слайды 3 и 4 - N-конец и C-конец после моей ревизии
2.2.c Создание и калибровка профиля
Для построения профиля используйте пакет HMMER. Он установлен на kodomo. Подсказка ко всем трём программам даётся опцией -h. Более подробную информацию можно получить, выполнив команду man hmm2build (аналогично с hmm2calibrate и hmm2search).
Постройте профиль программой hmm2build.
Откалибруйте профиль программой hmm2calibrate
2.2.d Поиск по профилю среди последовательностей из таблицы 2
Не запускайте hmm2search без опции --cpu=1! По-умолчанию он занимает все ядра процессора, что не допустимо на учебном сервере. Если запускаете в непопулярное у других студентов время, то можете использовать 2 ядра, не больше.
— ИР
- Скачайте из Uniprot все последовательности, содержащие выбранный домен [если вы ограничили таксон, то из этого таксона] в одном .fasta файле
- Выполните поиск по профилю командой hmm2search
- Внесите результат в таблицу 2. В таблице должны быть колонки: "входит в семейство" т.е. из табл. 1 - имеет выбранную доменную архитектуру; "входит в список находок", "вес находки", "E-value" (интересны, но не обязательны: "входит в выборку для построения профиля", "длина белка")
Программы пакета HMMER 2.3.2 (установлен на kodomo)
команда, вход, выход |
что делает |
полезные опции |
комментарии |
hmm2build <выходной файл с профилем> <входное выравнивание> |
Делает профиль по выравниванию |
-g <профиль для глобального выравнивания> |
--- |
hmm2calibrate <файл с профилем> |
добавляет в тот же файл-профиль строчку EDV с коэффициентами пересчета веса в нормализовнный |
--num <число случайных последовательностей, default=5000> |
Генерирует --num случайных последовательностей, строит выравнивание профиля с каждой, считает вес и рассчитывает коэффициенты пересчета |
hmm2search <профиль> <файл с последовательностями> |
находит домены в последовательностях |
-domE <порог E-value для доменов> -domT <порог веса T для доменов> --cpu <число ядер процессора> |
2.2.e выберите порог веса и оцените результат правила
- Сравните список находок с исходной таблицей можно средствами Excel или Python.
- Подберите порог веса (или Е-value) для предсказания того, что находка имеет нужную доменную архитектуру. Для этого
- (1) постройте распределение весов находок (сортировка по убыванию веса; график весов)
- для каждого возможного порога - строчки в списке вычислите чувствительность и специфичность предсказания состоящего в том, что все строчки выше предсказываются имеющими нужную архитектуру доменов, ниже - не имеют.
- Постройте т.н. ROC кривую. Здесь будет ссылка на презентацию с объяснением.
- Подберите порог, дающий наименьшее значение параметра F1. Содержательно, этот параметр позволяет найти порог, при котором наиболее сбалансированы частоты ложно положительных и ложно отрицательных предсказаний. См презентацию.
Дополнительная информация
На kodomo, помимо пакета HMMER 2.3.2, установлен более новый пакет HMMER 3.0. Его программы отличаются отсутствием двойки в названии (например, hmmbuild вместо hmm2build). К сожалению, hmmbuild не принимает выравнивания в обычных форматах (fasta, aln, msf), поэтому с hmm2build работать проще. Впрочем, Jalview умеет сохранять выравнивания в стокгольмском формате, который hmmbuild понимает, поэтому можете работать с ним. Калибровка профиля в HMMER 3.0 не требуется.
В EMBOSS есть оболочка для пакета HMMER 2.3.2. Удобна тем, что стандартный EMBOSS интерфейс. Команды такие ehmmbuild и т.д.
3.* Сравнение деревьев двух доменов из одной доменной архитектуры
Заданий и так много, поэтому советую это дополнительное задание не выполнять:). Но обещаю проверить и оценить, если почему-то вы его выполните.
Это задание на то, чтобы встретиться с проблемами интерпретации филогенетических деревьев.
Возьмите белки из выборки, по которой создавали профиль. Это для того - чтобы листьев на дереве было не много, можно было бы увидеть его на одной странице. Можете как-нибудь иначе составить выборку для дерева.
Выберите два домена - ваш и еще один, обозначим домены X и Y.
В каждой последовательности, по определению, есть и X, и Y.
Из выравнивания вырежьте домен X в отдельный файл X.fasta и домен Y - в другой файл Y.fasta.
Практическая проблема: домены X и Y должны из одного белка должны иметь одинаковые имена последовательности. Лучше всего, в качестве имён выбрать мнемоники таксонов (следите, чтобы в выравнивании не оказалось последовательностей с одинаковыми именами!) При сравнении деревьев нужно следить за листьями из одной исходной последовательности. (В этом месте, если переименовывать вручную, важно, чтобы последовательностей было немного! У меня есть скрипт, можно его попросить)
Постройте филогенетические деревья для X, и для Y одним и тем же методом. Визуализируйте их рядом, лучше листьями друг к другу. Что мы ожидаем? X и Y эволюционировали в составе одного белка1). Даже если скорость эволюции у них разная, то на топологию дерева это не должно было повлиять так как (физическое) время эволюции от узла до узла одно и то же. Значит, деревья должны совпадать.
Так ли это?
Если нет, найдите различия и попробуйте их объяснить. Ошибки алгоритма реконструкции филогении? К домену X в результате перетасовки доменов присоединён домен Y из другого белка в том же геноме, содержащего Y? Горизонтальный перенос - если это прокариоты? Генная конверсия? Или что-то еще. Знание таксономии белков (по мнемонике в названиях листьев) поможет в этом анализе.
1) Такое сопоставление называется танглеграмой (tanglegram). Есть программы для построения танглеграм. Найдите в гугле, или напишите мне - сейчас сразу не вспомню названия программы, но при необходимости найду в курсовой студента.


 2025
2025 2024
2024 2023
2023 2022
2022 2021
2021 2020
2020
 2018
2018 2017
2017