Задание 1
Для выполнения заданий данного практикума я выбрала семейство цитохромов С7
Домен представлен среди видов бактерий.
Выравнивание последовательностей белков, содержащих домен, было получено с помощью программы JalView и покрашено BLOSUM62 by conservation 20.
Для белков Q74CB4_GEOSL и H5EAE4_ECOLI доступны 3D структуры и аннотированы участки вторичной структуры в последовательности. Проект
Для дальнейшей работы были выбраны 2 наиболее представленных доменных архитектуры
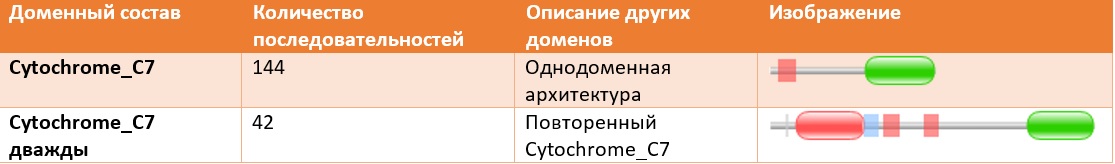
Таблица 1 Информация о выбранных архитектурах
Судя по LOGO домена, доступного на странице записи PFAM, для домена характерны только три небольших участка сильной консервативности, в которых расположены по 2 цитозина (разделенных двумя неконсервативными остатками) с гистидином, идущим непосредственно за вторым цитозином.
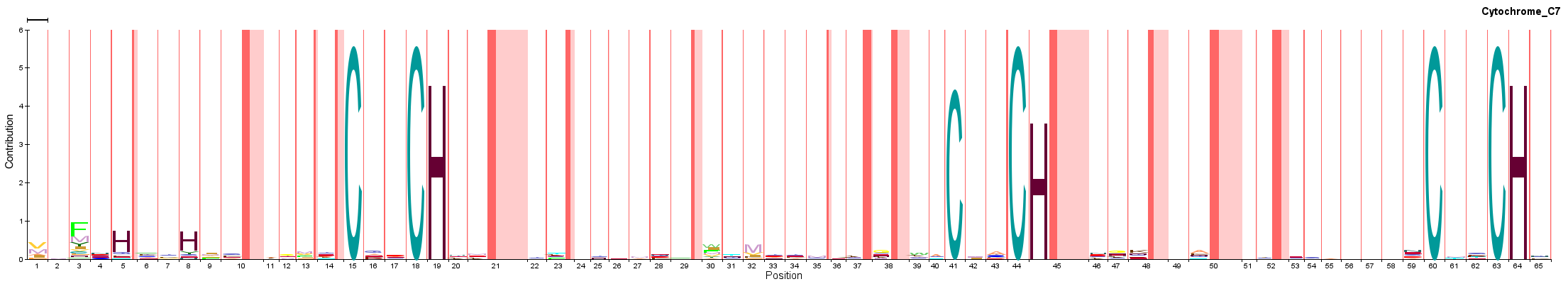
Рисунок 1 LOGO
Была получена выборка представителей, по 13 белков каждой архитектуры каждого подтаксона, всего 52.
Также отдельно был добавлен белок с известной структурой (он относится к типу 1_D, т.е. первая доменная архитектура, подтаксон дельтапротеобактерии).
Так как вторая доменная архитектура состоит из двух одинаковых рассматриваемых доменов, было построено два выравнивания - для домена второй архитектуры, расположенного раньше по последовательности, и дальше.
Полученные выравнивания были открыты в Jalview, созданы группы и раскрашены с помощью ClustalX.
Были удалены фрагменты доменов (неполные последовательности), невыровненные N- и C- концевые участки, выбивающиеся последовательности.
Итоговые выравнивания после обработки имели такой состав: первое 13 1G, 12 1D, 9 2G, 10 2D; второе 13 1G, 12 1D, 12 2G, 10 2D.
Задание 2
Для построения деревьев была использована программа MEGA, метод Maximum Likelihood с бутстрепом с числом итераций 300.
Деревья представлены на рис.1 и рис.2. Красный - 1G, желтый - 2G, голубой - 2D, синий - 1D. Скачать
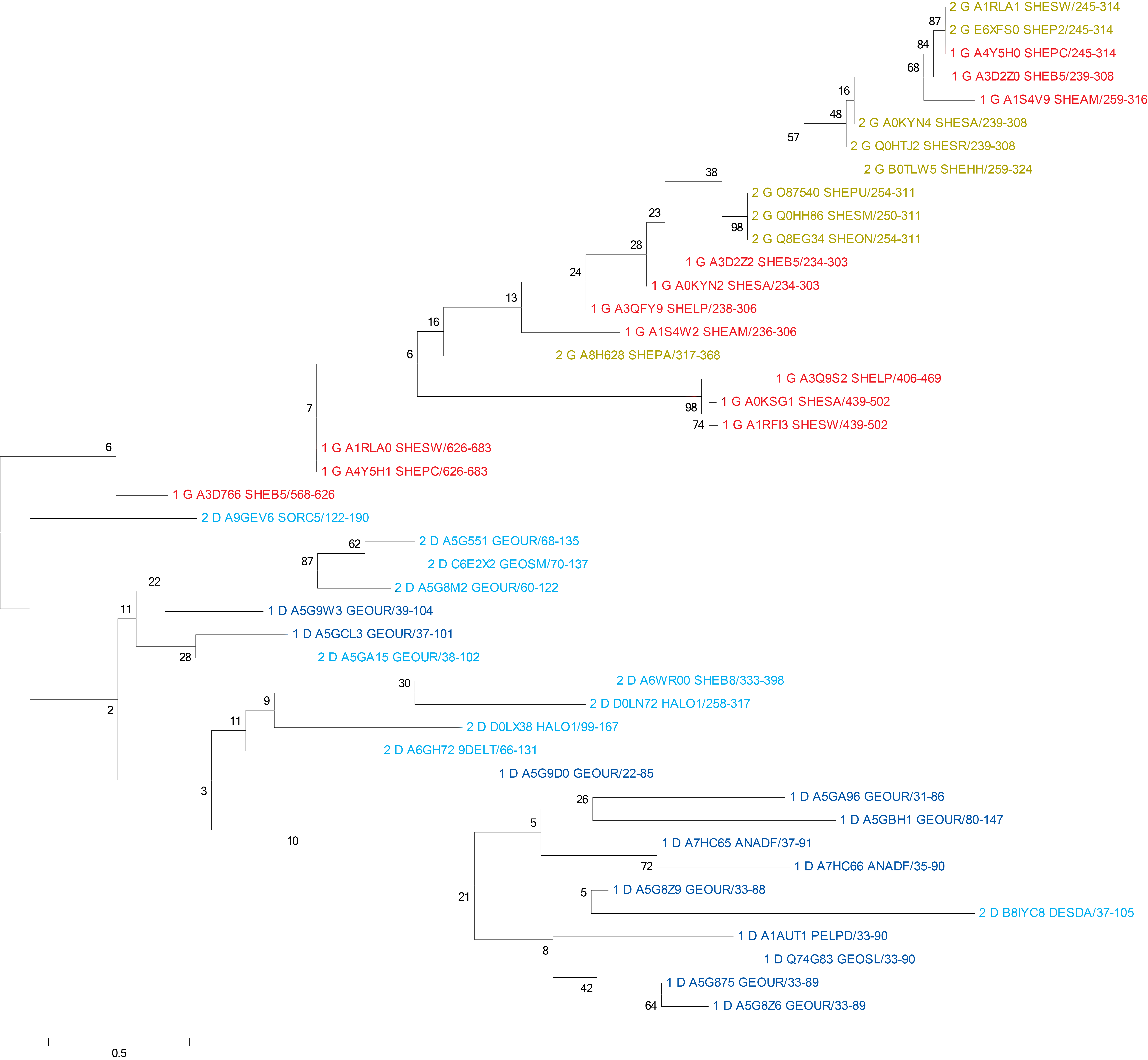
Рисунок 2.a Дерево, построенное по выравниванию домена Cytochrome_7 однодоменной архитектуры и первого из двухдоменной.
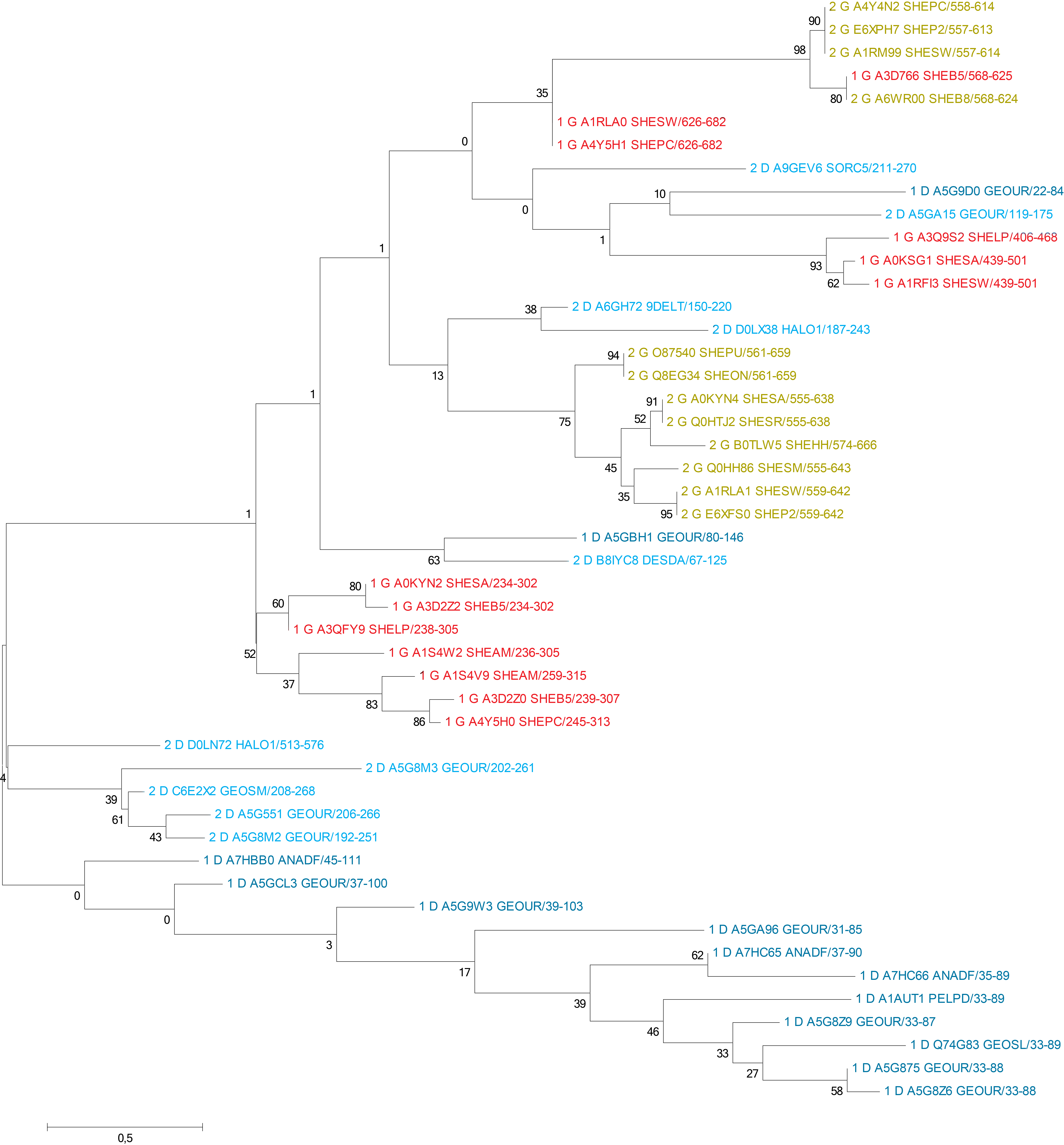
Рисунок 2.б Дерево, построенное по выравниванию домена Cytochrome_7 однодоменной архитектуры и второго из двухдоменной.
На первом дереве видно, что таксоны разошлись гораздо раньше, чем доменная архитектура, а она, в свою очередь, изменялась независимо много раз.
На втором же дереве разделение на таксоны не столь четкое, некоторые представители дельтапротеобактерий попали к гаммапротеобактериям.
Возможно, вторая копия домена у представителей двухдоменной архитектуры среди дельт менее функционально важна и поэтому имела меньшие ограничения на мутагенез, из-за чего стала более похожей на этот домен в другом подтаксоне, но это не объясняет вылеты дельт из своей клады в случае их однодоменной архитектуры.
Задание 3
В качестве подсемейства была выбрана клада представителей 1G, хорошо отделенных на обоих деревьях и с хорошей бутстреп-поддержкой. Это A3Q9S2, A0KSG1, A1RFI3.
По ним в дальнейшем был построен профиль и проведен поиск по всем последовательностям с этим доменом. В качестве p-value было выбрано значение 0,0055.
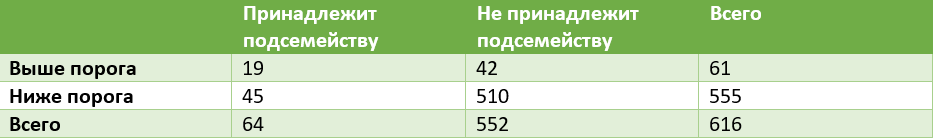
Таблица 2 Результаты поиска
При данном пороге e-value точность (precision) равна 31,15%, чувствительность (sensitivity) 29,7%, специфичность (specificity) 92,4%.
Была построена roc-кривая. По ней видно, что хотя предложенный профиль позволяет отобрать представителей подсемейства, качество его работы оставляет желать лучшего, т.к. площадь под кривой весьма мала.
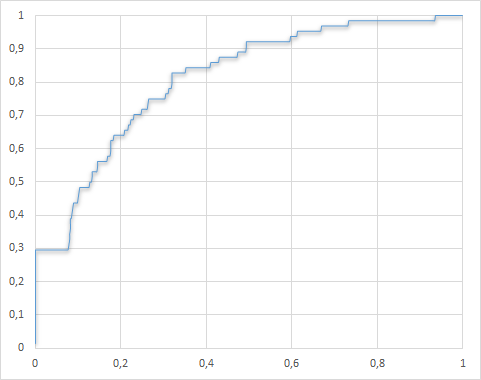
Рисунок 3 ROC-кривая
Скачать итоговую таблицу с полезными результатами