BLAST для нуклеотидных последовательностей
Определение таксономии и функции прочтенной последовательности

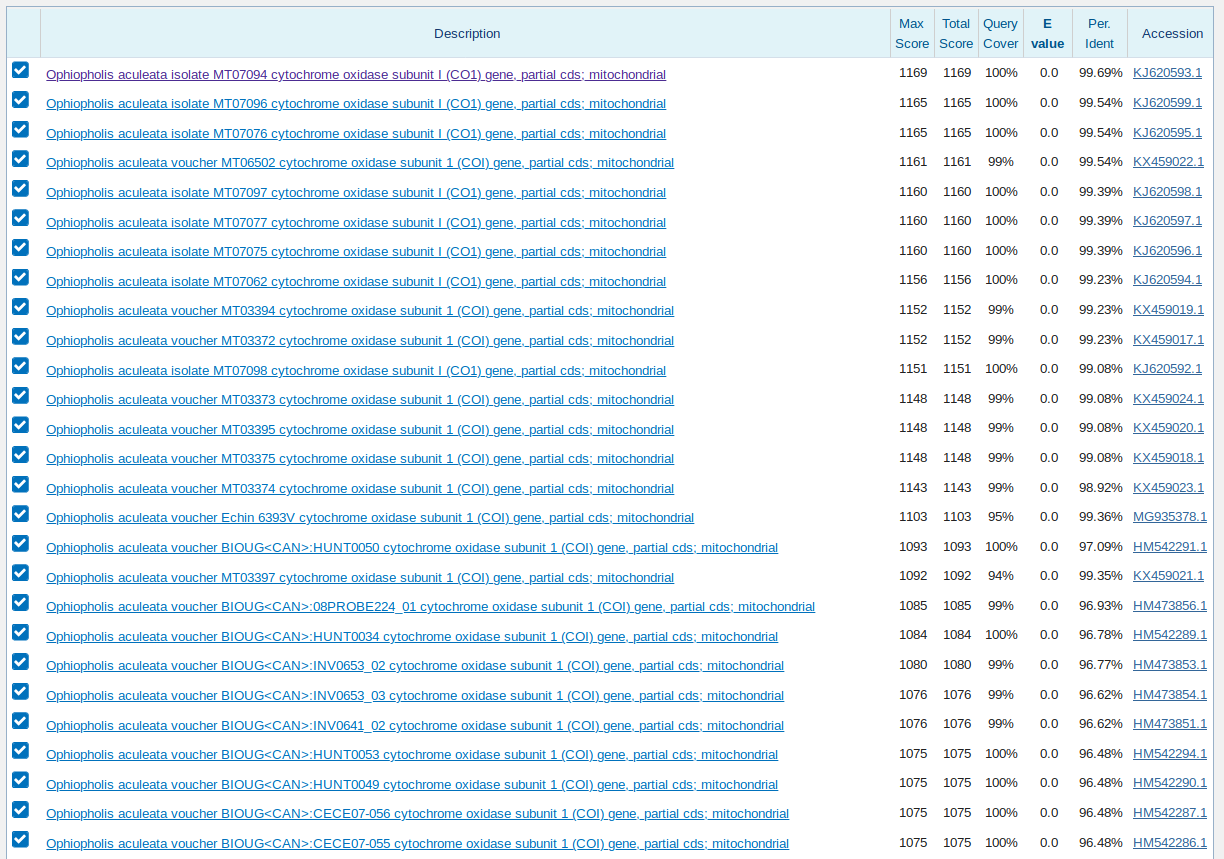
С помощью blastn с параметрами по умолчанию было установлено, что данная последовательность кодирует субъединицу I цитохром c-оксидазы (CO1)
и принадлежит организму Ophiopholis aculeata из класса Офиуры, тип Иглокожие (см. Рис. 1).
Это не вызывает никаких сомнений, так как первые 45 выравниваний blastn указывают именно на это,
при этом E-value=0.0, процент идентичности >95% (см. Рис. 2).
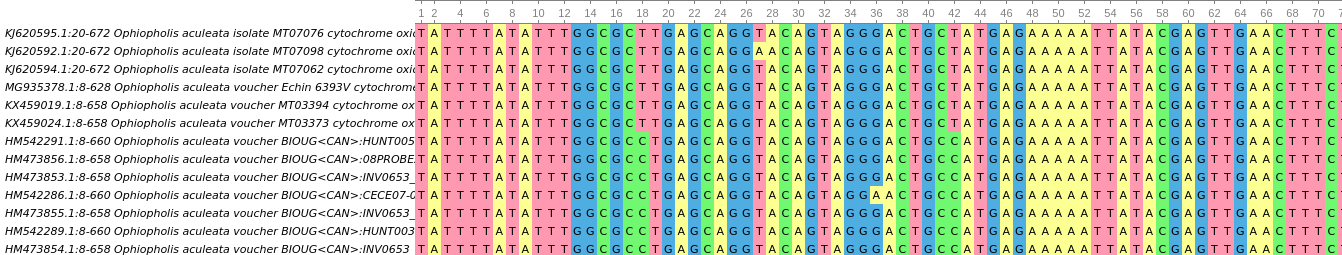
То, что есть несовпадения в отдельных позициях, нестрашно, так как сами найденные последовательности гена CO1 имеют SNP (см. Рис. 3).
Сравнение вариантов nBLAST
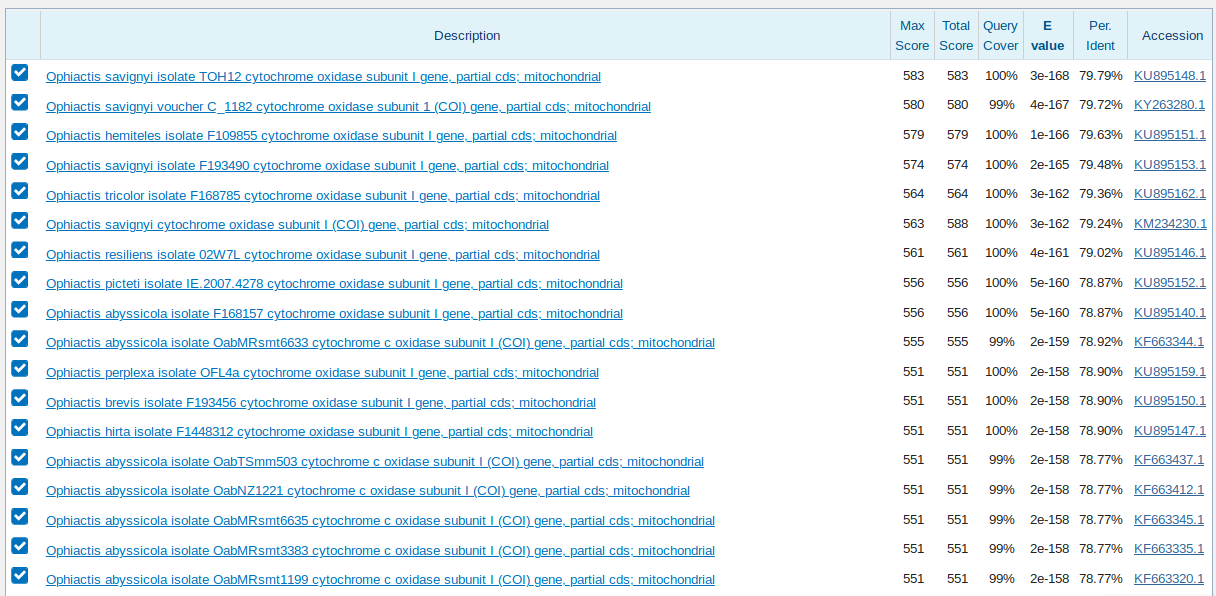
I. Последовательность гена Ophiopholis aculeata из первого задания (из практикума по Сэнгеру). Для всех вариантов поиска было поставлено
ограничение, что организм должен принадлежать семейству Ophiactidae, но не роду Ophiopholis.
Варианты:
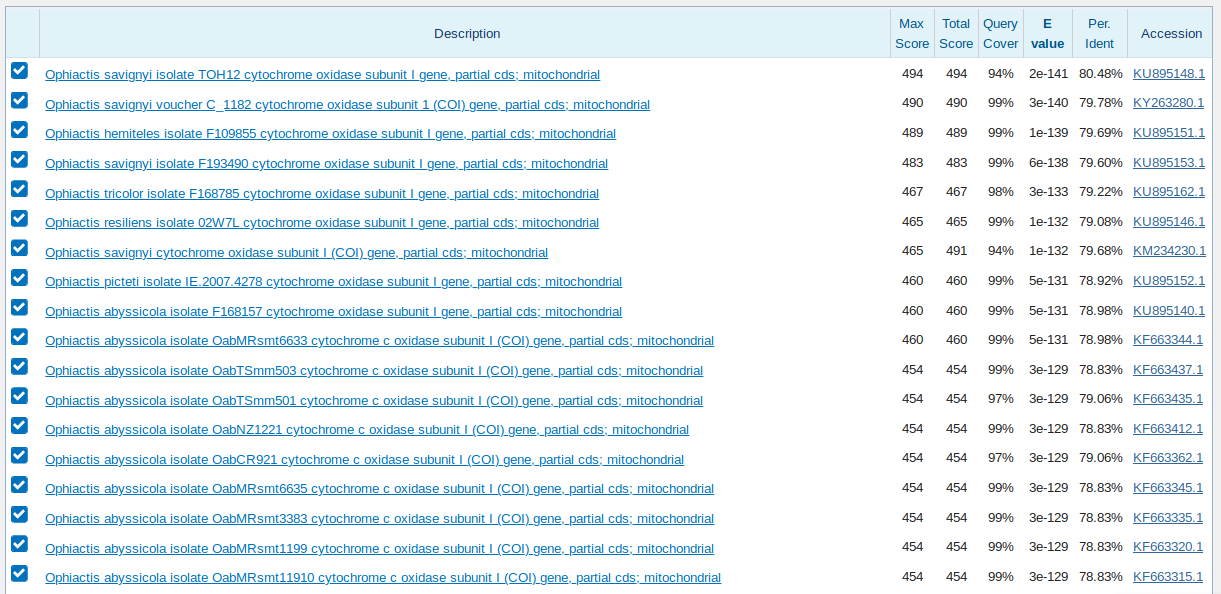
1. blastn с параметрами по умолчанию (см. Рис. 4).
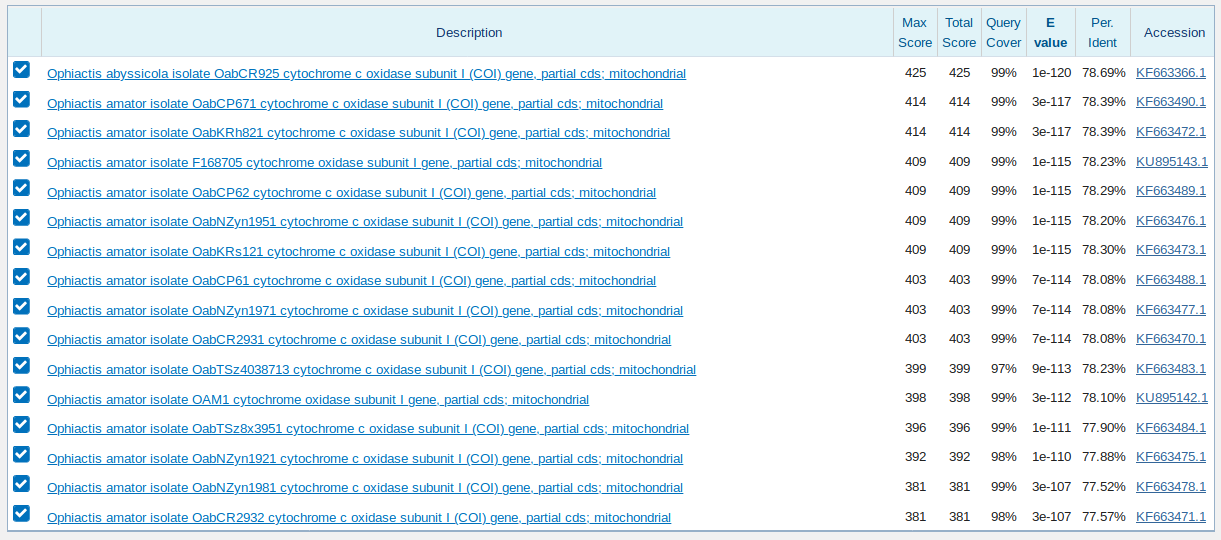
2. blastn с параметрами Word Size (WS) = 7, Match/Mismatch Scores (MMS) = 1,-2 (см. Рис. 5)
3. megablast (см. Рис. 6).
Во-первых, сравнение выдачи демонстрирует, что для данного поиска большой разницы в параметрах алгоритма blastn нет (не помогает это исправить
и введение дополнительных ограничений на таксономию, я проверял). Однако стоит заметить, что для одних и тех же находок вариант
blastn с уменьшенными штрафами и меньшим размером слова дает E-value на сколько-то порядков больше. И порядок последовательностей, отсортированных
по E-value, тоже несколько меняется.
В то же время, megablast выдал всего 15 последовательностей, причем с еще меньшим E-value. Любопытно, что
ни одна из находок не попала в топ находок blastn, и все они, кроме одной, принадлежат одному и тому же виду Ophiactis amator. Видимо,
его вариант гена субъединицы 1 цитохром c-оксидазы имеет больше именно длинных (28 нуклеотидов) полных или почти полных совпадений с геном этой же
субъединицы из Ophiopholis aculeata по сравнению с генами из видов, попавших в выдачу blastn.
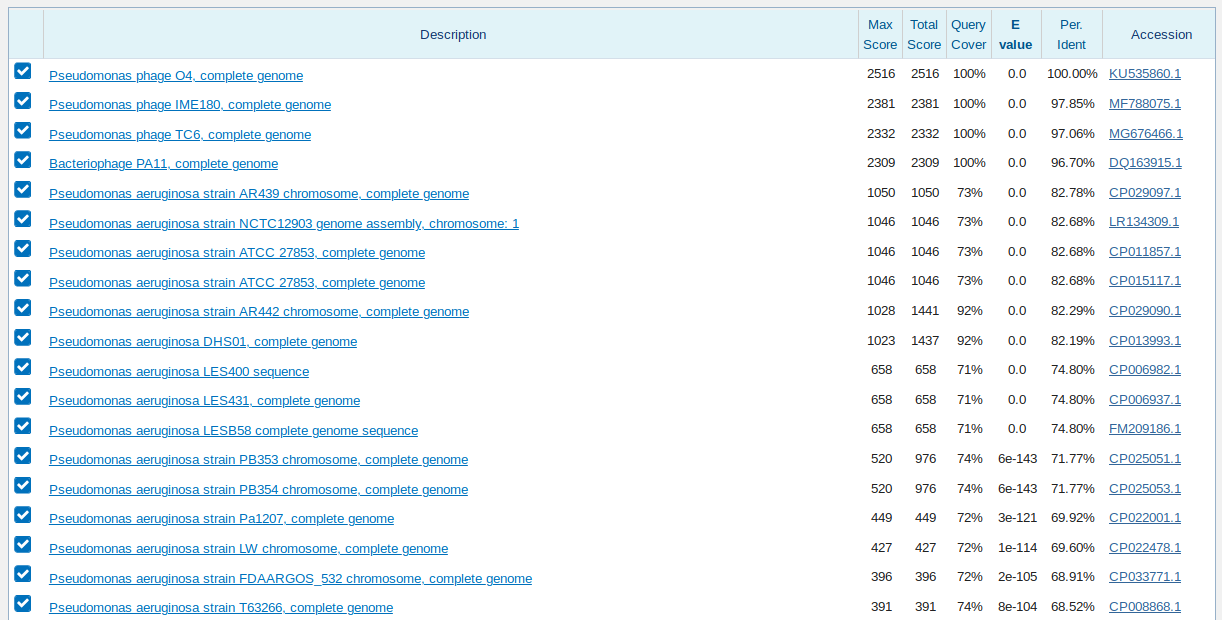
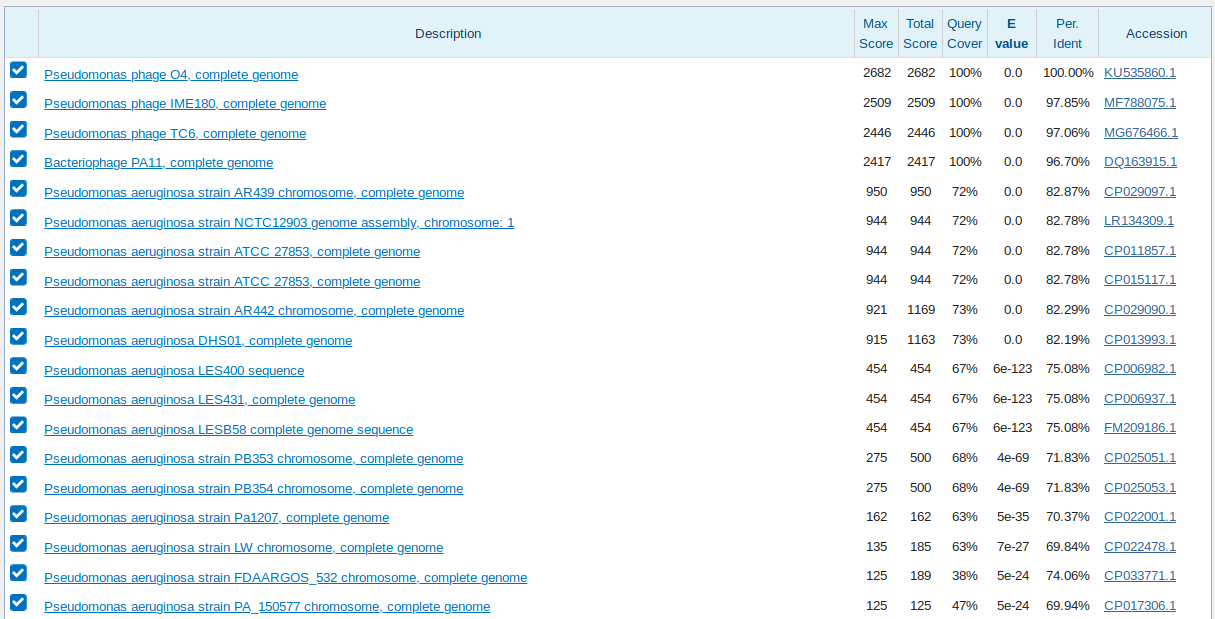
II. Последовательность одной из CDS из генома вируса Pseudomonas phage O4, а именно:
>lcl|NC_031274.1_cds_YP_009304482.1_3 [locus_tag=BJD45_gp03] [db_xref=GeneID:29126554] [protein=putative pectate lyase] [protein_id=YP_009304482.1] [location=1352..2746] [gbkey=CDS] ATGAGTACGTTGAGAGTAGACACTCTACAAAAACCTGATAACAGCGTCACTGTTAATCTGGAAGATATCA TTACGGAAGTGAATCTAGGTGATACCACTTCCCCCACTAAAGGAGCTGGCCTAGTAGGTTTCTCTAGTAG CGTTAGCTATCCTGATGGTACTATAGGGGCTGAAATCCTAGAGGTAAGCTCTAAGATTGGCGTTACAGTA AAAGACTATGGTGCTGTTGGTGATGGCGTCACTGATGATACTGTAGCAATTCAAGCAGCCATCACTGCGG TTGGACAATATAAAGACTTAGGTTGGCCTCGTGGTACCTACCTAATTACTCAGAAGTTGGTAATGCCAAC TGGCCAAAGATGGATAGGTTCTGGTGGCCAACGTGGTGCCACTATCAAGAAGGGCGCCAACATCGACATG GTAGAAGTAGGTACTCTGTGTACTGTTCTTGATATTAGTCTAGAAGGTGAGGGAGCTACTTACTCCGGTA AAGGGTTTGTAATTGCGTCTGGCTTCAGTCAGAACCTAGTGCGTTGCAGAGCCGTTAACATGGGTGGTGA ACCGCTATACTTCGCTAATAACGCAGGTGGCGGTGCAAACGTAACAGCGTTTGAAGGTTACCCTGTAGAT ACGGATACTTACGCTGGGTGCGTTATTGCAGGAGATACTTCTCCTCATCCTCGGTTCTTCCGTGGTATGT GGTTGAGCGGTGCTAACTTTGCCCTTGGCCCTGGTGCAGGTAATGGCGGATCGATGACTCAATTCTACAT TAGGGATTTGAGGTTTGATCCTACTTCAACTCTGTTCCACATTTCCAATGGACGTTGTGCAACTCTTGGA GCCACGACGACTCTTAAGGGTTTCGATCACTCAATTGATGGCGTTGCTTTTGCAGGTCCAGTTGCACTCG ATTCTGCTCAAGGCATTAACCTTGGTCCGTCTTGCTCTGTACCGTCACTCACGGAGAATGCGACTAACTC GCAATACAACTCCGTCTATGTACAGCGTAGAACCTACACTCCAACTTGGACGCAGACTAGTGCTACTCCT GCAATTGGCAATGGTACCTTAACTGGTAACTATGTCCGAGCTGGCCATATGTGTCATGTGCAGATTGAAT TGGTAGCAGGTTCGACTACGACCTTCGGTGATGCTGCATCAGGCTATCGTTTCTCTCTGCCGTTCCCTGG CCACCTGTCTTTCAACCAGAGGGGATTCCCTGTGCGAATCTACGACACTAGTGCTAGTGCAGATTTCACT GGGTGGGCATCCATTGGTGCGGGCCAAGATTACATCACCATCTCTGTGGGAGCACAGCAGGTTCGTTCTA CGTCACCTATGACTTGGGCCACTGGGGATACGCTGCATTGCTCGTTCTCCTATATGACCCGCTAA
Все варианты поиска нашли сам геном, из которого была взята CDS, а также еще 3 генома других бактериофагов. E-value=0 для всех этих находок.
Далее идут геномы различных штаммов Pseudomonas aeruginosa, в геном которой встраиваются эти фаги. Все они содержат только часть (без начала)
искомой последовательности. Для этих находок E-value, как и в случае с последовательностью из Ophiopholis, меньше всего для bastn с
параметрами по умолчанию, на сколько-то порядков выше для blastn с пользовательскими параметрами и самый высокий для megablast (впрочем,
значение все еще достаточно мало, чтобы говорить о значимости находки).
Число находок для разных вариантов алгоритма см. в Таблице 2. Максимальный размер выдачи был ограничен пятьюдесятью для сокращения времени
работы алгоритма.
| blastn по умолчанию | blastn с параметрами | megablast | |
|---|---|---|---|
| Последовательность из Ophiopholis aculeata | 50 (max) | 50 (max) | 16 |
| CDS Pseudomonas phage O4 | 50 (max) | 50 (max) | 14 |
Проверка наличия гомологов белков в неаннотированном геноме
Так как организм Amoeboaphelidium protococcarum в некоторой степени близок грибам, для поиска гомологов я брал последовательности белков
Saccharomyces cerevisiae. Белки искал в Uniprot, а именно в Swiss-Prot. Поиск гомологов в геноме проводил с помощью tblastn через программу UGENE
(порог на E-value: 0.05).
Были выбраны следующие белки:
1. Clathrin heavy chain (Uniprot ID: P22137).
Клатрин участвует в образовании большинства везикул в клетке эукариот, поэтому должен присутствовать у всех
эукариот за редкими исключениями. Он состоит из трех тяжелых и трех легких цепей, я взял последовательность тяжелой цепи.
Были найдены 2 гомолога (в скэффолдах 105 и 277), процент идентичности обоих чуть выше 50%, однако E-value=0, а оба выравнивания
были построены для практически полной последовательности белка, что говорит о том, что это, вероятно, действительно гомологи,
просто с большим числом одиночных замен в последовательности (см. Рис. 10).
2. Actin (Uniprot ID: P60010).
Актин - один из главных белков цитоскелета, именно из него состоят микрофиламенты, присутствующие в цитоплазме всех эукариотических клеток.
Они участвуют в изменении формы клетки, а вместе с миозином осуществляют сокращения, в т.ч. мышечные. Поэтому следовало ожидать, что
и у A. protococcarum актин имеется.
tblastn подтвердил это предположение. Найдено 17 последовательностей с E-value меньше 2e08, однако некоторые соответствуют
только части последовательности. Поэтому предполагаемых гомологов всего 5.
На Рис. 11 представлены 19 гомологов, так как изображение получено с помощью программы UGENE,
в которой невозможно указать генетический код локальной базы данных при поиске с помощью tblastn, поэтому результат работы
алгоритма слегка отличается от результата tblastn, запущенного из командной строки с указанием генетического кода.
3. Exportin-T (Uniprot ID: P33418).
Экспортин T участвует в экспорте из ядра аминоацилированных тРНК. Он есть и у грибов, и у животных, и у растений, и следовало ожидать, что
гомолог дрожжевому экспортину T найдется и у A. protococcarum. Однако tblastn не дал ни одной находки с E-value меньше единицы.
Я не могу предложить этому разумного объяснения.
Поиск гена белка в контиге
Была взята последовательность CAWI020039955.1 длиной 26,802 b.p. контига 1703 из генома Adineta vaga из моего предыдущего практикума.
Поиск генов белков был произведен с помощью blastx (для сокращения времени работы алгоритма только по базе Reference proteins, царству Animalia).
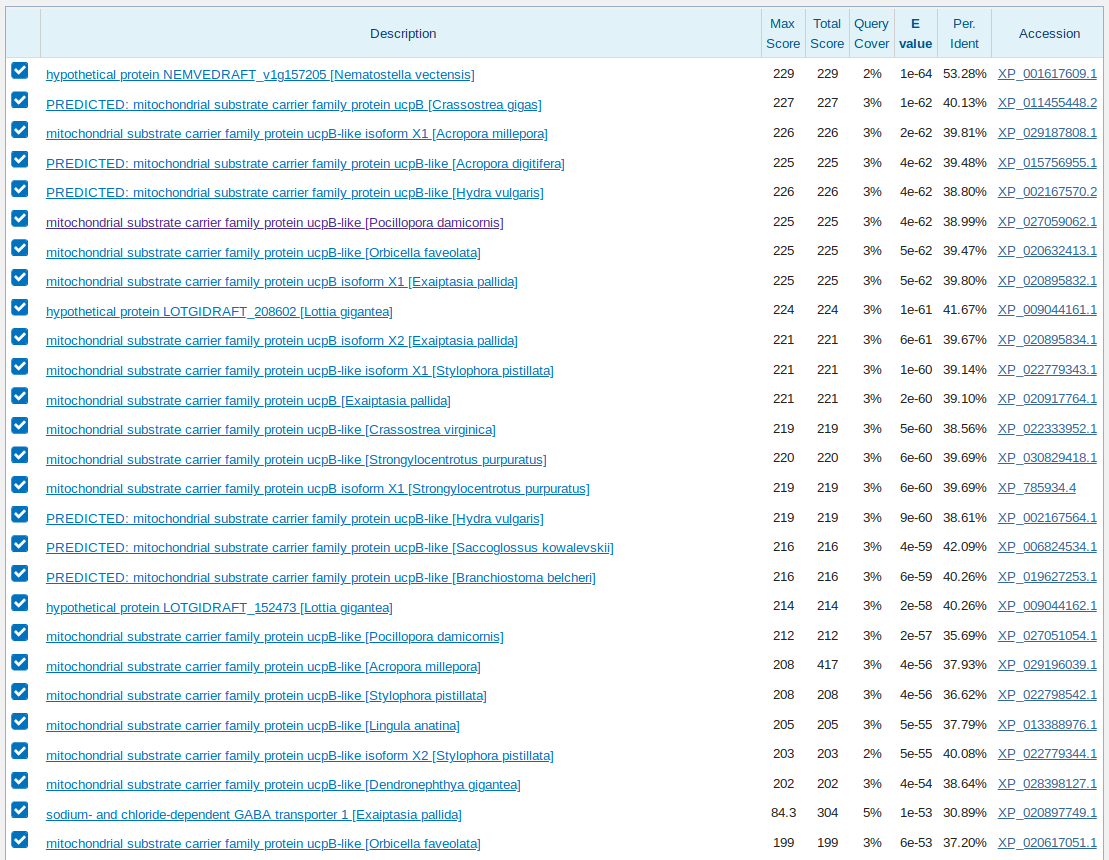
Предположительно был найден ген белка-переносчика через внутреннюю мембрану митохондрий (см. Рис. 12). Несмотря на большое число
находок blastx с низким E-value, процент идентичности с данными находками достаточно низкий (около 40%).