Поиск и описание полиморфизмов
Картирование ридов на хромосому 5 и аннотация полиморфизмов
Скачать все команды одним bash скриптом.
| hisat2-build chr5.fasta chr5 | Индексирование референса |
|---|---|
| fastqc chr5.fastq | Проверка качества чтений |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 chr5.fastq chr5_trimmed.fastq TRAILING:20 MINLEN:50 | Очистка чтений |
| fastqc chr5_trimmed.fastq | Проверка качества после очистки чтений |
| hisat2 -x chr5 -U chr5_trimmed.fastq -S chr5_align.sam --no-softclip --no-spliced-alignment | Выравнивание прочтений и референса |
| samtools view -b chr5_align.sam -o chr5_align.bam | Перевод выравнивания в бинарный формат |
| samtools sort chr5_align.bam chr5_align_sorted | Сортировка выравнивания по координате в референсе |
| samtools index chr5_align_sorted.bam | Индексирование отсортированного файла |
| samtools flagstat chr5_align.sam | Получение основной информации о картировании, в т.ч. числа откартированных чтений |
| samtools mpileup -uf chr5.fasta chr5_align_sorted.bam -o chr5_snp.bcf | Создание файла с полиморфизмами |
| bcftools call -cv chr5_snp.bcf -o chr5_snp.vcf | Перевод полученного файла в текстовый формат |
| vcftools --vcf chr5_snp.vcf --remove-indels --recode --out chr5_snp_noind | Удаление инделей из vcf-файла |
| convert2annovar.pl -format vcf4 chr5_snp_noind.recode.vcf -outfile chr5_snp.avinput | Получение входного файла для annovar |
| annotate_variation.pl -out chr5_refgene -build hg19 chr5_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по базе данных refGene |
| annotate_variation.pl -filter -out chr5_dbsnp -build hg19 -dbtype snp138 chr5_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по базе данных dbsnp |
| annotate_variation.pl -filter -dbtype 1000g2014oct_all -buildver hg19 -out chr5_1000genomes chr5_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по базе данных 1000 genomes |
| annotate_variation.pl -regionanno -build hg19 -out chr5_gwas -dbtype gwasCatalog chr5_snp.avinput /nfs/srv/databases/annovar/humandb.old/ | Аннотация по базе данных Gwas |
| annotate_variation.pl chr5_snp.avinput /nfs/srv/databases/annovar/humandb.old/ -filter -dbtype clinvar_20150629 -buildver hg19 -out chr5_clinvar | Аннотация по базе данных Clinvar |
Анализ качества и очистка чтений.
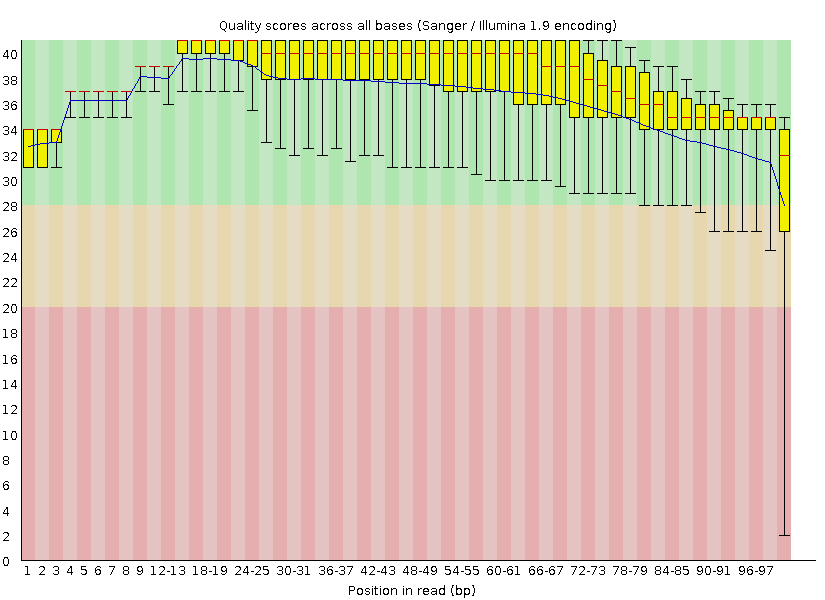
Исходно было получено 8208 чтений. Качество чтений до очистки отображено на Рис. 1.
С помощью Trimmomatic с конца каждого чтения были отрезаны нуклеотиды с качеством ниже 20,
после чего оставлены только чтения длиной не меньше 50 нуклеотидов. После очистки осталось
8114 ридов.
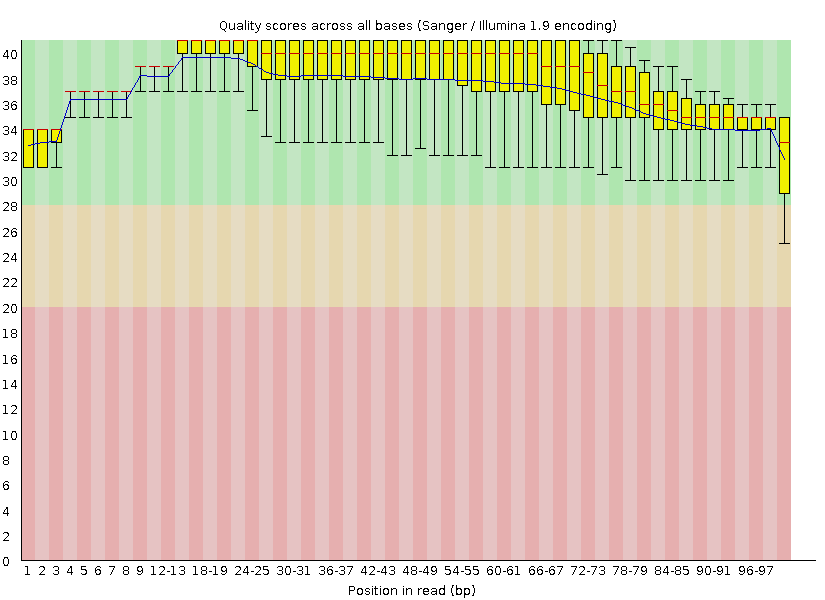
Качество чтений после очистки отображено на Рис. 2. Видно, что выбросов стало меньше.
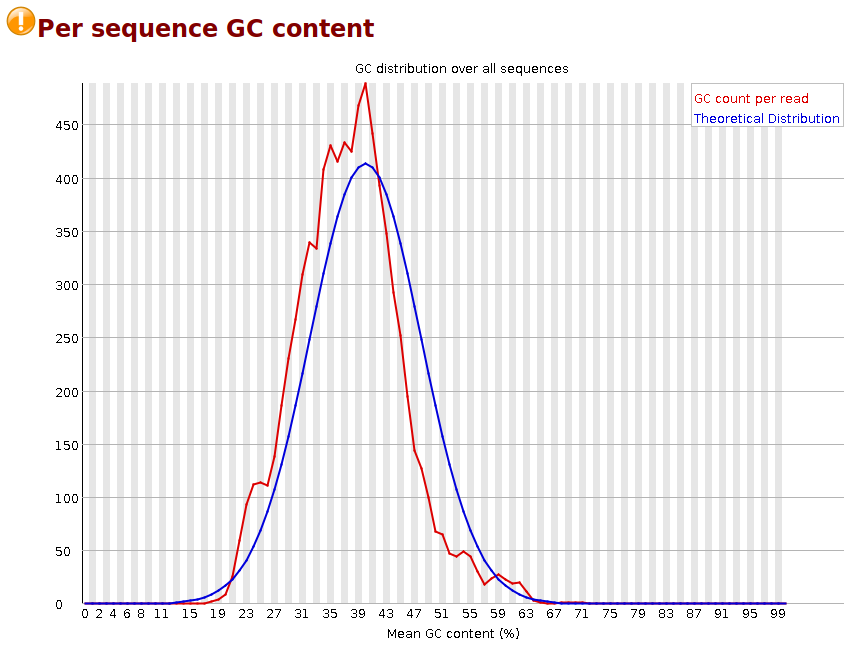
Также качество чтений можно проверять по распределению GC-контента ридов. В теории он должен
быть распределен нормально. Однако в нашем случае от нормального распределения есть отклонения
(см. Рис. 3).
Картирование ридов и аннотация полиморфизмов.
8084 (99.63%) чтений были картированы на 5 хромосому. Качество картирования
высокое. Описание трех из 32 полиморфизмов даны в Таблице 2.
Всего из 32 полиморфизмов: 28 SNP, 4 indels. В целом, покрытие и качество полиморфизмов хорошее,
это видно при визуализации полиморфизмов с помощью Integrative Genomics Viewer (пример см. Рис. 5).
База данных refGene делит snp по их расположению в последовательности.
Из моих 4 оказались экзонными, 24 - интронными, а один - в
3'-нетранслируемой области.
Из экзонных snp 3 попали в ген IL7R (рецептор интерлейкина-7),
а один - в ген CAPSL (кальцифозин-подобный белок).
Нуклеотидные замены: T197C (IL7R, exon2), G412A (IL7R, exon4), C731T (IL7R, exon6), G254A (CAPSL, exon3).
Соответствующие им аминокислотные замены: I66T, V138I, T244I, R85Q.
24 из 28 SNP имеют reference SNP ID number (rs).
Для 23 SNP, обнаруженных в базе данных 1000genomes,
средняя частота равна 0.42. Есть несколько редких интронных
полиморфизмов, а самый редкий полиморфизм из экзонных - по позиции 35874575 (частота 0.172524).
Согласно базе данных GWAS (genome-wide association studies), три SNP ассоциированы
с повышенным уровнем липопротеинов низкой плотности, а другие два - с диабетом I типа.
База данных ClinVar содержит информацию о трех SNP,
все из которых в гене IL7R. Два полиморфизма предположительно патогенны, один - неизвестно.
| Полиморфизм 1 | Полиморфизм 2 | Полиморфизм 3 | |
|---|---|---|---|
| Координата | 35857177 | 35857308 | 74652239 |
| Тип полиморфизма | Замена | Вставка | Делеция |
| Референс | G | T | CAG |
| Чтения | C | TC | C |
| Глубина покрытия | 113 | 40 | 9 |
| Качество чтений | 221.999 | 177.458 | 68.4664 |