Предсказание структуры тРНК и комплексы ДНК-белок
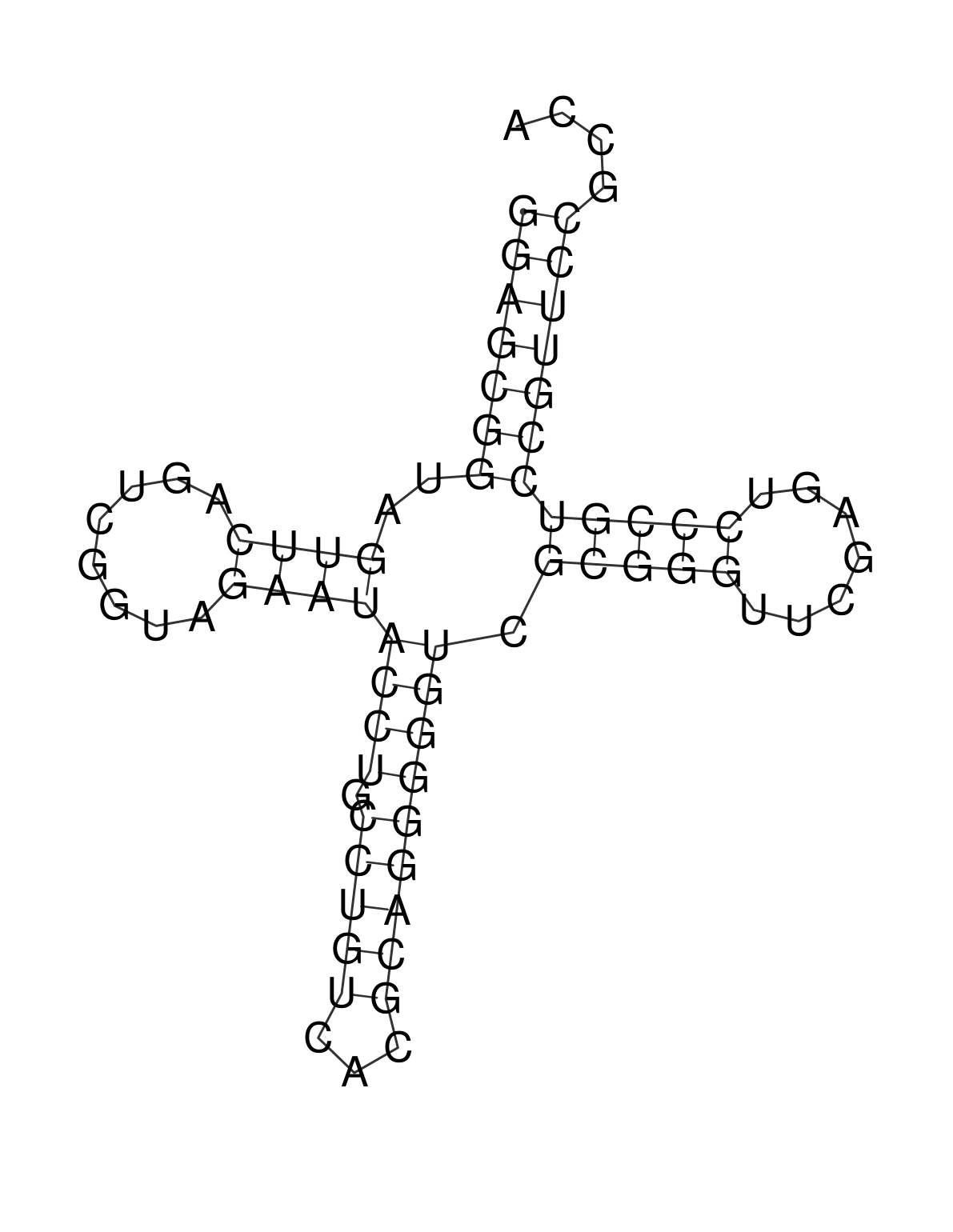
Предсказание вторичной структуры заданной тРНК
Структура тРНК была получена двумя способами: с помощью программы einverted из EMBOSS (с параметром threshold=0), которая ищет инвертированные участки
в нуклеотидных последовательностях, и
по алгоритму Зукера с помощью программы RNAfold из пакета Viena RNA Package.
Результаты доступны в Таблице 1 и на Рис. 1.
| Участок структуры | Позиции в структуре | Предсказание с помощью einverted | Предсказание по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 5'-601-607-3' 5'-666-672-3' |
- | 5'-601-607-3' 5'-666-673-3' Полное совпадение |
| D-стебель | 5'-610-613-3' 5'-622-625-3' |
- | 5'-610-613-3' 5'-622-625'3' Полное совпадение |
| T-стебель | 5'-649-653-3' 5'-665-661-3' |
5'-644-651-3' 5'-656-663-3' В целом, положение совпадает, но длина и конкретные координаты предсказаны неверно |
5'-649-653-3' 5'-665-661-3' Полное совпадение |
| Антикодоновый стебель | 5'-626-632-3' 5'-638-644-3' |
5'-627-634-3' 5'-636-643-3' Координаты практически верные, только немного смещены |
5'-626-635-3' 5'-639-647-3' Антикодоновый стебель по алгоритму Зукера получился длиннее и с петлей меньшего размера, чем в реальной структуре |
| Общее число канонических пар нуклеотидов | 19 | 13 | 22 |
Поиск ДНК-белковых контактов в заданной структуре
Для структуры комплекса ДНК с фактором транскрипции (PDB_ID: 1lq1) были написаны скрипты, выделяющие
отдельные группы атомов, а также визуализирующие их.
1. Определение групп атомов: sets_define.spt
2. Последовательное изображение структуры комплекса, только ДНК и визуализации всех групп атомов:
dna_prot.spt
3. Скрипт для анализа контактов разных типов: interactions.spt
Результаты анализа контактов в комплексе доступны в Таблице 1.
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 3 | 64 | 67 |
| остатками фосфорной кислоты | 35 | 41 | 76 |
| остатками азотистых оснований со стороны большой бороздки | 16 | 28 | 44 |
| остатками азотистых оснований со стороны малой бороздки | 17 | 43 | 60 |
Таким образом, 2'-дезоксирибоза образует с атомами белка, в основном, неполярные контакты, в то время как остатки фосфорной кислоты связаны с белком и полярными, и неполяными контактами. Со стороны малой бороздки в данном ДНК-белковом комплексе больше контактов белка с остатками азотистых основаниями, чем со стороны большой бороздки. При этом с остовом ДНК контактов больше, чем с остатками оснований.
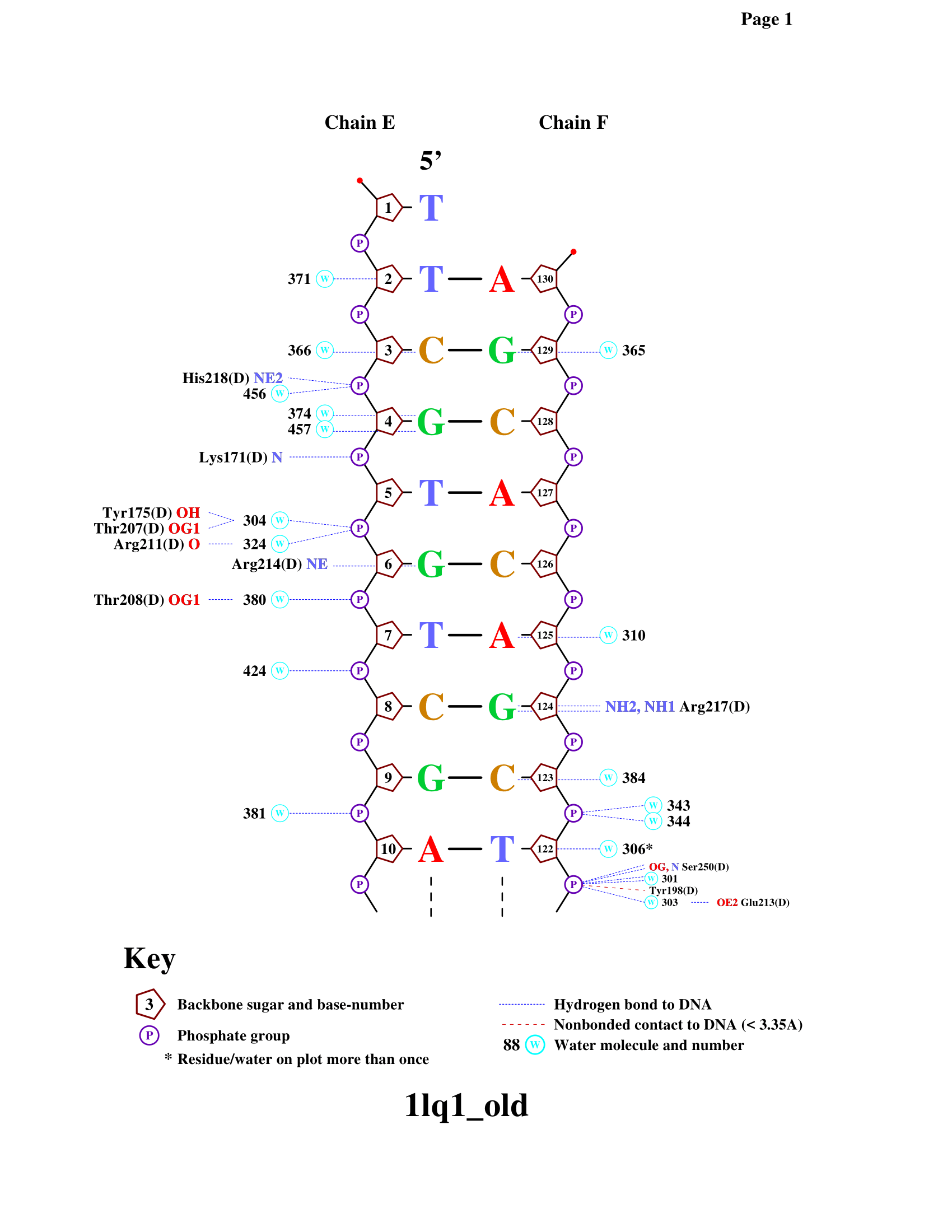
Контакты белка с ДНК можно визуализовать также с помощью программы nucplot. Полученные изображения - см. Рис. 2.
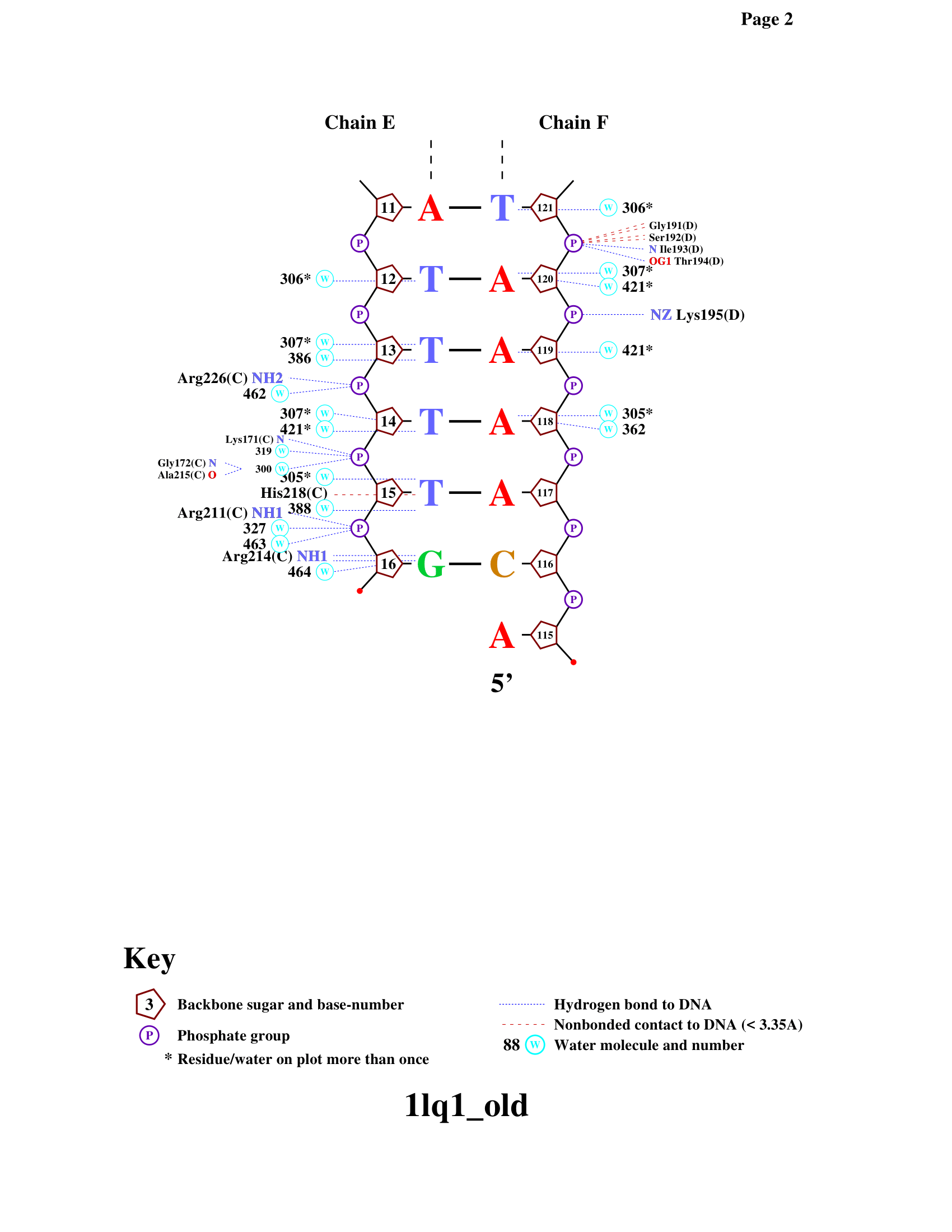
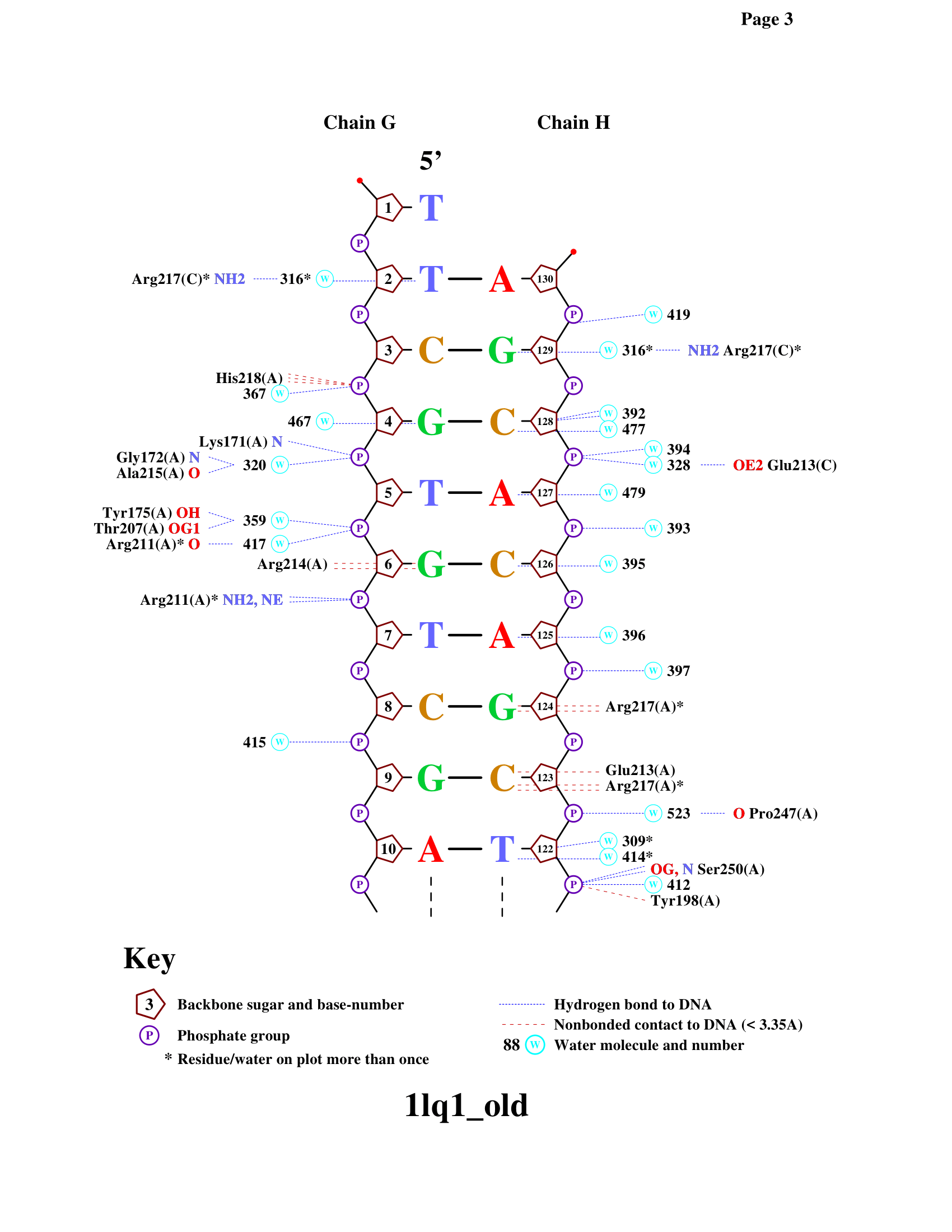
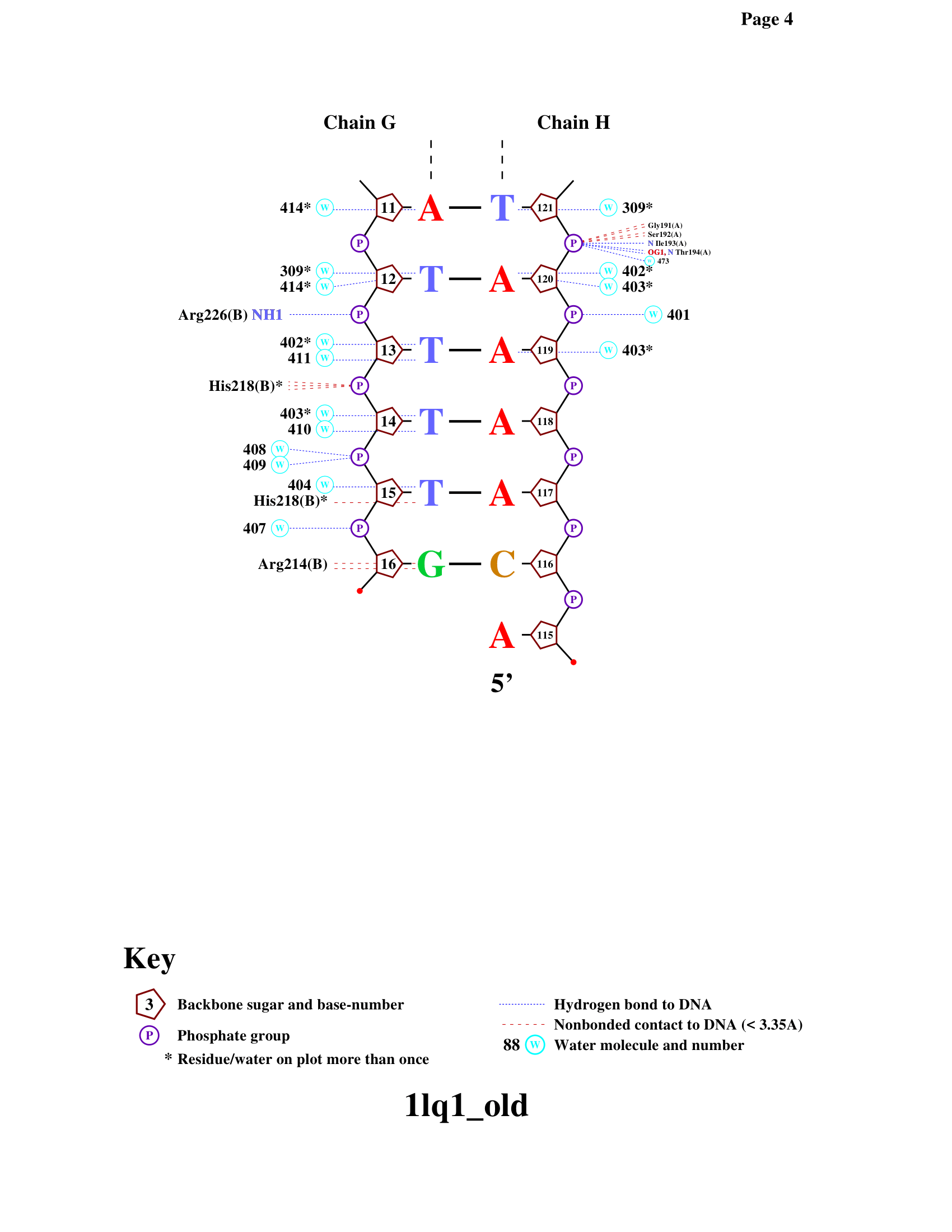
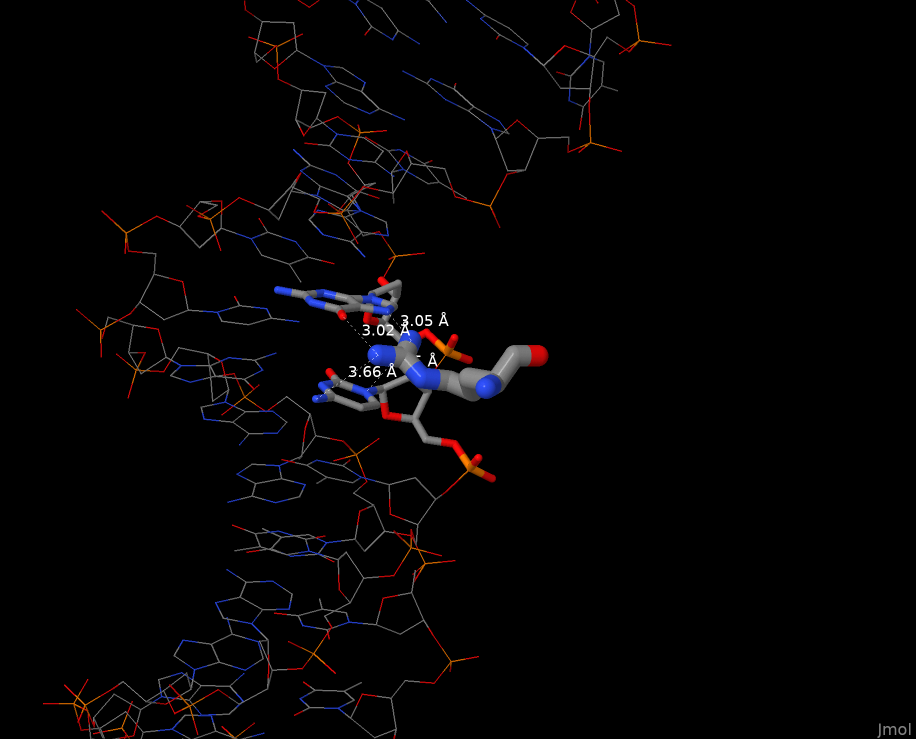
Наибольшее число контактов с ДНК образует остаток аргинина 217 цепи A (см. Рис. 3).
Он связывается с гуанином 124 и цитозином 123 цепи H ДНК.
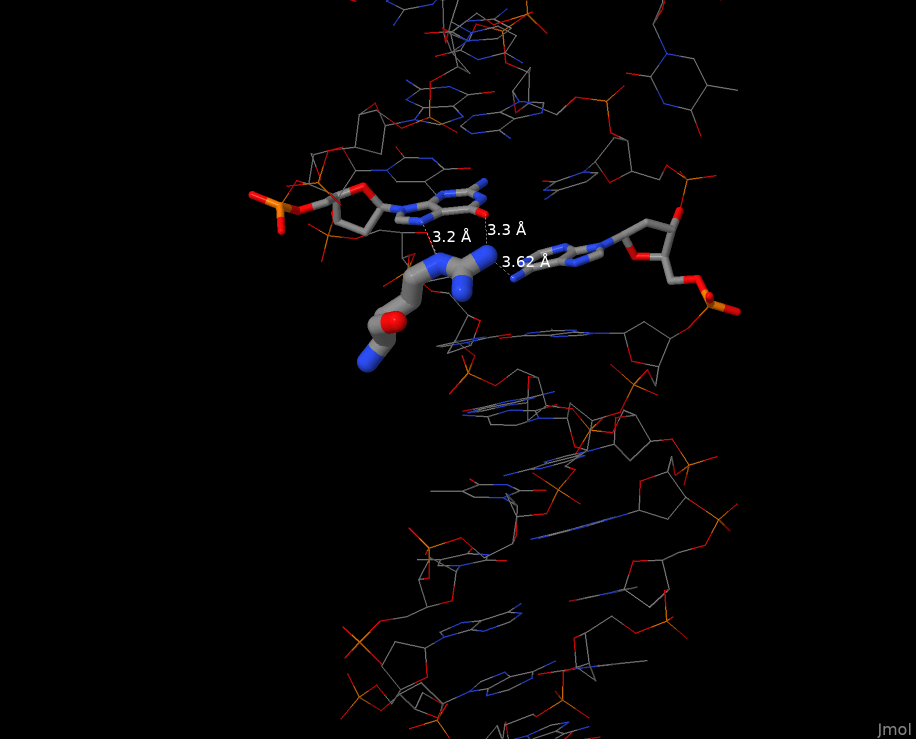
Кроме него, для распознавания последовательности, вероятно, очень важен остаток аргинина 214 цепи
A (см. Рис. 4). Он может образовывать контакты как с гуанином 6 цепи G, так и с аденином 125 цепи H.