Сборка de novo генома бактерии Buchnera aphidicola
Cборка генома Buchnera aphidicola, проект SRR4240387
| fastqc SRR4240387.fastq | Проверка качества чтений |
|---|---|
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 SRR4240387.fastq srr_ad.fastq ILLUMINACLIP:adapters.fasta:2:7:7 | Удаление адаптерных последовательностей |
| java -jar /nfs/srv/databases/ngs/suvorova/trimmomatic/trimmomatic-0.30.jar SE -phred33 srr_ad.fastq srr_trimmed2.fastq LEADING:10 TRAILING:25 SLIDINGWINDOW:5:15 | Очистка чтений |
| fastqc srr_trimmed2.fastq | Проверка качества после очистки чтений |
| velveth . 31 -short -fastq srr_trimmed2.fastq | Подготовка k-меров длины 31 |
| velvetg . -cov_cutoff auto | Сборка последовательностей на основе k-меров |
Анализ качества и очистка чтений.
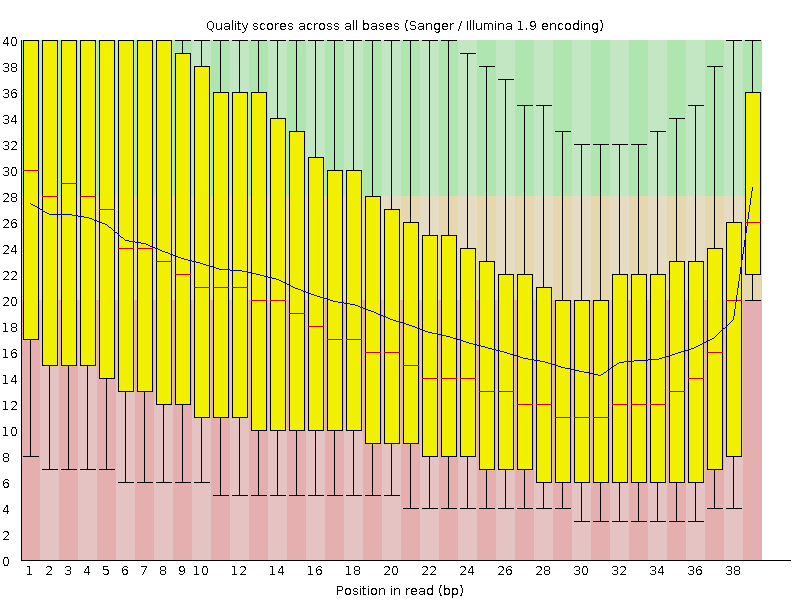
Исходно было получено 15'032'810 чтений. Качество чтений до очистки отображено на Рис. 1.
С помощью Trimmomatic сперва были удалены адаптерные последовательности. 3075 последовательностей
ридов оказались остатками адаптеров.
Затем с конца каждого чтения были отрезаны нуклеотиды с качеством ниже 20,
после чего оставлены только чтения длиной не меньше 32 нуклеотидов. После очистки осталось
3'665'895 ридов. Таким образом, процедуру очистки прошли только 24.4% ридов.
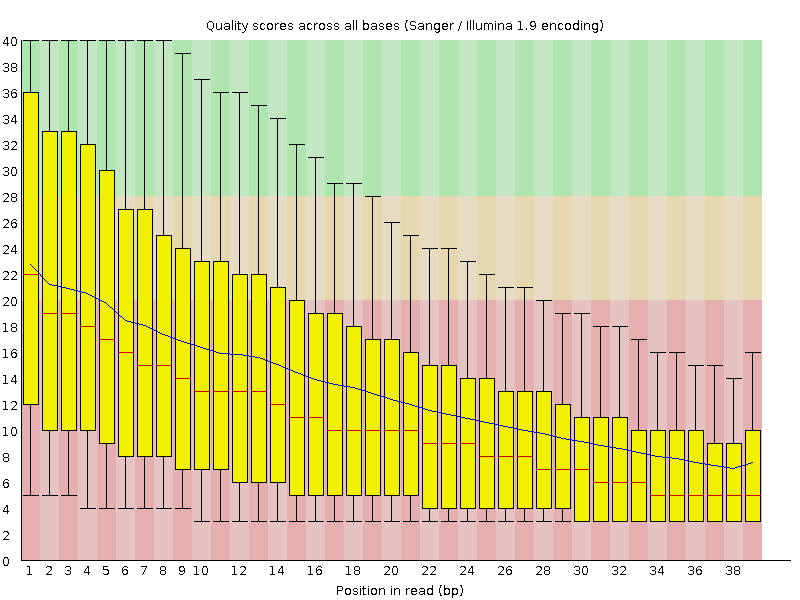
Однако анализ чтений с помощью FastQC после очистки показал, что их качество
все еще очень низкое (см. Рис. 2).
Поэтому пришлось ужесточить параметры trimmomatic: LEADING:10 TRAILING:25 SLIDINGWINDOW:5:15 MINLEN:32.
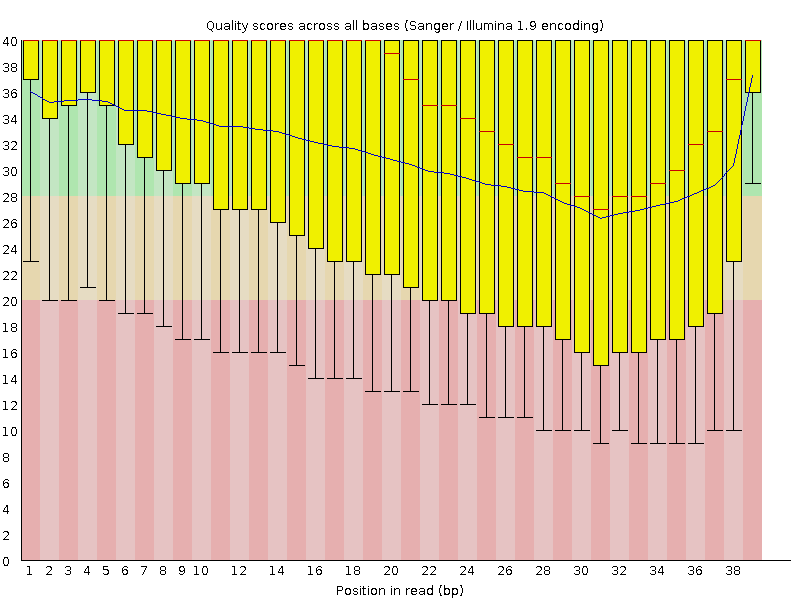
Теперь очистку прошли 552'581 (3.68%) ридов. Размер файла fastq до очистки равен 1.6 GB, после - 57 MB.
Качество чтений после очистки отображено на Рис. 2.
Параметр "Per base sequence quality" пришел в порядок, однако множество параметров все еще оставляют желать лучшего. Например,
перепредставлена последовательность "AAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAAA", составляя 8.2% от
всех последовательностей. Тем не менее, дальнейшее ужесточение параметров FastQC (LEADING:10 TRAILING:25
SLIDINGWINDOW:5:15 MINLEN:32) только увеличивает этот процент, поэтому дело
в самих данных, а не в уровне шума. В связи с этим, дальнейшая работа велась с файлом, полученным с параметрами
trimmomatic LEADING:10 TRAILING:25 SLIDINGWINDOW:5:15 MINLEN:32.
Сборка и анализ.
N50=98. Данные по трем самым длинным контигам доступны в Таблице 2.
Кроме того, обнаружены 3 контига с аномально высоким покрытием, их длины и покрытие доступны в Таблице 3.
В связи с тем, что их длина меньше 31, velvetg не включила их в итоговый fasta-файл с контигами.
Если отбросить эти 3 контига, среднее покрытие равно 5.2. Контигов с аномально низким покрытием не обнаружено (есть
множество контигов с покрытием немного большим 3).
| ID | Длина | Покрытие | |
|---|---|---|---|
| Контиг 1 | 26 | 1198 | 36.73 |
| Контиг 2 | 194 | 934 | 47.11 |
| Контиг 3 | 22 | 883 | 41.08 |
| ID | Длина | Покрытие | |
|---|---|---|---|
| Контиг 1 | 1301 | 1 | 434583 |
| Контиг 2 | 1304 | 1 | 5206 |
| Контиг 3 | 1302 | 11 | 441.5 |
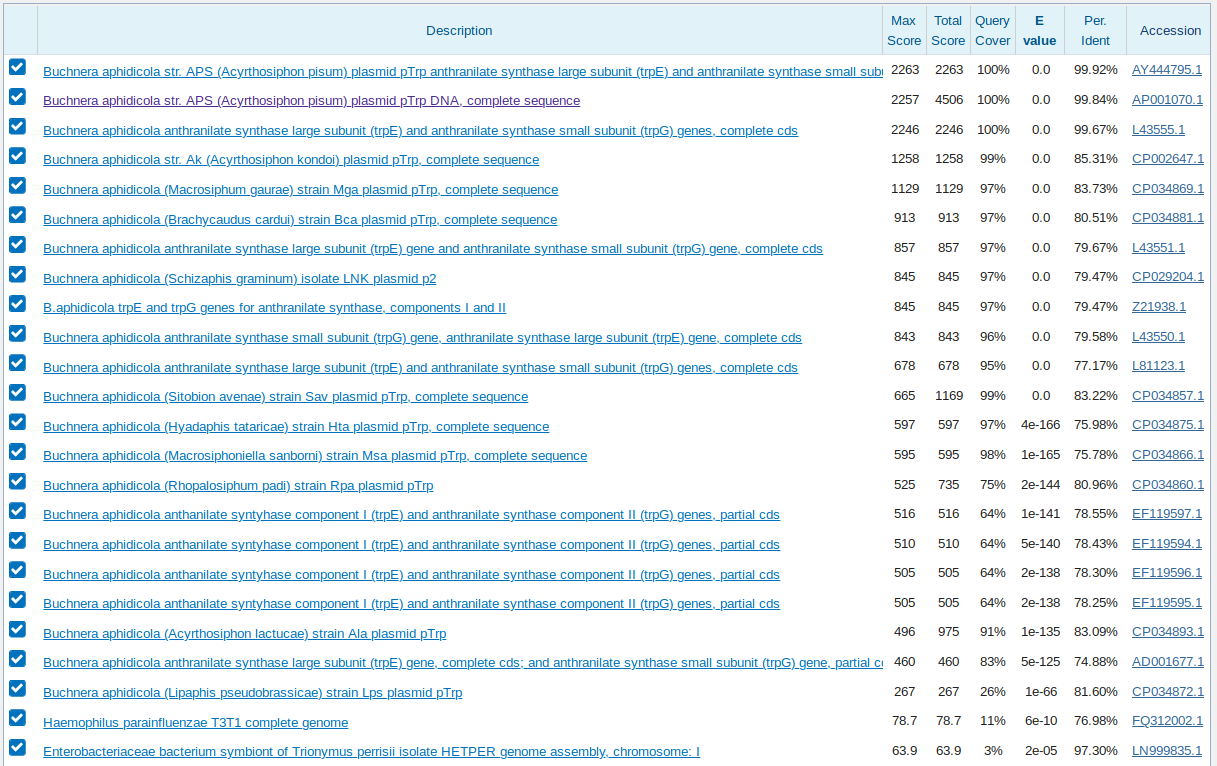
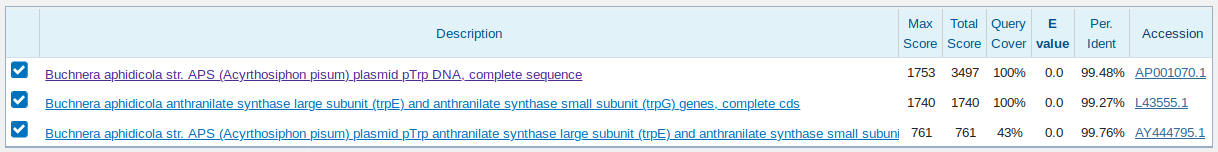
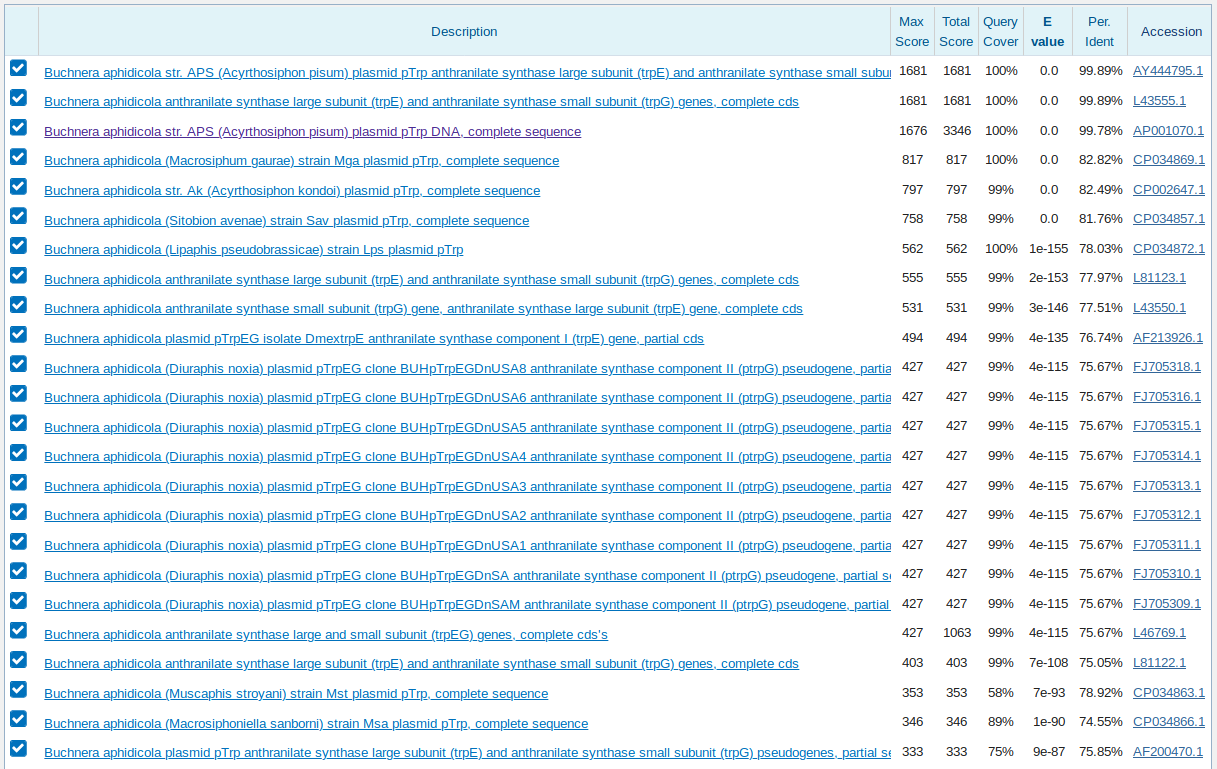
Ни для одного из трех самых длинных контигов megablast не выдал никакого выравнивания с
хромосомой Buchnera aphidicola (GenBank/EMBL AC — CP009253).
Однако megablast по базе Nucleotide collection (nr/nt) для всех трех контигов построил
выравнивание с плазмидой pTrp данной бактерии. Этот любопытный результат говорит о том,
что в данном эксперименте больше всего амплифицировалась эта плазмида, а не какой-либо
участок бактериальной хромосомы.
А именно, все три контига ложатся на разные участки последовательности генов большой (trpE) и малой (trpG) субъединиц антранилат-синтетазы.