KEGG database
KEGG - Ascorbate and aldarate metabolism
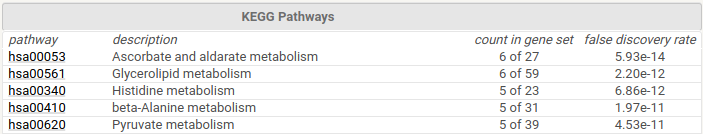
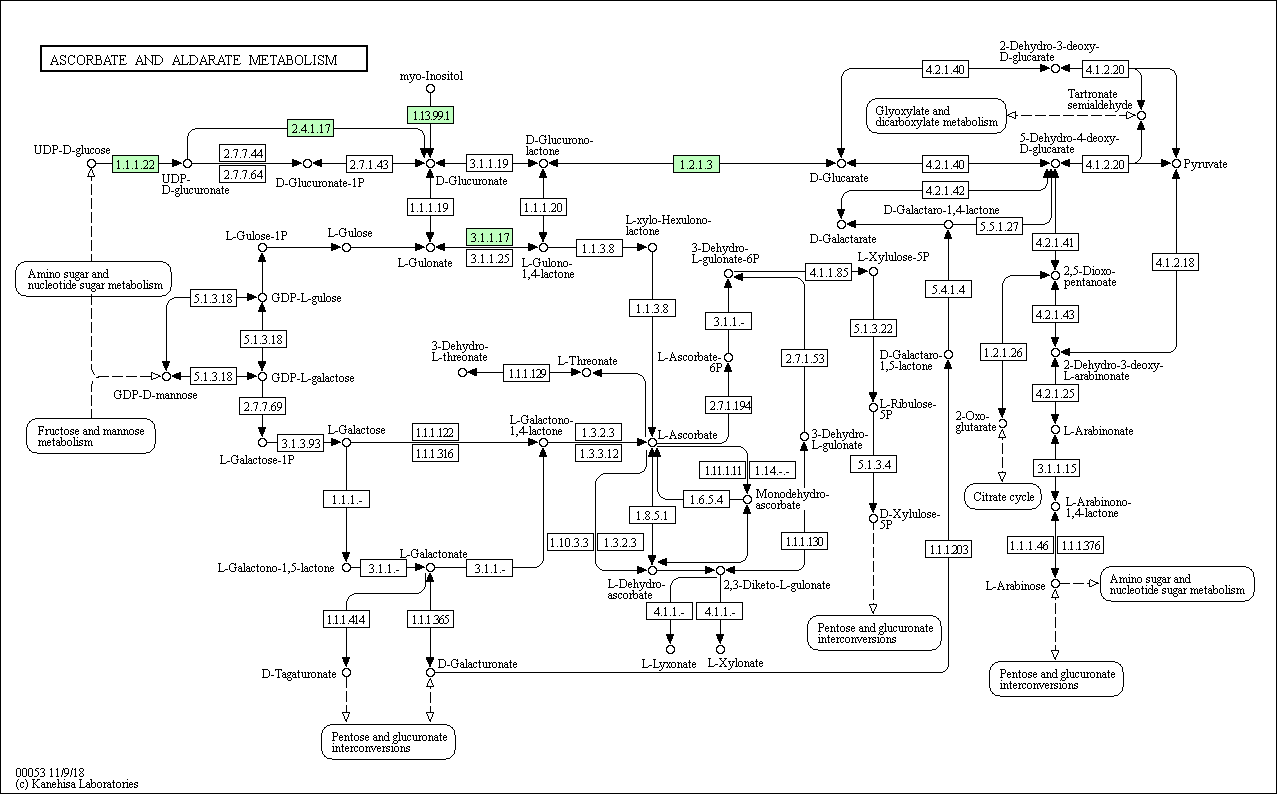
1. Общий вид моего референсного пути map00053 приведен на Рис. 1.
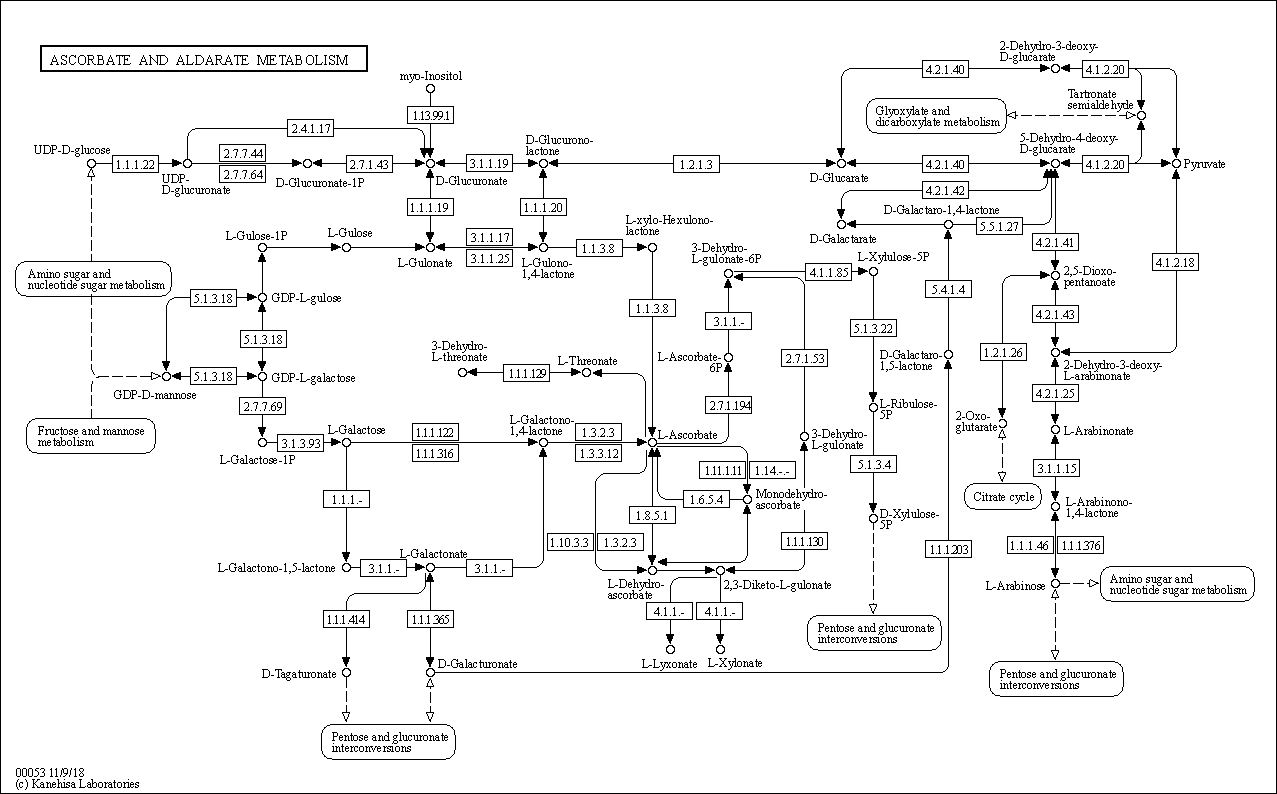
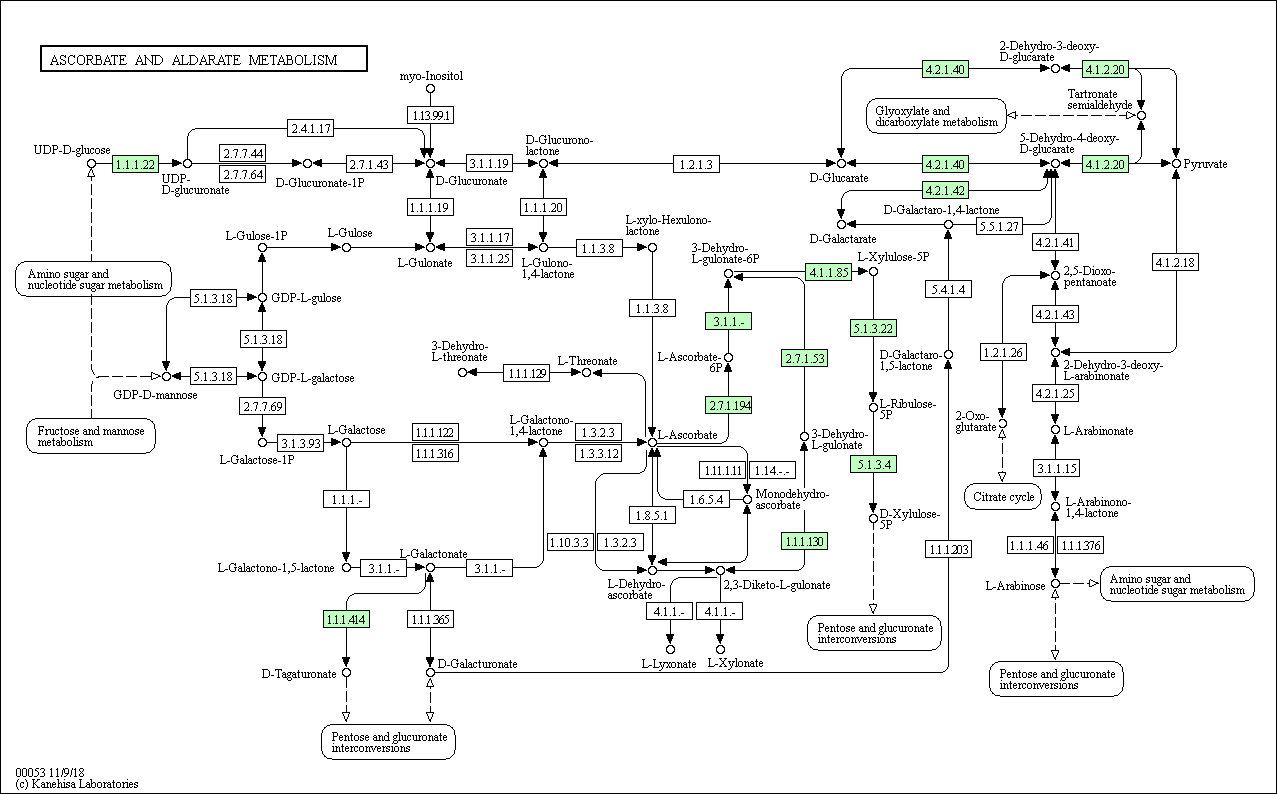
2. Общий вид пути с отмеченной цветом реакцией R01108 приведен на Рис. 2.
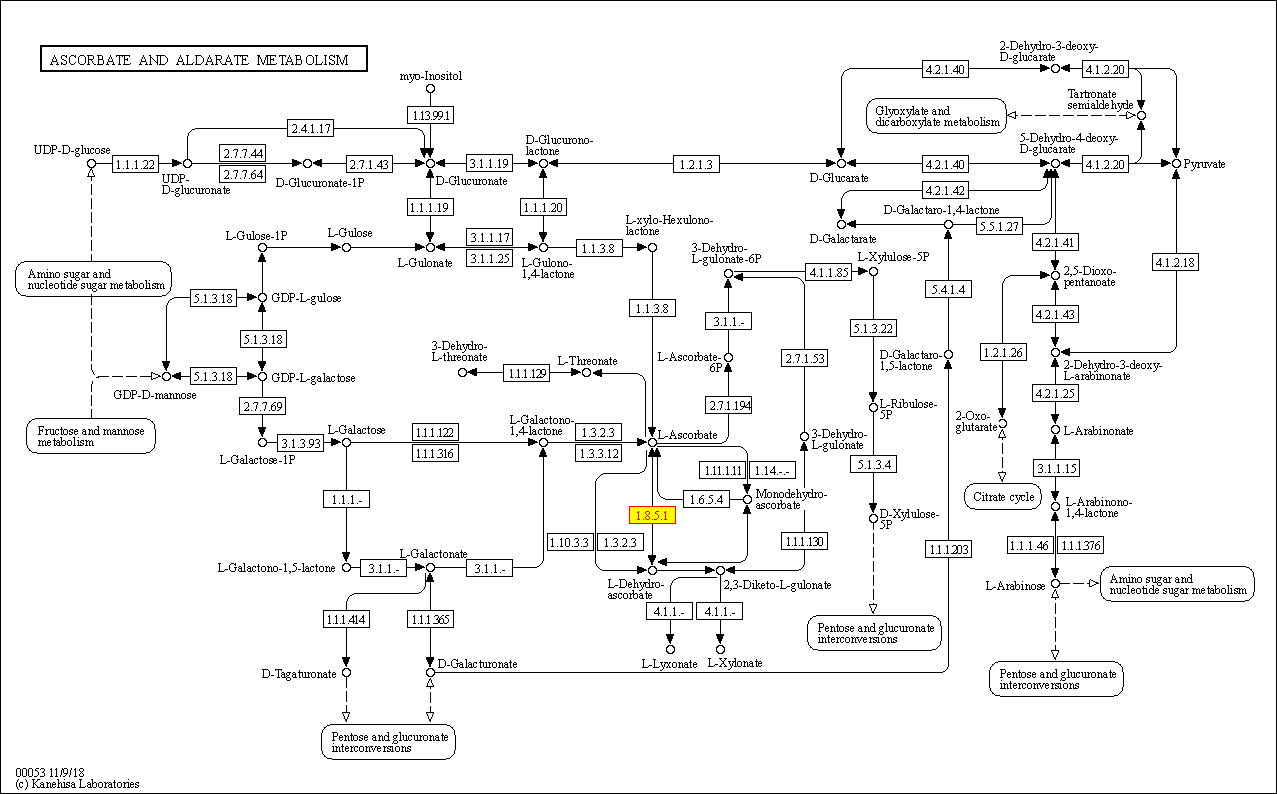
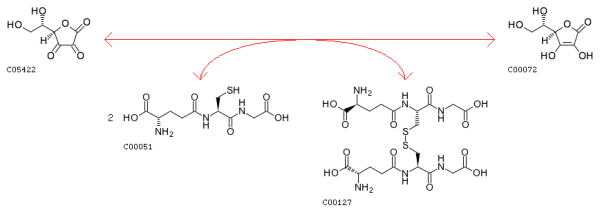
3. Реакция R01108 - это реакция восстановления дегидроаскорбиновой кислоты до аскорбиновой или, наоборот, окисления аскорбиновой кислоты до дегидроаскорбиновой. Протоны для восстановления отдают глутатионы, которые сшиваются дисульфидной связью. Формула реакции приведена на Рис. 3.
4. Реакцию катализирует глутатиондегидрогеназа (EC:1.8.5.1).
5. Разберем код фермента. Фермент принадлежит классу 1 - оксидоредуктазы, то есть осуществляет окислительно-восстановительную реакцию. Подкласс 1.8 - "acting on a sulfur group of donors", то есть донором протонов является тиольная группа. Под-подкласс 1.8.5 - "with a quinone or similar compound as acceptor", то есть акцептором является похожее на хинон соединение. И, наконец, 1.8.5.1 - это код данного фермента, глутатиондегидрогеназы.
6. Картинки пути для Homo sapiens и Escherichia coli K-12 MG1655 доступны на Рис. 4 и 5.
7. Для человека в KEGG имеется 337 метаболических путей, 19855 белок-кодирующих генов и 2641 РНК-кодирующих генов.
8. С помощью KEGG API я установил, что число общих для Homo sapiens и Escherichia coli K-12 MG1655 метаболических путей равно 77.
Сначала я получил списки путей для обоих организмов следующим образом: http://rest.kegg.jp/list/pathway/eco и http://rest.kegg.jp/list/pathway/hsa. Затем я скачал полученные списки, распарсил их и получил пересечение с помощью следующего скрипта на R:
library(stringr)
hum <- read.table("hsa_path.tsv",sep="\t")
eco <- read.table("eco_path.tsv",sep="\t")
hum_path <- str_split(hum[,1],":",simplify = T)
hum_path <- str_sub(hum_path[,2],4)
eco_path <- str_split(eco[,1],":",simplify = T)
eco_path <- str_sub(eco_path[,2],4)
length(intersect(hum_path,eco_path))
9. С помощью STRING для списка генов, использованного мной в предыдущем практикуме, было получено обогащение KEGG путями. Топ находок приведен на Рис. 6.