Сигналы и мотивы 2
PSI-BLAST
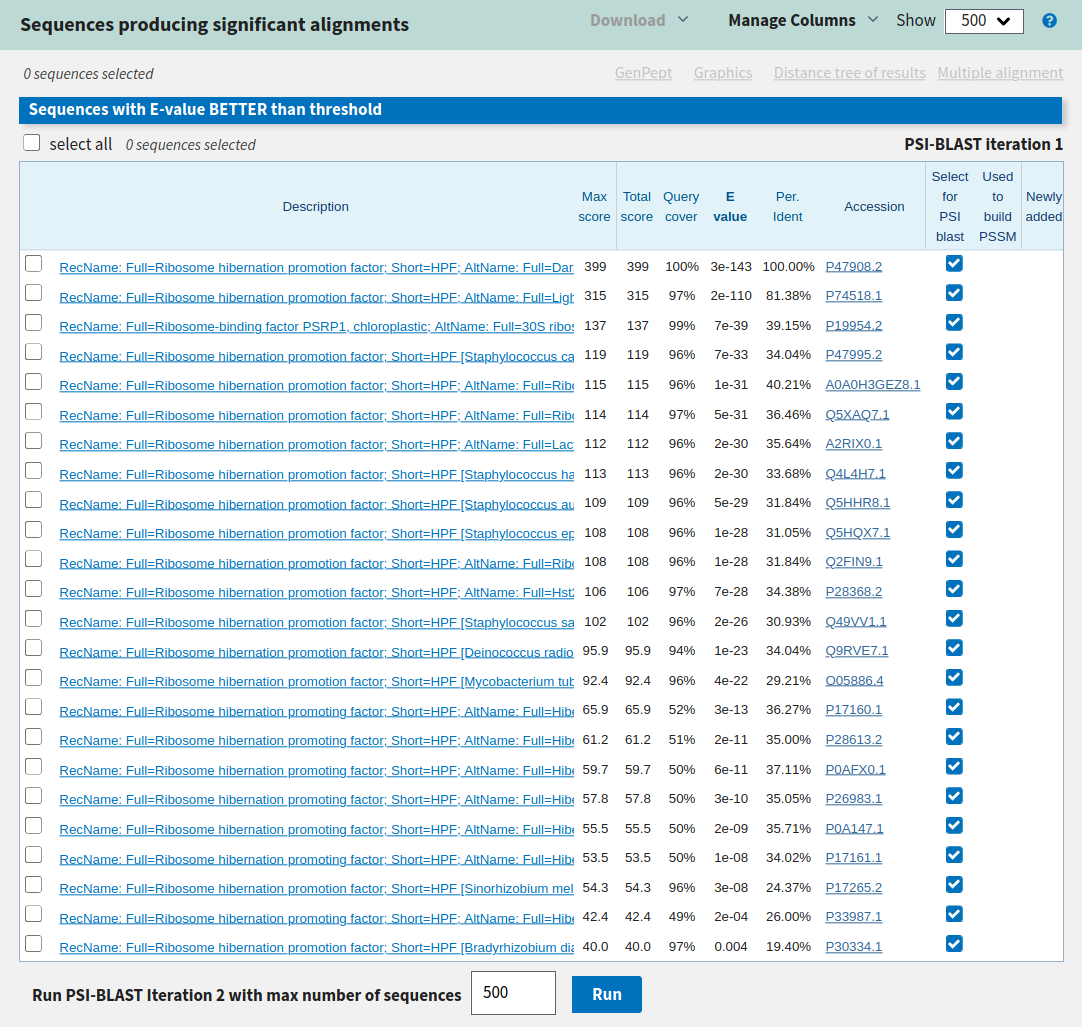
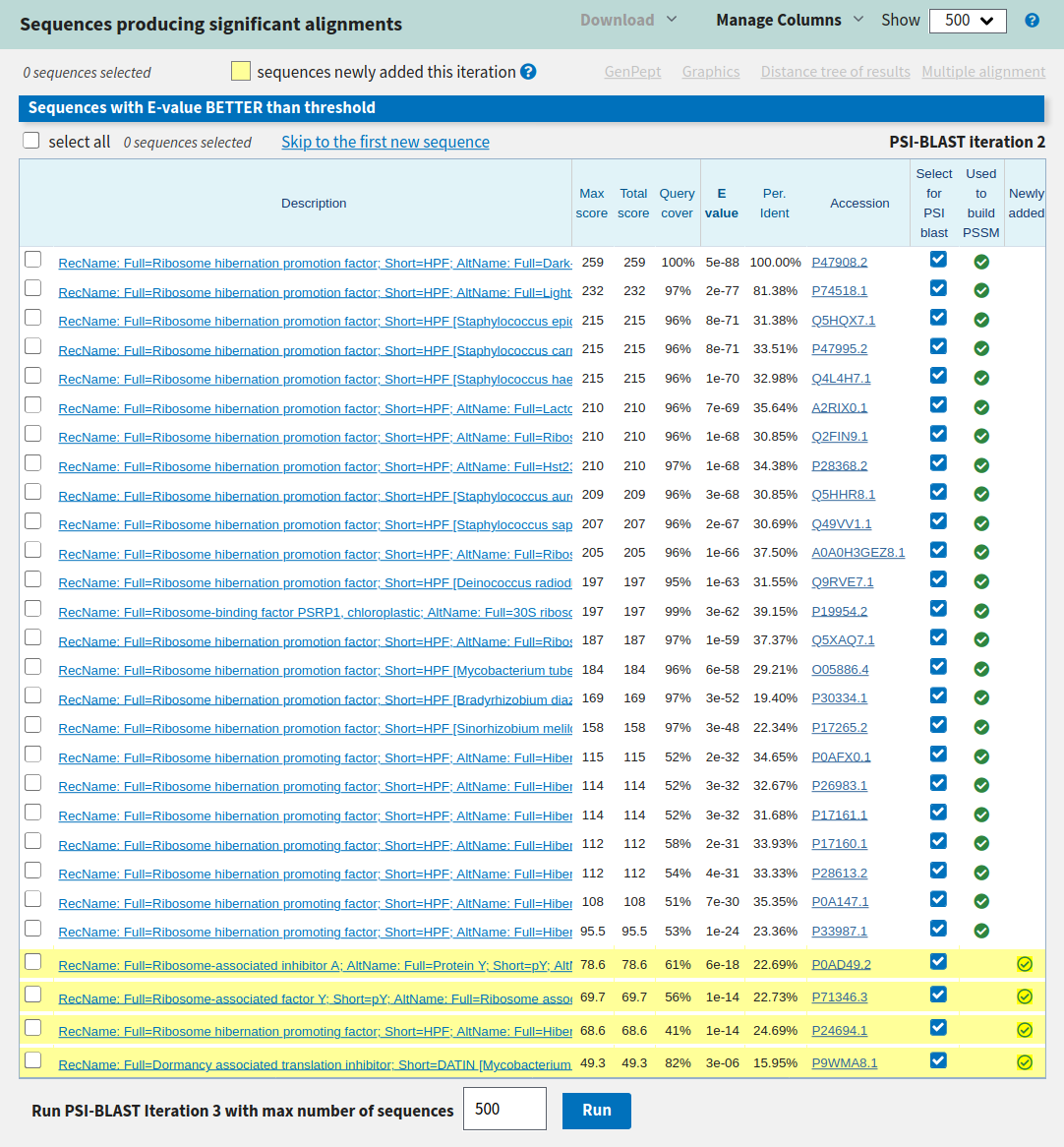
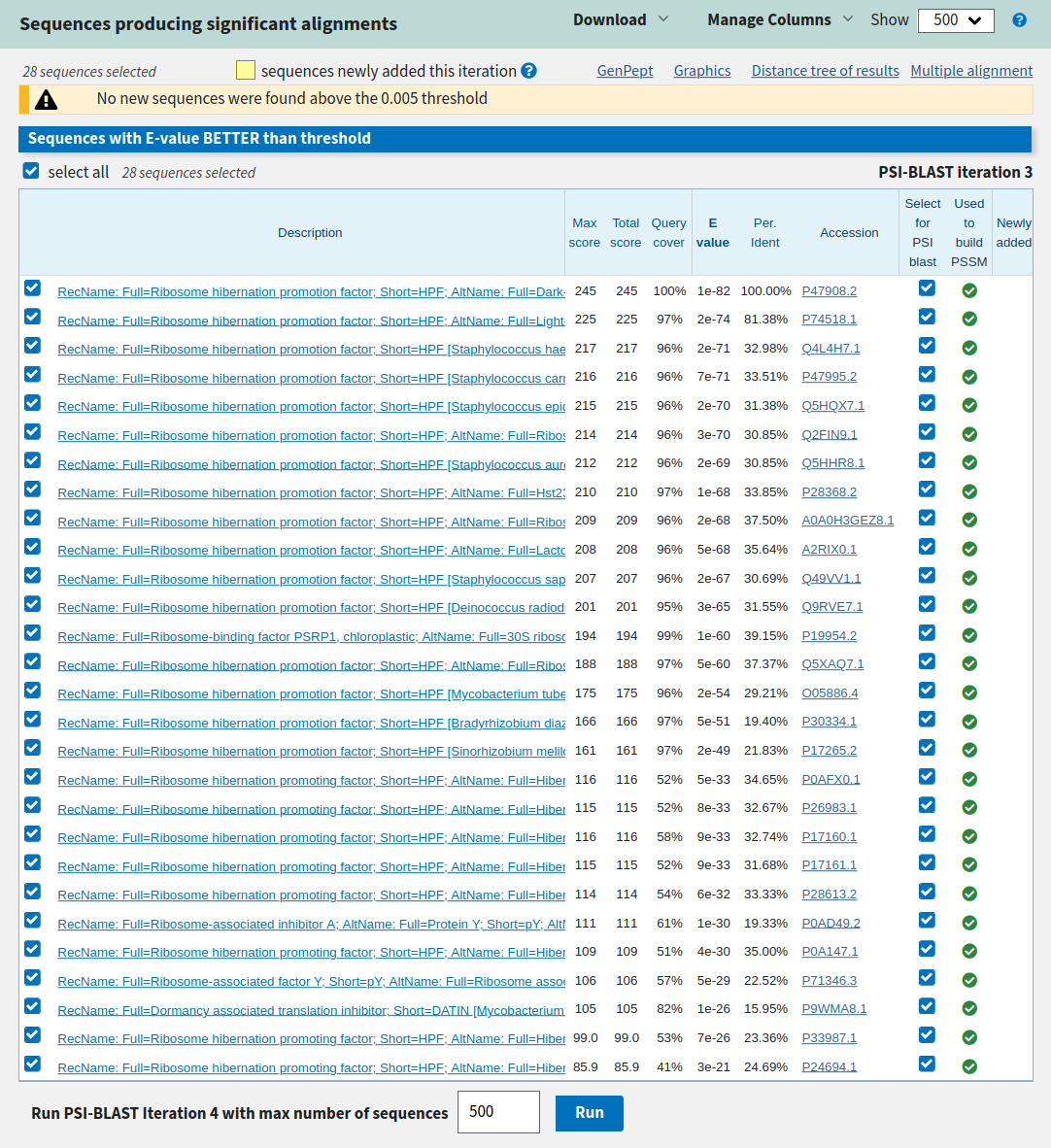
Я выбрал белок P47908. Это ribosome hibernation promotion factor из Synechococcus sp. После третьей итерации не добавилось новых находок, семейство белков хорошее.
Это мое задание не было принято с первой попытки со следующим комментарием: "После 1й итерации указано "P30334.1 0.004" и "P47908.2 3.00E-143". Так быть не может!"
Однако это именно то, что выдает PSI-BLAST. Скрины выдачи привожу ниже (Рис. 1-3).
Эндонуклеазы рестрикции
Я получил список сайтов с помощью следующего скрипта на R:
endonucl <- read.table("TypeII_REs.tsv",sep="\t")
endonucl <- endonucl[which(endonucl$V5!="-"),]
endonucl <- endonucl[which(!duplicated(endonucl$V5)),]
endonucl <- endonucl[nchar(as.vector(endonucl$V5))>2,]
writeLines(as.vector(endonucl$V5),con=file("re_sites.txt"),sep=" ")
После исключения повторяющихся сайтов, а также сайтов в 1-2 нуклеотида (так как поиск таких сайтов в геноме будет бессмысленным) осталось 207 различных сайтов, которые я через пробел записал в файл.
После этого на полученном списке сайтов рестрикции я запустил программу CBcalc (см. команду ниже), которая оценивает представленность сайтов в геноме с помощью метода, предложенного Chris Bruge и Samuel Karlin. Метод заключается сперва в оценке ожидаемого числа находок данного сайта, а затем подсчета реального числа, после чего считается отношение Observed/Expected. Чем это значение больше, тем более представленным является сайт. Сайты я искал в геноме бактерии Cellvibrio japonicus.
cbcalc -s re_sites.txt -o cbcalc.tsv -K cellvibrio_japonicus.fna
Результат работы программы доступен в таблице (манипуляции с ней и отбор сайтов выполнялись в LibreOffice Calc). Для нас наибольший интерес представляют сайты с наименьшей представленностью, так как против этих сайтов идет наиболее сильный отбор, потому что ДНК в них может подвергаться гидролизу эндонуклеазами (например, при ошибках в метилировании этих сайтов).
Сначала решил установить порог на O/E в 0.8, как было предложено в задании: сразу многие из найденных сайтов имели O/E немного выше, так что такое пороговое значение действительно выглядело разумным.
Однако полученный с таким порогом список эндонуклеаз включал 389 экспериментально проверенных белков из-за того, что сайт "TCTAGA", имеющий O/E ratio 0.754, слишком "популярный". При снижении порога до 0.7 число белков снизилось до 22, поэтому я решил остановиться на данном пороговом значении.
Скрипт на R, с помощью которого были получены экспериментально подтвержденные эндонуклеазы:
sites <- c('CTCGAG','CTAG','CGTACG','GGATCC','GAGCTC')
endonucl <- read.table("TypeII_REs.tsv",sep="\t")
endonucl <- endonucl[which(endonucl$V5 %in% sites),]
endonucl <- endonucl[which(endonucl$V11 == 'no'),]
names_endonucl <- as.vector(endonucl$V1)
writeLines(names_endonucl,con=file("found_endonucl.txt"),sep="\n")
Итак, список полученных эндонуклеаз рестрикции:
AbrI BamFI BamHI BfaIA BfaIB Bsp98I BstVI BsuMIA BsuMIB BsuMIC CchI DdsI MjaI MthZI PaeR7I R1.BsuMI R2.BsuMI R3.BsuMI SacI Sgr13350I TliI XhoI
Информацию о них можно найти в базе REBASE, из которой и был получен изначальный список эндонуклеаз с сайтами. Например, эндонуклеаза AbrI была выделена из почвенной бактерии Azospirillum brasilense.