Паралоги и ортологи
Составление списка белков
Поиск гомологов CLPX_ECOLI по протеомам выбранных мной бактерий с помощью blastp выдал 31 находку. Список находок:
Sequences producing significant alignments: Score (Bits) E-Value sp|Q2RL30|CLPX_MOOTA ATP-dependent Clp protease ATP-binding sub... 533 0.0 sp|A5I6W0|CLPX_CLOBH ATP-dependent Clp protease ATP-binding sub... 530 0.0 sp|Q81LB9|CLPX_BACAN ATP-dependent Clp protease ATP-binding sub... 513 0.0 sp|Q5HNM9|CLPX_STAEQ ATP-dependent Clp protease ATP-binding sub... 499 6e-177 sp|Q833M7|CLPX_ENTFA ATP-dependent Clp protease ATP-binding sub... 498 3e-176 sp|Q9CGE6|CLPX_LACLA ATP-dependent Clp protease ATP-binding sub... 490 1e-173 tr|Q5FKR6|Q5FKR6_LACAC ATP-dependent Clp protease ATP-binding s... 484 4e-171 sp|Q834K4|HSLU_ENTFA ATP-dependent protease ATPase subunit HslU... 103 3e-24 sp|Q5FKD8|HSLU_LACAC ATP-dependent protease ATPase subunit HslU... 101 1e-23 tr|Q2RJP5|Q2RJP5_MOOTA ATP-dependent protease ATPase subunit Hs... 100 4e-23 sp|Q81WK6|HSLU_BACAN ATP-dependent protease ATPase subunit HslU... 98.2 2e-22 sp|Q5HPT8|HSLU_STAEQ ATP-dependent protease ATPase subunit HslU... 93.6 7e-21 tr|A5I766|A5I766_CLOBH ATP-dependent zinc metalloprotease FtsH ... 52.0 2e-07 tr|Q2RLR4|Q2RLR4_MOOTA AAA domain-containing protein OS=Moorell... 50.1 6e-07 tr|Q5FHW6|Q5FHW6_LACAC UVR domain-containing protein OS=Lactoba... 48.9 2e-06 tr|Q2RLP6|Q2RLP6_MOOTA AAA domain-containing protein OS=Moorell... 48.5 2e-06 tr|Q5FM98|Q5FM98_LACAC UVR domain-containing protein OS=Lactoba... 47.8 6e-06 tr|A5I7Q0|A5I7Q0_CLOBH ATP-dependent zinc metalloprotease FtsH ... 46.2 1e-05 tr|Q5FLA7|Q5FLA7_LACAC UVR domain-containing protein OS=Lactoba... 46.2 1e-05 tr|A0A347ZXP1|A0A347ZXP1_BACAN ATP-dependent zinc metalloprotea... 43.1 1e-04 tr|Q2RM95|Q2RM95_MOOTA ATP-dependent zinc metalloprotease FtsH ... 43.1 1e-04 tr|A0A1Q4LW06|A0A1Q4LW06_BACAN AAA domain-containing protein OS... 42.4 2e-04 sp|P46469|FTSH_LACLA ATP-dependent zinc metalloprotease FtsH OS... 42.7 2e-04 tr|Q5FMA3|Q5FMA3_LACAC ATP-dependent zinc metalloprotease FtsH ... 42.4 2e-04 sp|Q5HPD3|Y979_STAEQ Uncharacterized protein SERP0979 OS=Staphy... 41.6 3e-04 tr|A5I501|A5I501_CLOBH AAA domain-containing protein OS=Clostri... 41.6 3e-04 tr|Q839B1|Q839B1_ENTFA ATP-dependent zinc metalloprotease FtsH ... 41.6 4e-04 tr|A5HYU4|A5HYU4_CLOBH ATP-dependent zinc metalloprotease FtsH ... 41.2 4e-04 tr|Q5HRP3|Q5HRP3_STAEQ ATP-dependent zinc metalloprotease FtsH ... 41.2 6e-04 sp|Q9CI09|CLPE_LACLA ATP-dependent Clp protease ATP-binding sub... 40.8 6e-04 tr|Q2RJJ8|Q2RJJ8_MOOTA AAA domain-containing protein OS=Moorell... 40.4 9e-04
Реконструкция и визуализация
Множественное выравнивание найденных белков было получено с помощью алгоритма MUSCLE, реализованного в UGENE.
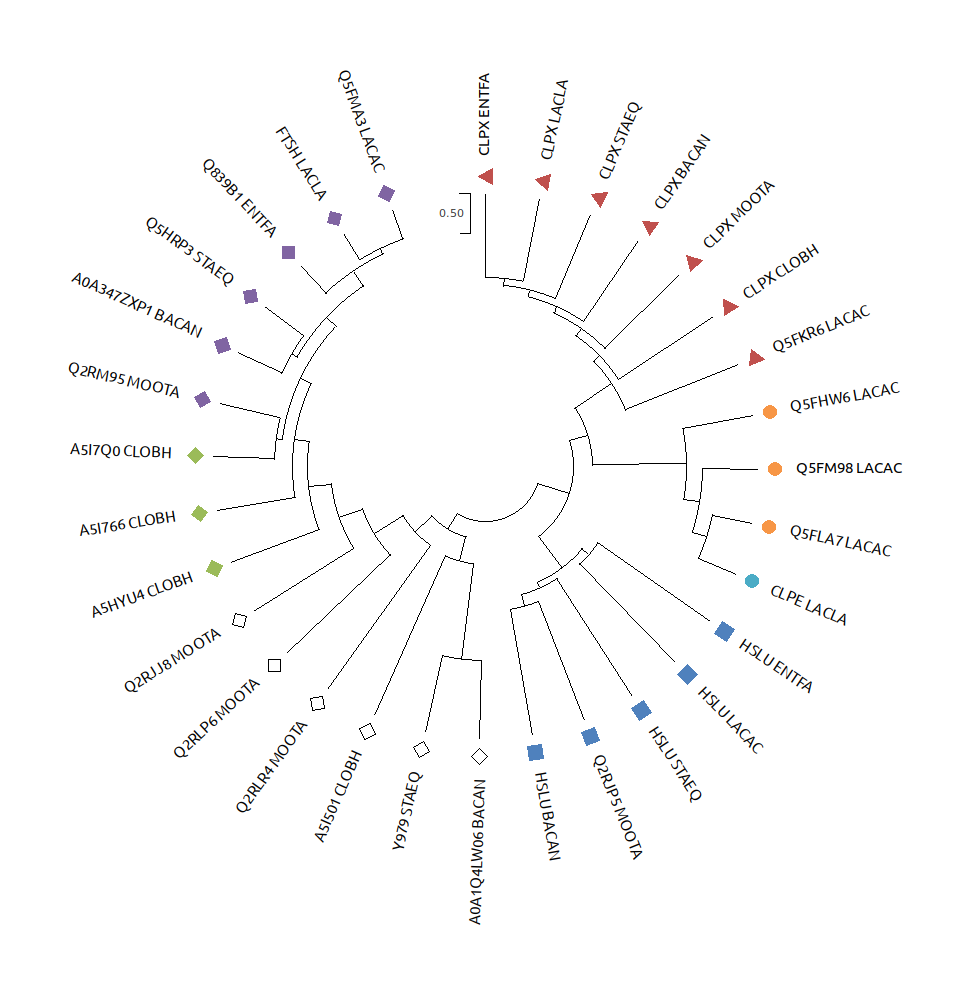
Дерево белков реконструировано алгоритмом Maximum-Likelihood в программе MEGA. Визуализация получена с помощью программ
MEGA и FigTree. Результат реконструкции доступен на Рис. 1.
Примеры пар ортологов:
1. Q5FHW6_LACAC и Q5FM98_LACAC.
2. A5I7Q0_CLOBH и A5I766_CLOBH.
3. Q2RJJ8_MOOTA и Q2RLP6_MOOTA.
Примеры пар паралогов:
1. CLPX_ENTFA и CLPX_LACLA.
2. CLPE_LACLA и Q5FLA7_LACAC.
3. HSLU_STAEQ и HSLU_BACAN.
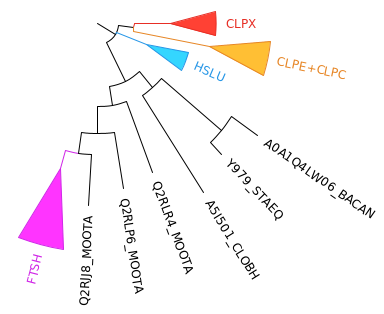
1. "CLPX" - ATP-dependent Clp protease ATP-binding subunit ClpX, присутствует во всех 7 видах.
2. "CLPE + CLPC" - судя по всему, белки других субъединиц ATP-dependent Clp protease. Группа из четырех белков, три из которых - паралоги из одного организма, Lactobacillus acidophilus.
3. "HSLU" - ATP-dependent protease ATPase subunit HslU. Имеется у всех видов, кроме Lactococcus lactis subsp. lactis и Clostridium botulinum.
4. "FTSH" - ATP-dependent zinc metalloprotease FtsH. Присутствуют во всех 7 видах, причем у Clostridium botulinum имеются 3 паралога. Единственная группа, внутри которой дерево ортологов полностью (за исключением расположения двух паралогов из CLOBH) совпадает с деревом самих видов.
5. Остальные белки, не объединенные в группы, - по большей части, вероятно, нефункциональные паралоги белков из группы FTSH.