Сигналы и мотивы
Позиционная весовая матрица
Поиск сайтов регуляции разрывной транскрипции sgmRNA коронавируса нетопырей HKU5
Я выбрал для анализа Pipistrellus bat coronavirus HKU5 (NC_009020.1). В его геноме 9 генов: один ген полипротеина и 8 поздних генов. Большинство из них кодируются
с перекрыванием. Для поиска мотивов я выбрал upstream область перед геном полипротеина длиной 260 нуклеотидов и фрагменты по 100 нуклеотидов
перед каждым из поздних генов.
Входные параметры программы MEME и ее выдача доступны по ссылке. Изначально были и другие находки,
но с E-value>1.
Был найден один мотив с E-value=1.4e-06. Его LOGO и расположение доступны в выдаче MEME. Стоит сказать, что последовательность
найденного мотива сильно отличается от приведенной CS в предложенной статье: Zúñiga S, Sola I, Alonso S, Enjuanes L. Sequence motifs involved in the
regulation of discontinuous coronavirus subgenomic RNA synthesis. J Virol. 2004;78(2):980–994. doi:10.1128/jvi.78.2.980-994.2004.
Тем не менее, его расположение и E-value находки позволяют предположить, что это именно сигнал TRS-L и TRS-B.

Подтверждение мотива с помощью FIMO
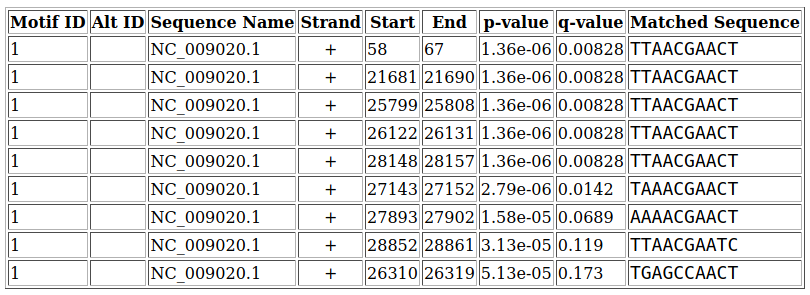
Выдача FIMO доступна по ссылке, находки - на Рис. 2. Было найдено 9 последовательностей с низким E-value, что как раз соответствует девяти генам вируса. Таким образом, FIMO обнаружил последовательность мотива перед геном NS3a, которую изначально я не нашел с помощью MEME из-за того, что она отстоит от начала гена немного более, чем на 100 нуклеотидов.
Мотив, как я уже писал, не очень похож на приведенный в статье 2004 года. Он длины 10, а не 6, и последовательность тоже сильно отличается. Тем не менее, сигнал есть перед каждым геном (и полипротеина, и поздними), а E-value находок достаточно низкое.
Последовательности Козак генов коронавируса HKU5
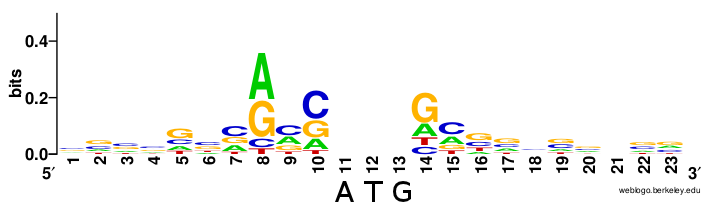
Я искал последовательности с помощью MEME, ограничив поиск 20 нуклеотидами вокруг начала каждого гена. Был найден один мотив с низким E-value (5.2e-02). Поскольку данный мотив был также обнаружен вокруг начала гена полипротеина (p=1.3e-03), я решил дополнительно не ограничивать поиск поздними генами. Результат доступен по ссылке, а сам мотив - на Рис. 3. Чтобы получить не только мотив ATG, я задал минимальный размер мотива равным 10. Интересно, что найденный сигнал расположен, за исключением одного нуклеотида, после ATG. При этом две позиции непосредственно после ATG действительно похожи на фрагмент последовательности Козак человека: наиболее часто встречается G перед C (см. Рис. 4). Кроме того, любопытно, что у вируса имеются "свои" сигналы - например, T или C в 10 позиции мотива. Не стоит забывать, что это может быть связано, например, с отличиями последовательности Козак нетопырей от человеческой.
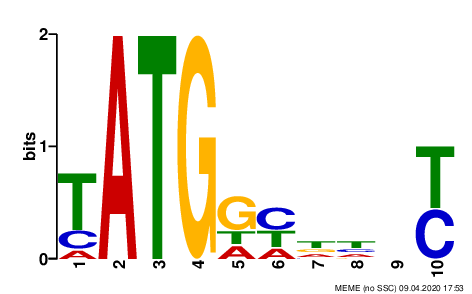
Поиск мотива из Задания 2 в геномах других коронавирусов
Я искал мотив с помощью FIMO в геномах Bat coronavirus HKU5-5 (EF065512.1, другой штамм HKU-5) и Bat coronavirus HKU4-1 (NC_009019.1, родственный вид из подрода Merbecovirus.
Результаты для HKU5-5 и HKU-4.
В геноме HKU5-5 найдено восемь последовательностей, в том числе перед геном полипротеина (всего генов 9). В геноме HKU4-1 найдено 11 последовательностей, и маловероятно, что все они действительно являются сайтами регуляции разрывной транскрипции, однако несколько из них имеют достаточно высокий FDR, так что при более детальном изучении можно выяснить, какие последовательности действительно могут быть функциональными.
Полученные результаты показывают, что найденный мной мотив вероятно является консервативным, что еще раз подтверждает, что это неплохой кандидат на роль сайта регуляции синтеза sgRNA.