Домены и профили
Список белков с выбранными доменами
Информация о выбранных мной доменах доступна в Таблице 1.
Стоит заметить, что про домен TetR_N достаточно мало известно.
В базе данных InterPro он описан как ДНК-связывающий домен со структурой спираль-поворот-спираль, который обнаруживается во
многих регуляторах транскрипции бактерий и архей, таких как TetR, репрессор устойчивости к тетрациклину. Данный домен
обнаруживается в подсемействе белков, включающем в себя регуляторы транскрипции TetR, TetC, AcrR, BetI, Bm3R1, EnvR, QacR,
MtrR, TcmR, Ttk, YbiH, и YhgD. Многие из этих белков функционируют как репрессоры, контролирующие уровень чувствительности
к гидрофобным антибиотикам и детергентам.
В базе данных Pfam название этого домена дано по названию семейства регуляторов TetR. Другой выбранный мной домен, TetR_C_37,
представляет собой C-концевой домен некоторых белков семейства TetR, участвующий в димеризации.
Соответственно, выбранная доменная архитектура - это домен TetR_C_37, следующий за доменом TetR_N. По данным Pfam существует
83 последовательности с данной архитектурой.
Список белков.
| ID | TetR_N | TetR_C_37 |
|---|---|---|
| AC | PF00440 | PF18665 |
| Название | Bacterial regulatory proteins, tetR family (см. текст отчета) | Tetracyclin repressor-like, C-terminal domain |
| Число последовательностей среди бактерий в Uniprot | 636,643 | 275 |
HMM профиль белков с выбранной доменной архитектурой
На Рис. 1 изображено распределение длин белков с выбранной доменной архитектурой. Распределение бимодальное (кроме основного пика на 195-210 а.о., есть второй пик на 185-190 а.о.). Это, возможно, отражает наличие двух подгрупп среди данных белков.
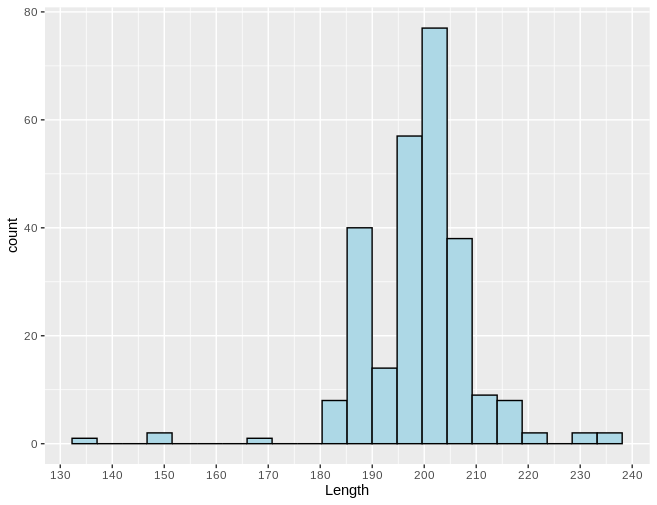
Из данного 261 белка были отобраны 226 белков длиной от 185 до 210 а.о. Ниже приведены таксономические группы, белки бактерий из которых попали в отобранный список. Все выбранные белки обладают только двумя доменами Pfam (собственно, с интересующей нас доменной архитектурой).
Отделы: Firmicutes Bacteroidetes Proteobacteria Spirochaetes Actinobacteria Семейства: Staphylococcaceae Streptococcaceae Chitinophagaceae Cyclobacteriaceae Clostridiaceae Sphingobacteriaceae Cytophagaceae Flammeovirgaceae Prolixibacteraceae Spirochaetaceae Flavobacteriaceae Saprospiraceae Desulfobacteraceae Desulfobulbaceae Rhodobacteraceae Catalimonadaceae
Множественное выравнивание с помощью MUSCLE показало, что данные белки делятся на две группы по размеру (см. Рис. 2). Это следовало ожидать из гистограммы длин. Для дальнейшего анализа я решил оставить только более длинную группу, которая ниже на Рис. 2, так как таких белков сильно больше. Осталось 179 белков, для которых я заново построил выравнивание и выполнял дальнейшие действия.

Затем я убрал малоконсервативные участки с концов выравнивания и с помощью команд hmm2build и hmm2calibrate построил HMM профиль полученного выравнивания. Затем я скачал из Uniprot все последовательности, содержащие домен PF00440.
К сожалению, таких последовательностей очень много, 636,6 тысяч. Однако второй домен из выбранной архитектуры - это один из вариантов C-концевого домена белков семейства TetR, для которых характерен первый домен. Иными словами, во всех белках, имеющих домен PF18665, обязательно имеется домен PF00440. Поэтому для проверки HMM профиля нельзя использовать белки с PF18665.
С помощь программы hmm2search с порогом на E-value 0.1 я провел поиск по всем белкам с доменом PF00440.
После этого я вручную выбрал из полученного в результате работы hmm2search файла строки, содержащие информацию о Score и E-value последовательностей. После этого с помощью скрипта на питоне я распарсил полученный файл, а затем провел анализ.
Дальнейший анализ с комментариями можно посмотреть на html версии моего Jupyter-Notebook или же скачав оригинальный ноутбук.