Реконструкция эволюции доменной архитектуры
Выбор домена
Для выполнения задания был выбрано семейство доменов HTH_Tnp_Tc3_2(PF01498).
Домены данного семейства представляют собой мотив «спираль-поворот-спираль» (Helix-turn-helix), входящий в N-концевой участок транспозаз Tc3.
Транспозаза — это фермент, связывающий одноцепочечную ДНК и встраивающий последнюю в геномную ДНК. Выбранный домен обеспечивает связывание с ДНК.
На рис. 1 представлена структура ДНК-связывающего домена Tc3, связанного с ДНК (PDB ID: 1U78).
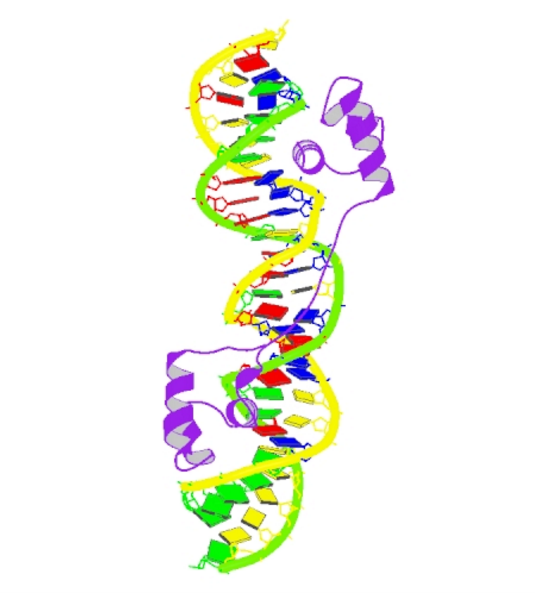 |
| Рис. 1. Комплекс ДНК-связывающего домена Tc3 с ДНК (PDB ID: 1U78) |
Выбор доменных архитектур
Данный домен представлен в 126 доменных архитектурах, большей части которых соответствует 1 последовательность.
Из списка доменных архитектур, представленных на данной странице Pfam были выбраны следующие 2
доменные архитектуры:
Таблица 1.Выбранные доменные архитектуры HTH_Tnp_Tc3_2
| Схема |
Домены |
Обозначение |
Количество последовательностей |
Характеристика |
 |
HTH_Tnp_Tc3_2(PF01498) |
1 |
1138 |
Состоит из одного (исходного) домена |
 |
HTH_Tnp_Tc3_2(PF01498), DDE_3(PF13358) |
2 |
1717 |
Помимо исходного домена содержит домен DDE (эндонуклеаза) |
Выбор таксонов и подтаксонов
На рис.2 представлено распределение последовательностей, входящих в семейтство HTH_Tnp_Tc3_2 по таксонам.
В большинстве своем данный домен встречается у эукариот (3760 последовательности) очень редко - у бактерий (22 последовательности.
Для дальнейшей работы были выбран таксон Eukaryota и два подтаксона - царства Metazoa (2778 последовательностей)
и Fungi (885 последовательностей).
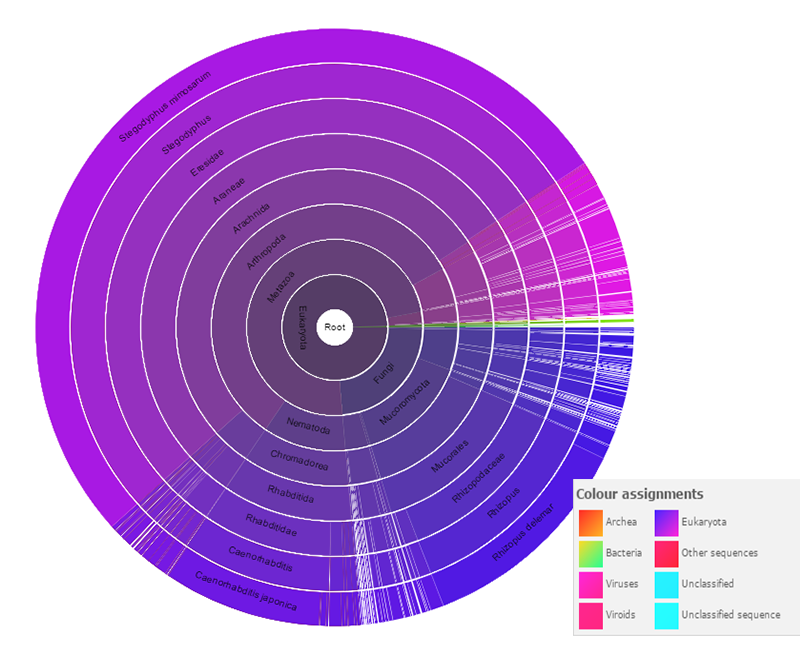 |
| Рис. 2. Распределение последовательностей, входящих в семейтство HTH_Tnp_Tc3_2 по таксонам |
Построение выравнивания последовательностей домена
Ссылка на проект Jalview с выравниванием из Pfam для семейства доменов HTH_Tnp_Tc3_2 + PDB-структура белка TC3A_CAEEL: pr11.jvp
Выравнивание из Pfam для семейства доменов HTH_Tnp_Tc3_2: pr11.fasta
Составление сводной таблицы
С помощью скрипта swisspfam-to-xls.py была получена таблица с информацией об архитектуре всех последовательностей,
содержащих домен PF01498:
python swisspfam-to-xls.py -z /srv/databases/pfam/swisspfam.gz -p PF01498 -o pr11.xls
К этой же таблице были добавлены колонки с указанием доменной архитектуры ("1" или "2", см. табл.1) и таксономической принадлежности. Для этого
с Uniprot были скачаны полные записи всех последовательностей из .xls файла с помощью "Retrieve" в тексовом формате. Полученные файл был
обработан скриптом uniprot-to-taxonomy.py. Полученная таксономия была перенесена в основную таблицу
с помощью ВПР (VLOOKUP). Также была добавлена колонка с длиной выбранного домена из каждой последовательности.
Из полученной таблицы отбирались последовательности каждой из рассматриваемых доменных архитектур. Для каждой из архитектур отбиралось 60 последовательностей,
30 из которых принадлежали подтаксону "Fungi" (далее обозначается как F), а оставшиеся 30 - "Metazoa" (обозначается M)
.
Критериями выбора последовательности служили одинаковая длина домена (примерно 70 п.н.) и хорошее представительство подтаксонов в выборке.
Кроме того, к последовательностям была добавлена та, для которой была извествна 3D структура, несмотря на то, что она имеет другую
доменную архитектуру.
Таким образом было отобрано 121 последовательность.
Выравнивание отобранных последовательностей
Из общего выравниваня последовательностей данного семейства доменов (ссылка была приведена выше) были оставлены только отобранные последовательности.
Это производилось с помощью скрипта filter-alignment.py:
python filter-alignment.py -i pr11.fasta -m 1 -o pr11_filter.fasta -a "/"
В итоге было получено выравнивание из 97 последовательностей.
Из полученного выравнивания были удалены пустые колонки и некоторые последовательноти, неправильно выровненные с остальными. Также были одалены N- и C- концевые
участки последовательностей.
Ссылка на полученный проект JalView: pr11_filter_final.jvp.
Построение дерева последовательностей домена
По выравниванию последоовательностей доменов было построен дерево методом Maximum Likelihood. Оно было укоренено в среднюю точку с помощью программы retree
пакета PHYLIP. Изображение полученного дереао приведено на рис. 3.
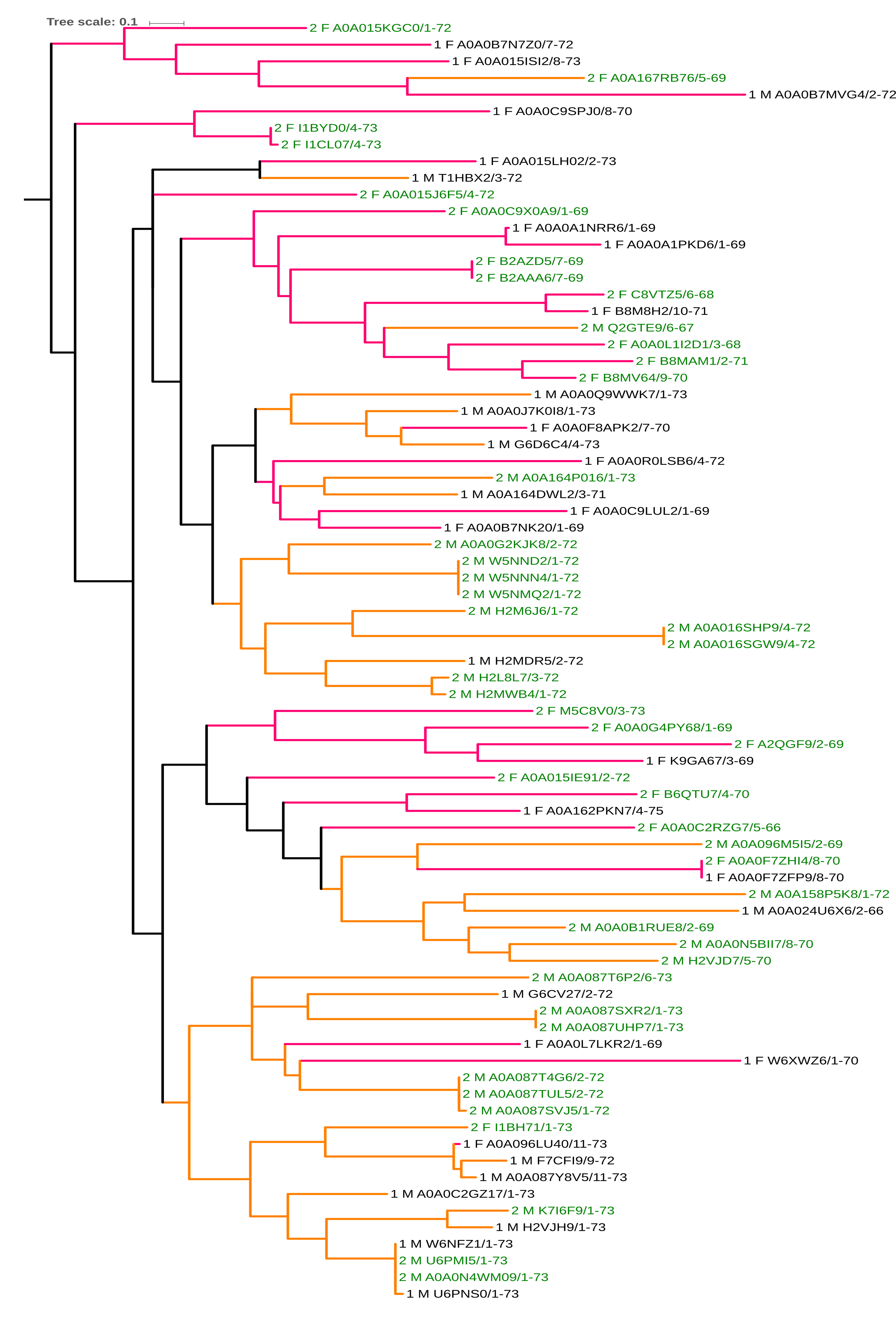 |
| Рис. 3. Дерево, построенное по последовательностям домена методом Maximum Likelihood. Зеленым цветом обозначены названия последовательностей,
относящихся к доменной архитектуре №2, черным - №1. Ветви раскрашены по подтаксонам: розовые - Fungi (F), оранжевые - Metazoa(M). |
Скобочная структура дерева: tree.nwk
В полученном дереве можно выделить отдельные клады, соответствующие подстаксонам F и M (хотя он и не совсем "чистые"). Однако распределение доменных архитектур
имеет гораздо более сложную структуру. Можно предположить, что построенное дерево не совсем правильное, т.к. длина домена довольно мала (70 a.о.), а учитывая, что
дерево было построено по выравниванию, то оно было построено по более меньшему количеству позиций (48).
