Занятие 4
Построение дерева по нуклеотидным последовательностям
Последовательности 16S рибосомальной РНК каждой из бактерий были получены из
базы полных геномов NCBI. Соответствующие последовательности для каждой бактерии из файлов с расширением .frn были объединены в файл
all_16s.fasta. Последовательности были выровнены в JalView с помощью Muscle with Defaults. Выравнивание сохранено в файл
align_16s.fasta. Данный файл был импортирован в программу MEGA и построено дерево с помощью метода Maximum Likelihood.
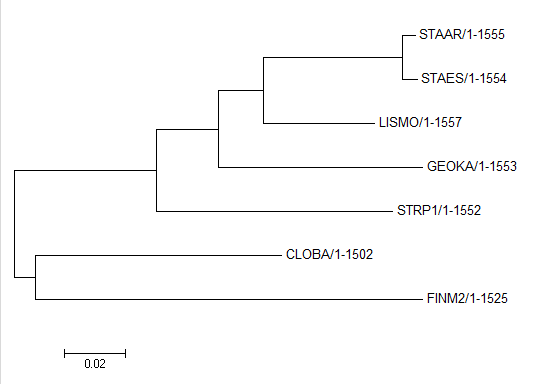
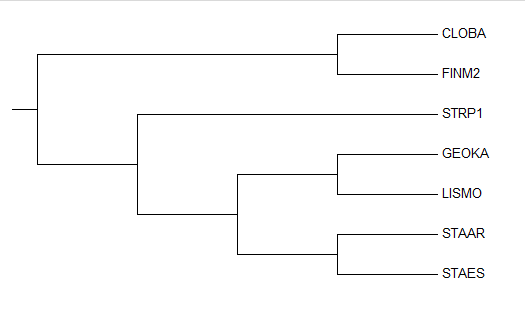
На рис.1 представлено полученное дерево (слева), а также исходное дерево (справа).
Рис.1 Филогенетичское дерево бактерий, построенное по последовательностям 16S рРНК методом Maximum Likelihood (слева).
Исходное дерево (справа).
Ветви исходного дерево с указанием наличия в новом дереве, построенном по 16S рРНК методом ML:
{CLOBA, FINM2} против {STRP1, GEOKA, LISMO, STAAR, STAES} - есть
{CLOBA, FINM2, STRP1} против {GEOKA, LISMO, STAAR, STAES} - есть
{CLOBA, FINM2, STRP1, GEOKA, LISMO} против {STAAR, STAES} - есть
{CLOBA, FINM2, STRP1, STAAR, STAES} против {GEOKA, LISMO} - нет
Вместо последней ветви в новом дереве присутствует другая ветвь:
{CLOBA, FINM2, STRP1, GEOKA} против {STAAR, STAES, LISMO}
Полученное дерево совпадает тем, что было построено методом Neighbor-Joining using % identity по выравниванию белков RSMH.
Построение и анализ дерева, содержащего паралоги
Из файлов, лежащих в директории P:\y15\term4\Proteomes был создан объединенный файл all.fasta с протеомами отобранных бактерий:
gunzip *.fasta
cat *.fasta >> all.fasta
Программой blastp был проведен поиск гомологов белка CLPX_BACSU:
makeblastdb -in all.fasta -dbtype prot -out db.fasta
blastp -query CLPX.fasta -evalue 0.001 -db db.fasta -out outfile.txt
результаты представлены в файле outfile.txt . Всего было найдено 33 гомолога с evalue <= 0.001. Но для построения дерева были выбраны первые 20
последовательностей. Выравнивание этих белков с помощью Muscle представлено на рис.2.
 |
| Рис. 2. Выравнивание гомологов белка CLPX_BACSU среди отобранных бактерий
| |
На основе этого выравнивания в MEGA было построено филогенетическое дерево методом Neighbor-Joining (рис.3). На нем также обозначены основные эволюционные события: дупликации
гена, разделение путей эволюции белков в результате видообразования, а также ортологи и паралоги.
*Два гомологичных белка будем называть ортологами, если они:
а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования.
Два гомологичных белка из одного организма будем называть паралогами.
 |
Рис. 3. Филогенетическое дерево, построенное по выравниванию гомологов белка CLPX_BACSU. Обозначения:
розовый круг - дупликация гена;
зеленый круг - разделение белков в результате видообразования;
желтая рамка - ортологи;
оранжевая рамка - паралоги.
| |