Anna Zheltova
Seventh term (Седьмой семестр):
Electron density in PyMol (Электронная плотность в PyMol)
Crystal recovery in PyMol (Восстановление кристалла в PyMol)
Expansion of a function in a Fourier series (Разложение функции в ряд Фурье)
Analysis of the NMR file (Анализ ЯМР-файла)
Definition of the secondary structure (Определение вторичной структуры)
Locating hydrophobic clusters (Нахождение гидрофобных кластеров)
Surface construction and analysis in PyMOL (Построение и анализ поверхностей в PyMOL)
Задание 1. Построение изображения электронной плотности вокруг остова полипептидной цепи
К сожалению, белка, с которым я работала в первом семестре, нет в БД PDB. Поэтому была выбрана дезоксиуридин-5"-трифосфат-нуклеотидгидролаза (дУТФаза) Escherichia coli (DEOXYURIDINE 5'-TRIPHOSPHATE NUCLEOTIDO HYDROLASE (D-UTPASE) ). Идентификатор в БД PDBe 1DUP . Идентификатор в БД PDB 1DUP .
Фермент dUTPase катализирует гидролиз dUTP и поддерживает низкую внутриклеточную концентрацию dUTP, так что урацил не может быть включен в ДНК. dUTPase из Escherichia coli строго специфична для ее субстрата dUTP за счет активного центра, различающего нуклеотиды по отношению к сахарному фрагменту, а также пиримидинового основания. Данная трехмерная структура 1DUP была получена группой ученых в составе: Cedergren-Zeppezauer ES, Larsson G, Nyman PO, Dauter Z, Wilson KS. Статья Crystal structure of a dUTPase (PMID:1311056; DOI: 10.1038/355740a0) была опубликована в Nature [01 Feb 1992, 355(6362):740-743]. С текстом статьи можно ознакомиться по этой ссылке.
В статье рассматривают трехмерную структуру DUTPase E. coli, определенную рентгеновской кристаллографией с разрешением 1,9 А. Фермент представляет собой симметричный тример, 152 аминокислотных остатка в субъединице, первые 136 видны в кристаллической структуре. Третичная структура напоминает "jelly-roll" складку и не показывает «классический» нуклеотидсвязывающий домен. В четвертичной структуре существует сложное взаимодействие между субъединицами, которое может иметь важное значение для катализа.
Для данного белка в БД есть EDS map.Наличие электронной плотности означает, что автор модели положил в PDB не только саму модель, но и экспериментальные данные. Экспериментальные данные представлены в документе "Файл структурных факторов" (Strucure factors).
Проверка PDBeFold -> launch -> Query -> 1DUP показала, что в таблице с находками было 6 разных белков с RMSD (колонка с оценкой сходства) от 0.8 до 3.0 ангстрем и N_align (колонка с числом сопоставленных Cα) от половины до 90% от числа аминокислотных остатков входного белка.
Перейдем к построению изображения электронной плотности вокруг остова полипептидной цепи:
Для построения изображения использовали следующие команды:
load pdb1dup.ent, pdb1dup #загружаем PDB-файл для белка, содержащий изображение поверхности.
load 1dup.ccp4, 1dup #загружаем файл, содержащий данные об электронной плотности.
hide (resn HOH)
isomesh new_surface, 1dup, 1.5, visible, 2.5 #new_surface- имя нового объекта с изображением поверхности, 1dup - имя объекта с электронной плотностью, 1.5 - уровень - значения электронной плотности, по которым строится поверхность, visible - множество, электронную плотности вокруг которого надо изобразить, 2.5 - аргумент – "buffer".
Получившиеся результаты визуализации электронной плотности с помощью программы PyMOL с различными уровенями подрезки

Рис.1.Уровень подрезки электронной плотности = 0. Остов полипептидной цепи выделен синим.

Рис.2.Уровень подрезки электронной плотности = 1. Остов полипептидной цепи выделен синим.

Рис.3.Уровень подрезки электронной плотности = 1,5. Остов полипептидной цепи выделен синим.

Рис.4.Уровень подрезки электронной плотности = 2. Остов полипептидной цепи выделен синим.

Рис.5.Уровень подрезки электронной плотности = 2,5. Остов полипептидной цепи выделен синим.
Перейдем к построению изображения электронной плотности вокруг трех различных аминокислотных остатков.
Для выполнения данного задания были выбраны следующие аминокислотные остатки: ala, trp, his.
Построение изображения электронной плотности вокруг ala.
Для построения электронной плотности вокруг ala с помощью программы PyMOL были использованы следующие команды:
load pdb1dup.ent, pdb1dup #загружаем PDB-файл для белка, содержащий изображение поверхности.
load 1dup.ccp4, 1dup #загружаем файл, содержащий данные об электронной плотности.
hide all #скрываем все
show sticks, resn ala #отображаем только все ala, входящие в состав белка 1dup
select ala, resi 24 #был выбран аланин под номером 24
show sticks, ala #отображаем только выбранный аланин
isomesh new_sur, 1dup, 1.0, ala, 1.6 #1.0 -уровень - значения электронной плотности, по которым строится поверхность, варьировали от 1,0 до 3,0 с шагом в 0,5; значение аргумента 1.6 было выбрано для более удобного отображения электронной плотности (для уменьшения отображения ЭП окружения, не относящейся к данной аминокилоте).
Получившиеся результаты визуализации электронной плотности вокруг ala с помощью программы PyMOL с различными уровенями подрезки
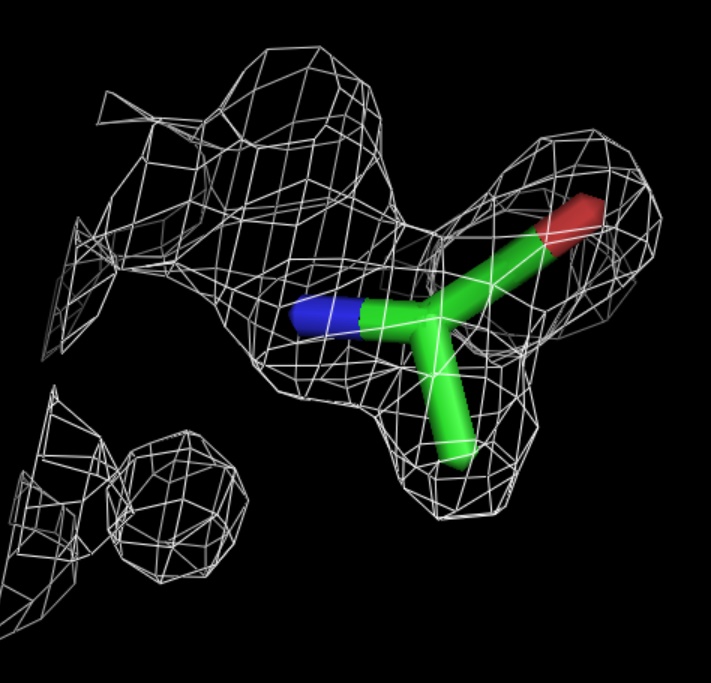
Рис.6.Уровень подрезки электронной плотности = 1,0.
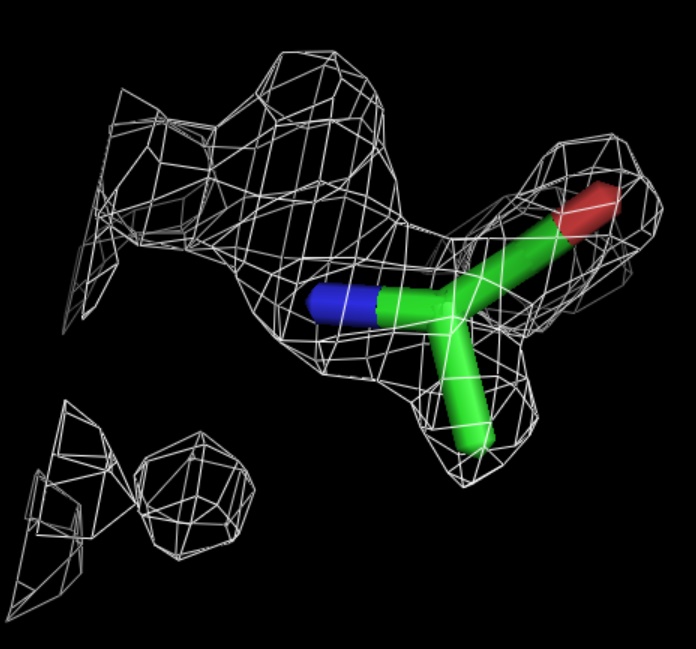
Рис.7.Уровень подрезки электронной плотности = 1,5.

Рис.8.Уровень подрезки электронной плотности = 2,0.
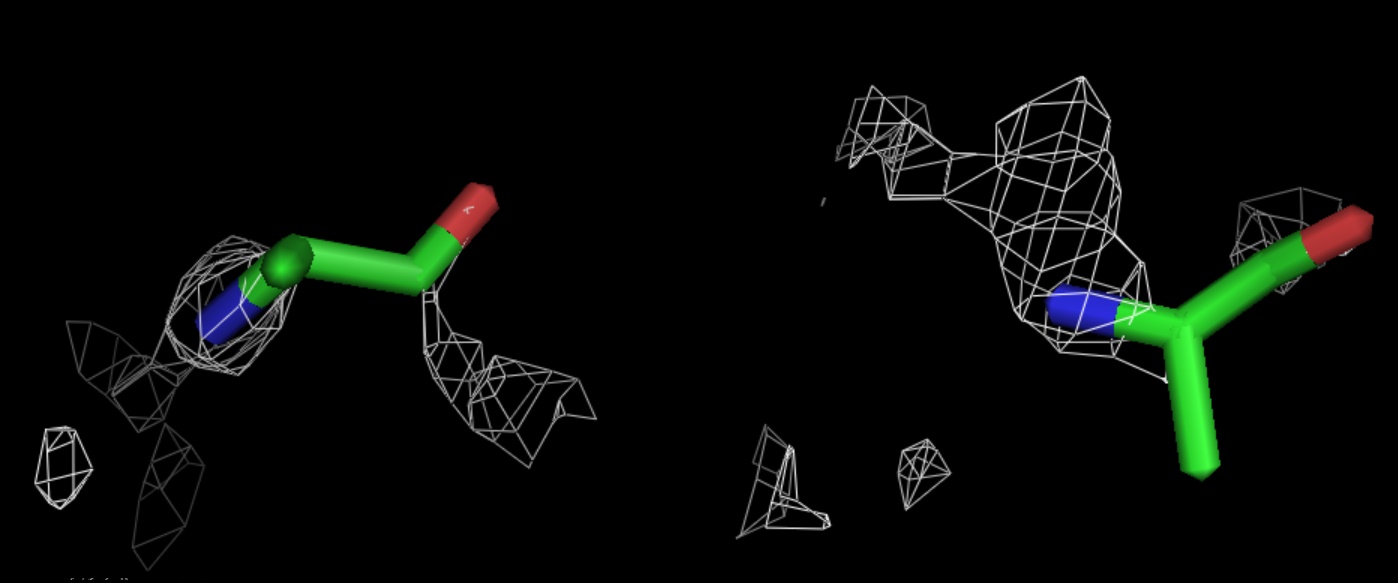
Рис.9.Уровень подрезки электронной плотности = 2,5.
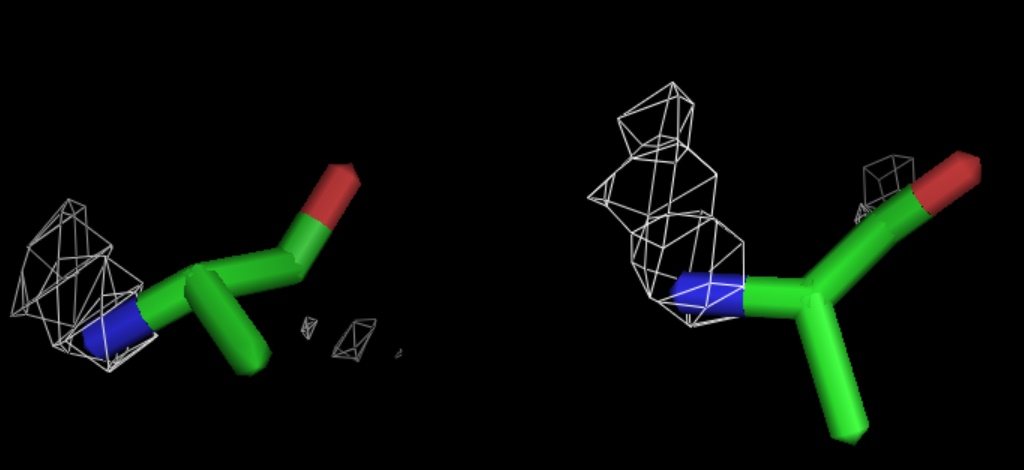
Рис.10.Уровень подрезки электронной плотности = 3,0.
Построение изображения электронной плотности вокруг trp.
Для построения электронной плотности вокруг trp с помощью программы PyMOL были использованы следующие команды:
#предыдущие команды аналогичны описанным для ala
show sticks, resn trp #отображаем только все ala, входящие в состав белка 1dup
select trp, resi 102 #был выбран trp под номером 102
show sticks, trp #отображаем только выбранный trp
isomesh new_sur, 1dup, 1.0, trp, 0.7 #1.0 -уровень - значения электронной плотности, по которым строится поверхность, варьировали от 1,0 до 3,0 с шагом в 0,5; значение аргумента 0.7 было выбрано для более удобного отображения электронной плотности (для уменьшения отображения ЭП окружения, не относящейся к данной аминокилоте).
Получившиеся результаты визуализации электронной плотности вокруг trp с помощью программы PyMOL с различными уровенями подрезки
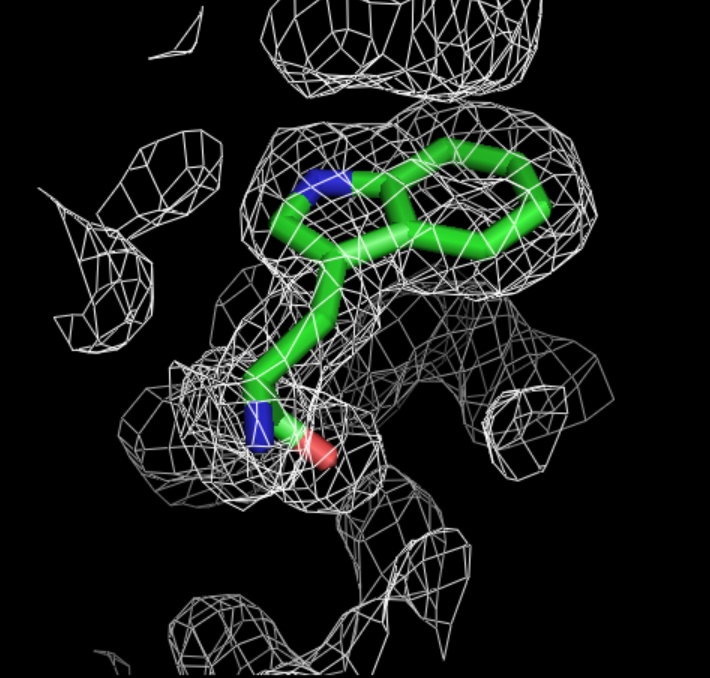
Рис.11.Уровень подрезки электронной плотности = 1,0.
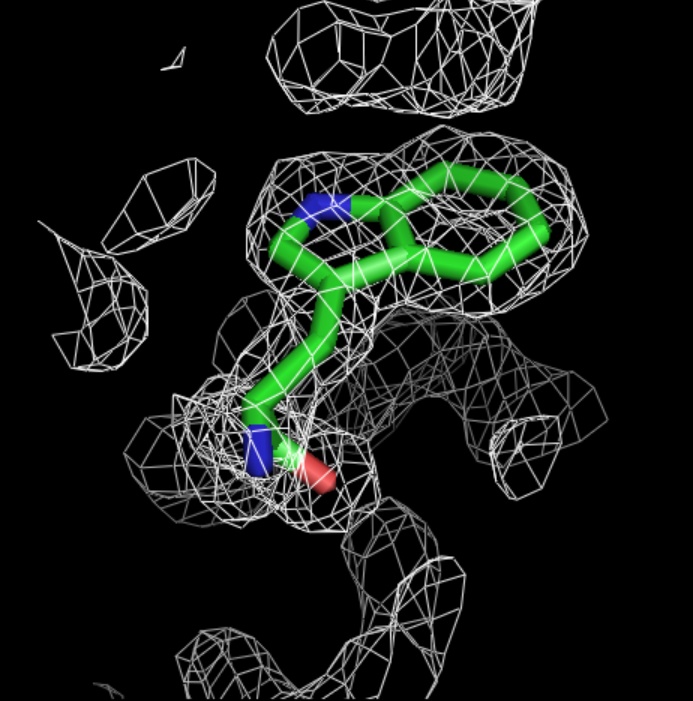
Рис.12.Уровень подрезки электронной плотности = 1,5.
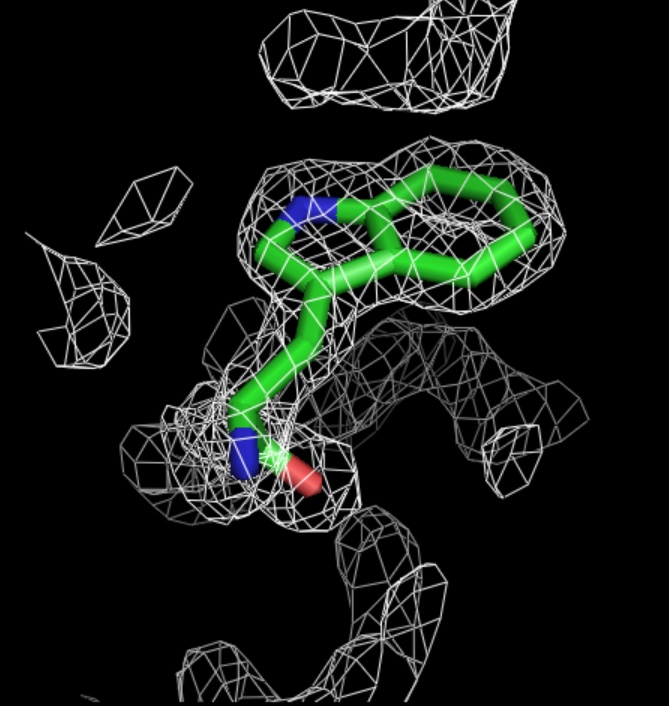
Рис.13.Уровень подрезки электронной плотности = 2,0.
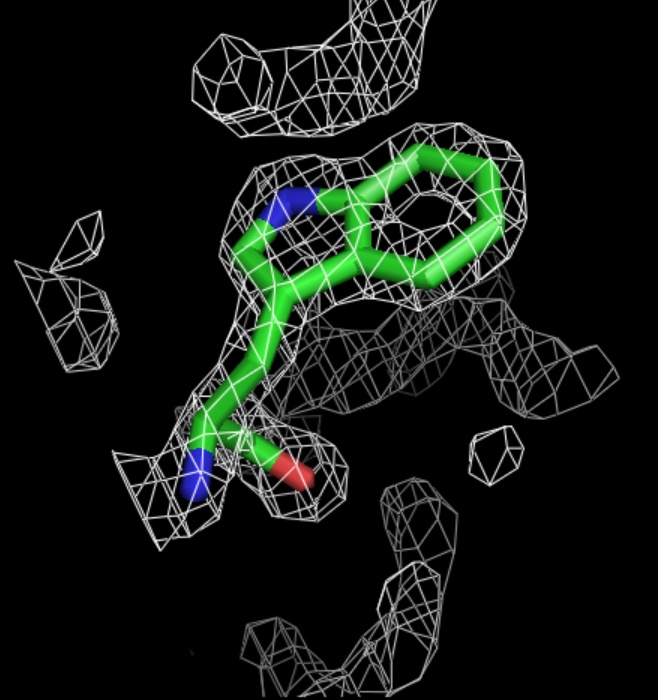
Рис.12.Уровень подрезки электронной плотности = 2,5.
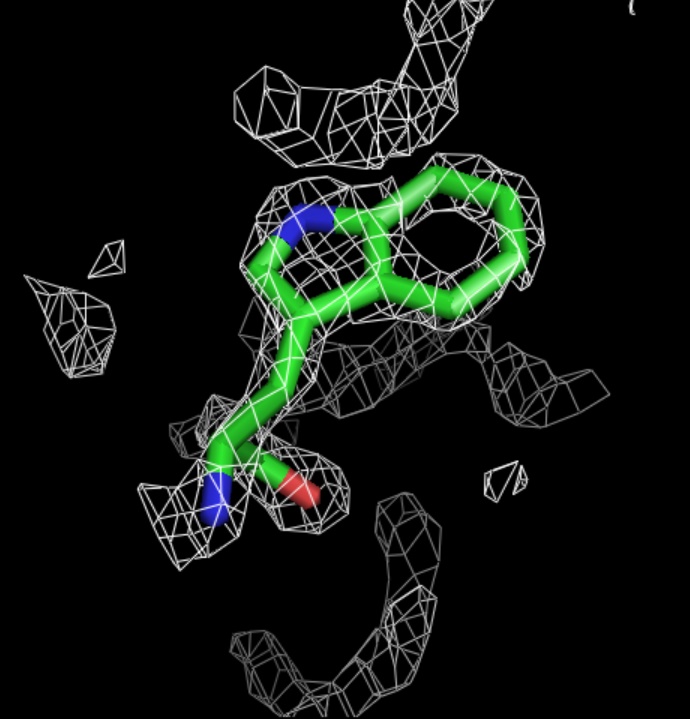
Рис.13.Уровень подрезки электронной плотности = 3,0.
Построение изображения электронной плотности вокруг his.
Для построения электронной плотности вокруг his с помощью программы PyMOL были использованы следующие команды:
#предыдущие команды аналогичны описанным для ala
show sticks, resn his #отображаем только все his, входящие в состав белка 1dup
select his, resi 76 #был выбран his под номером 76
show sticks, his #отображаем только выбранный his
isomesh new_sur, 1dup, 1.0, his, 1.0 #1.0 -уровень - значения электронной плотности, по которым строится поверхность, варьировали от 1,0 до 3,0 с шагом в 0,5; значение аргумента 1,0 было выбрано для более удобного отображения электронной плотности (для уменьшения отображения ЭП окружения, не относящейся к данной аминокилоте).
Получившиеся результаты визуализации электронной плотности вокруг his с помощью программы PyMOL с различными уровенями подрезки
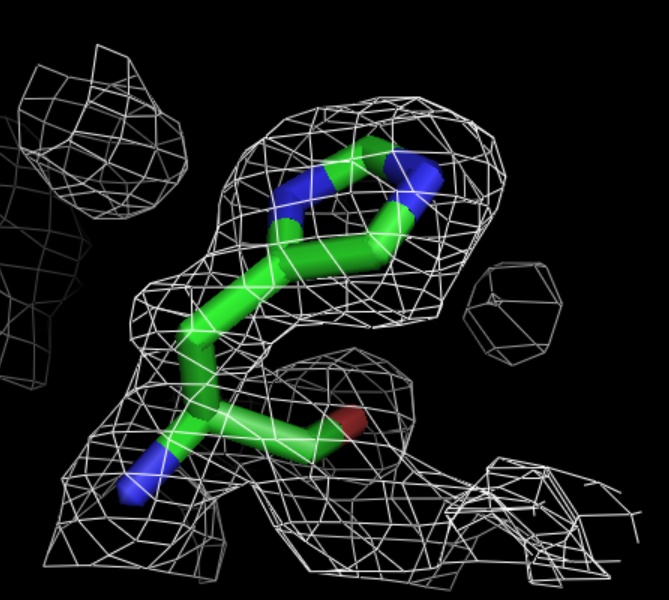
Рис.11.Уровень подрезки электронной плотности = 1,0.
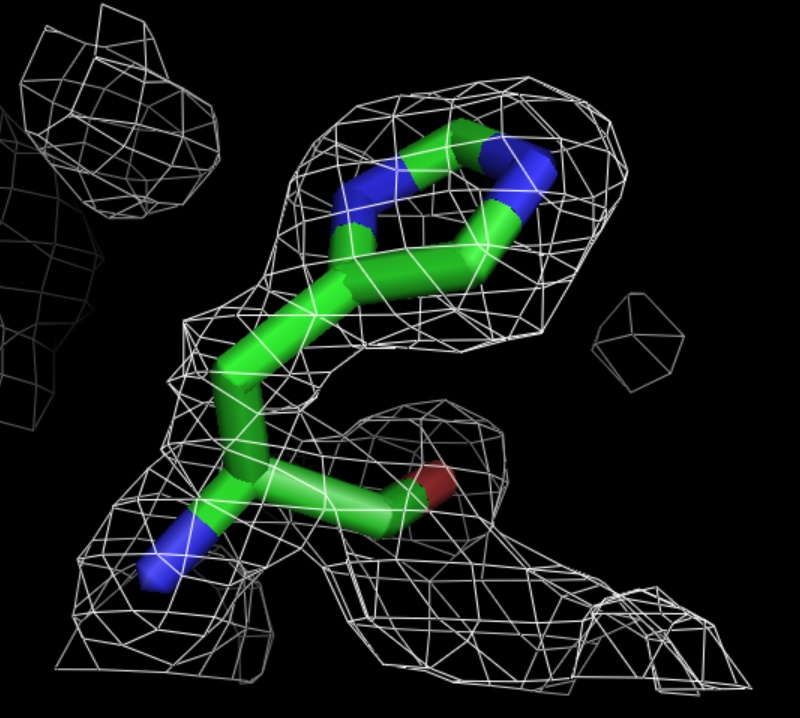
Рис.12.Уровень подрезки электронной плотности = 1,5.
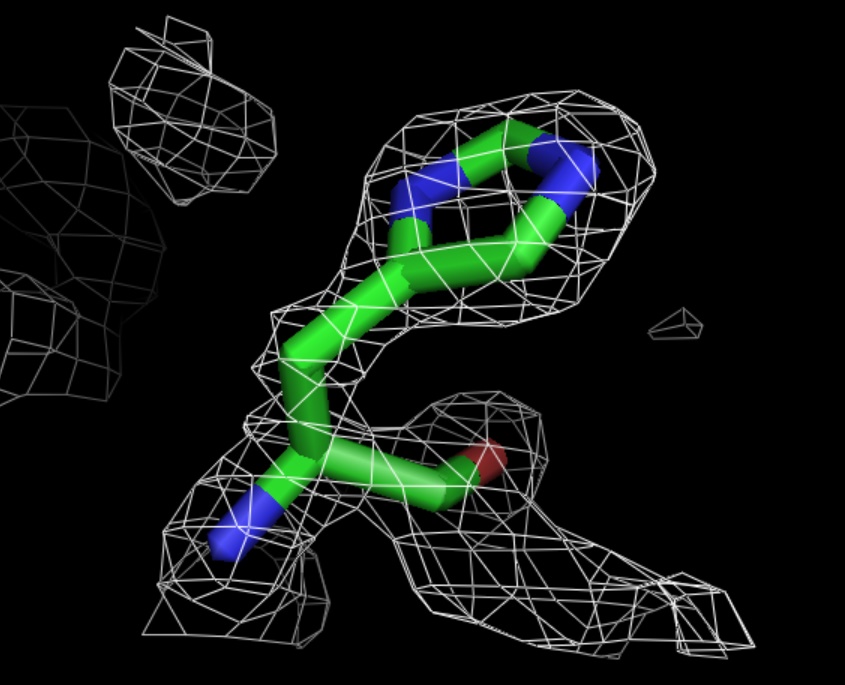
Рис.13.Уровень подрезки электронной плотности = 2,0.
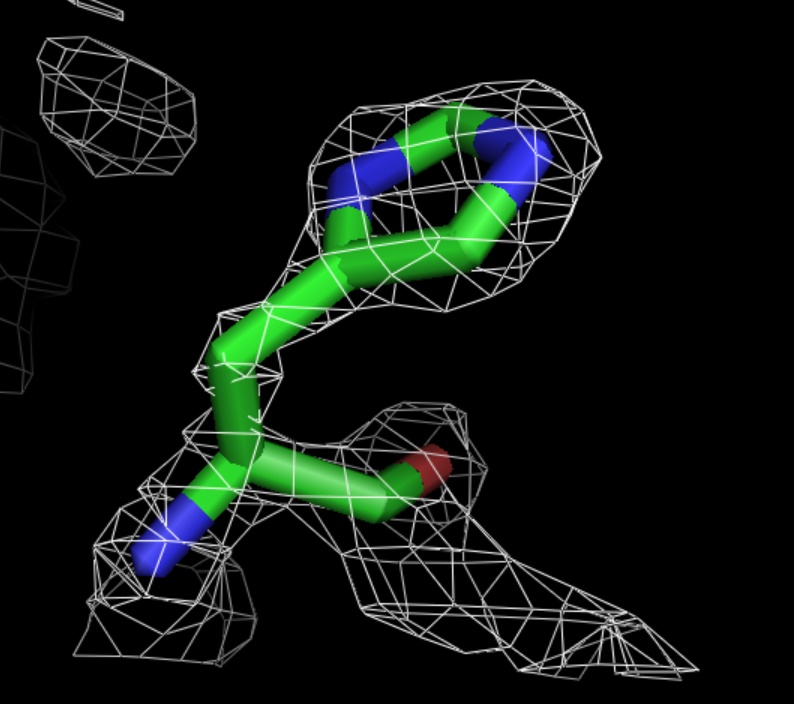
Рис.12.Уровень подрезки электронной плотности = 2,5.
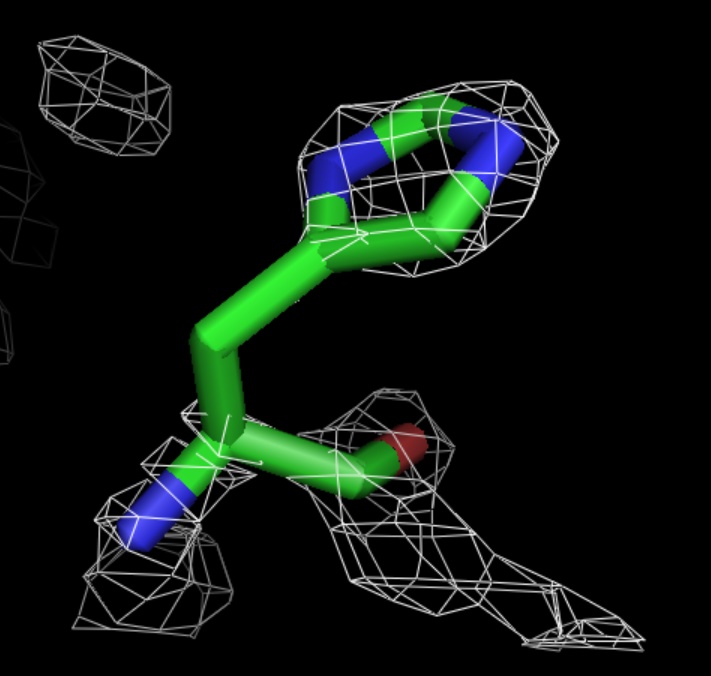
Рис.13.Уровень подрезки электронной плотности = 3,0.
Обсуждение и выводы:
Для уровнях подрезки электронной плотности 1,0 и 1,5, электронная плотность окружает все атомы, входящие в состав аминокислот во всех рассмотренных случаях (ala, his, trp).
Однако при повышении уровня подрезки электронной плотности до 2,5 в случае ala и 3,0 в случае his, не все атомы окружены электронной плотностью. В случае trp при изменении уровня подрезки от 1,0 до 3,0 все атомы окружены электронной плотностью.
Таким образом, при использовании различных уровней подрезки можно выяснить, что координаты атомов соответствуют сгущениям электронной плотности, однако при изменении уровня подрезки не все атомы становятся покрыты сеткой электронной плотности.
Разрешение структуры составляет 1,9 ангстрем, что, по-моему мнению, является недостаточным для утверждения, что все аминокислоты точно описываются картой электронной плотности, так как работать с уровнем подрезки 1-1,5 достаточно сложно, а при увеличении уровня наблюдается отсутствие сетки электронной плотности у отдельных атомов. Суммируя предыдущие выводы, я считаю, что с данным разрешением установить точное расположение атомов достаточно сложно.