Anna Zheltova
Seventh term (Седьмой семестр):
Electron density in PyMol (Электронная плотность в PyMol)
Crystal recovery in PyMol (Восстановление кристалла в PyMol)
Expansion of a function in a Fourier series (Разложение функции в ряд Фурье)
Analysis of the NMR file (Анализ ЯМР-файла)
Definition of the secondary structure (Определение вторичной структуры)
Locating hydrophobic clusters (Нахождение гидрофобных кластеров)
Surface construction and analysis in PyMOL (Построение и анализ поверхностей в PyMOL)
Определение вторичной структуры
В данном задании необходимо было определить вторичную структуру белка. Для определения была выбрана структура белка 1DUP. Подробнее о структуре данного белка можно почитать в предыдущих отчетах (validation).
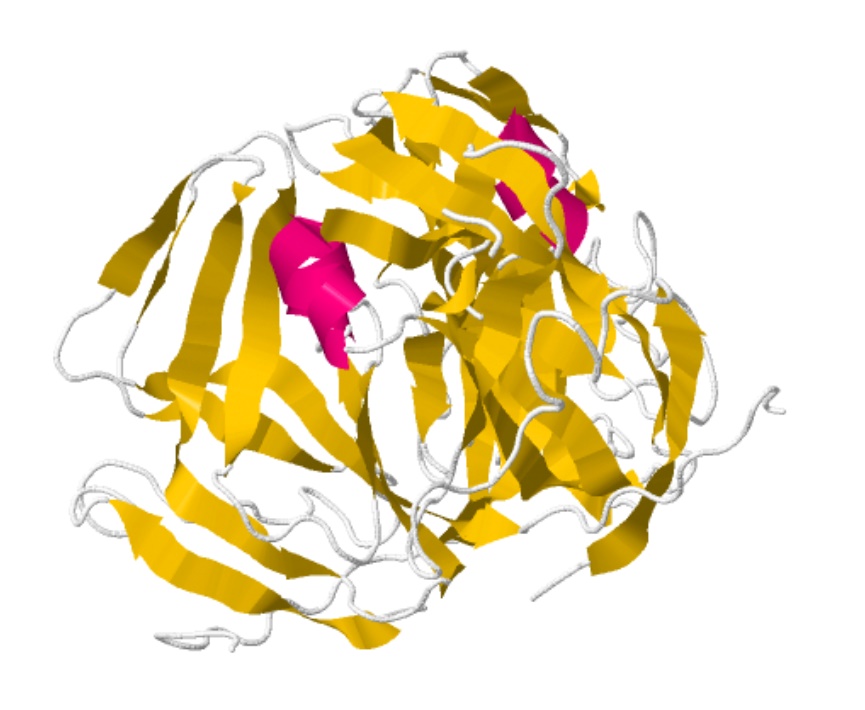
Белок 1DUP (раскраска по структуре).
Вторичная структура была определена с помощью программ DSSP.
Выдача программы DSSP представлена в данном файле .
Проведем сравнение границ четырёх элементов вторичной структуры, полученных программой, с границами, приведенными в заголовке PDB файла.
Однако для начала сравним сами выдачи:
В заголовке PDB файла была найдена следующая информация о вторичной структуре
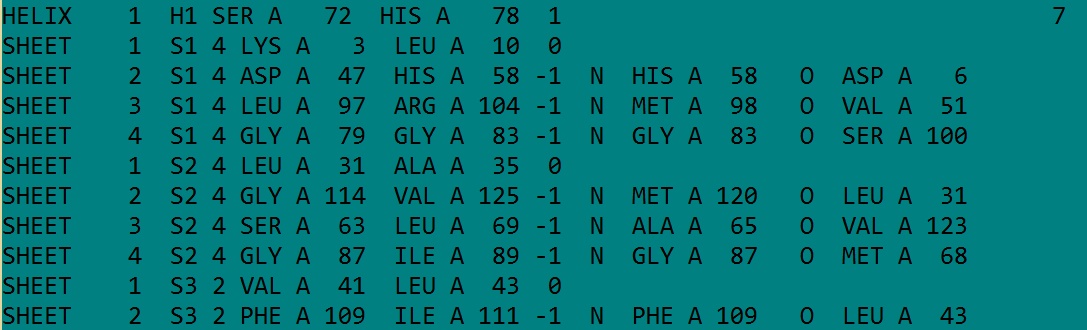
Из PDB файла следует, что белок с идентификатором в БД PDB 1DUP содержит одну альфа-спираль и 10 бета-листов.
Рассмотрим данные, полученные с помщью DSSP (для лучшего восприятия визуализируем выдачу):

Из выдачи следует, что белок с идентификатором в БД PDB 1DUP содержит одну альфа-спираль и 10 бета-листов. При это также имеются 2 участка "extended strand, participates in β ladder ", обозначенные "E", поэтому можно утверждать, что бета-листов 12.
При сравнении двух выдачей было замечено различие в количестве обнаруживаемых бета-листов, однако это не удивительно, так как структура была расшифрована в 1995 году, а последняя версия DSSP вышла в 2017.
Проведем сравнение границ элементов вторичной структуры.
Для этого визуализирую похожим образом информацию из PDB файла:

Сравним две выдачи:

Наблюдаются отличия в границах бета-листов, однако границы альфа-спирали совпадают в обоих случаях и встречаются бета-листы, в которых границы все же совпадают. Скорее всего это связано с прогрессом методов определения вторичной структуры по последовательности. Видно, что DSSP дает лучшую аннотацию, в которую включены не только классические альфа-спираль и бета-лист.
При рассмотрении структуры с помощью DSSP, было найдено несколько редких элементов: два бета-мостика (VAL14 и PHE18).

Интересно, что данные бета-мостики расположены рядом, а перед каждым из этих мостиков находится поворот.
Были найдены повороты: PRO12-ARG13, GLY15-GLU17, SER26-GLY27, PRO45-GLY46, PRO62-SER63, GLY83-ASN84, SER91-ASP92, PRO113-GLY114.
Поворотов достаточно много для такого маленького белка, примечательно, что большинство поворотов - это скорее полуповороты, состоящие из 2-3 аминокислот.
Прочитанная литература о том, что полуповорот состоит из 3 аминокислот, а не из двух, привела к мысли о проверке данной аннотации. Сравним аннотацию, полученную с помощью DSSP и STRIDE.
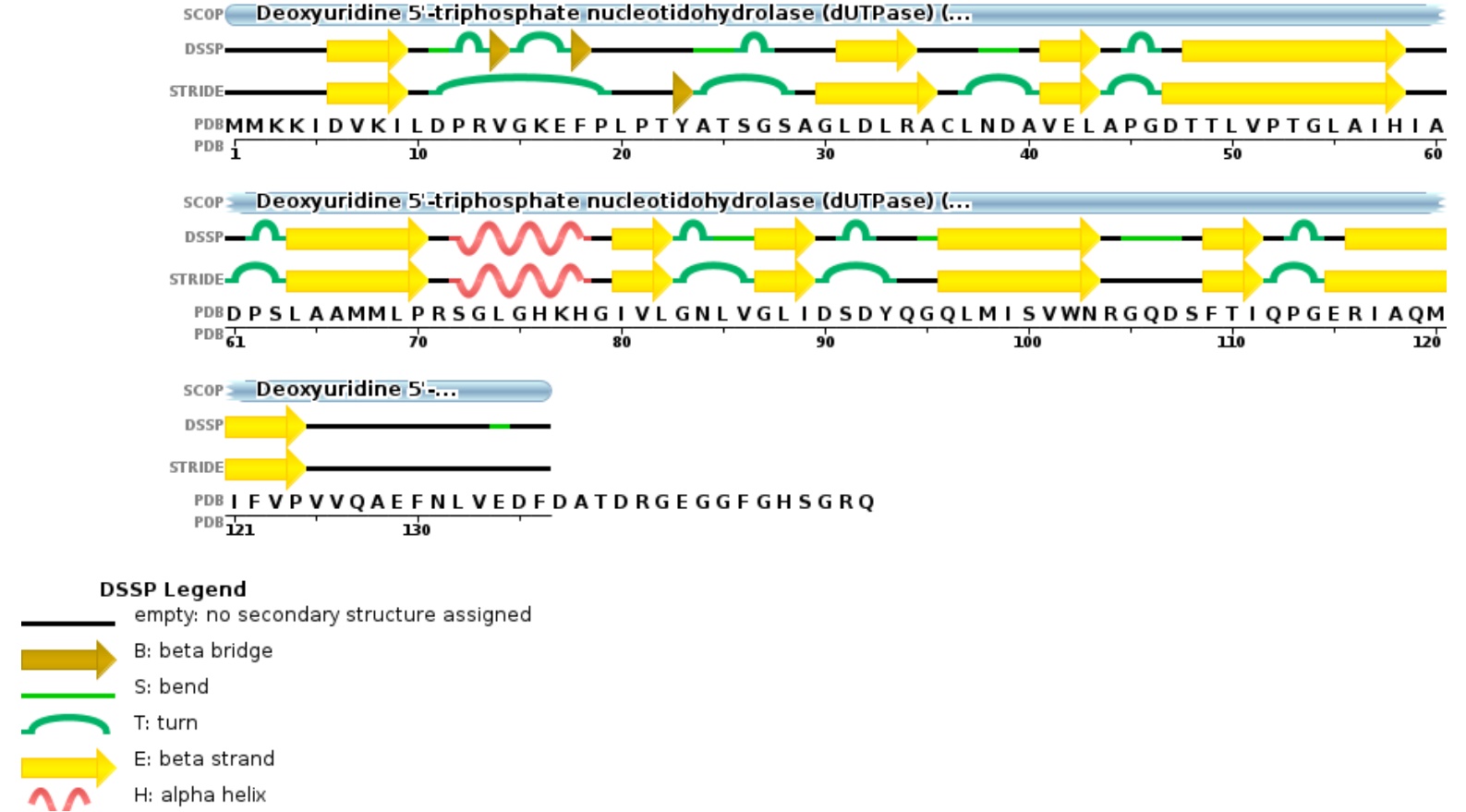
Данные по поротам, полученные программой STRIDE показались мне более достоверными, так как полуповороты состоят минимум из 3 аминокислот. Итересно, что STRIDE находит 10 бета-листов и 1 бета-мостик. При этом границы элементов вторичных структур не отличаются для альфа спирали и для всех бета-листов, кроме трех. При этом отличие на 1-2 аминокислоты больше/меньше перед/после поворота. Возможно, данные программы используют разные критерии для аннотации.
В заключении хотелось бы отметить, что различные данные полученные с помощью программ и из исходного PDB-файла отличаются. Отличие от PDB файла объяснимо улучшениями методов предсказания и расшифровки структур, а вот отличие между двумя оставшимися программами, с моей точки зрения, скорее всего объяснимы разными пороговыми критериями.