Занятие 5: Банк EMBL
Знакомство со структурой банка EMBL посредством поисковой системы SRS
При выполнении работы использовался SRS на сайте EBI.
Последний релиз EMBL (113) был проиндексирован в системе 18 сентября 2012 года и содержит 82772678 записей.
Список классов банка EMBL (Data Class):
| Обозначение класса | Описание класса | Число записей, проиндексированных SRS для релиза 113 EMBL |
| CON | Constructed sequence | - |
| EST | Expressed Sequence Tag | - |
| GRV | Genome Reviews | - |
| GSS | Genome Survey Sequence | 34528104 |
| HTC | High Throughput cDNA sequencing | 491770 |
| HTG | High Throughput Genome sequencing | 152599 |
| MGA | Mass Genome Annotation | - |
| PAT | Patent | 24364832 |
| SET | Project set (EMBL WGS Masters only) | - |
| STD | Standard | 13920617 |
| STS | Sequence Tagged Site | 1322570 |
| TSA | Transcriptome Shotgun Assembly | 7992186 |
| WGS | Whole Genome Shotgun | - |
Как мы видим, в SRS не проиндексированы записи классов CON, EST, GRV, MGA, SET и WGS.
Список разделов банка EMBL (Division):
| Обозначение класса | Описание класса | Число записей в релизе 113 EMBL |
| ENV | Пробы среды (Environmental Samples) | 7762556 |
| FUN | Грибы (Fungi) | 2402829 |
| HUM | Человек (Human) | 11304977 |
| INV | Беспозвоночные (Invertebrates) | 7398340 |
| MAM | Другие млекопитающие (Other Mammals) | 6741732 |
| MUS | Домовая мышь (Mus musculus) | 5163724 |
| PHG | Бактериофаги (Bacteriophage) | 8503 |
| PLN | Растения (Plants) | 20284404 |
| PRO | Прокариоты (Prokaryotes) | 1639517 |
| ROD | Грызуны (Rodents) | 1313761 |
| SYN | Искусственные (Synthetic) | 4045013 |
| TGN | Трансгенные (Transgenic) | 285306 |
| UNC | Неклассифицированные (Unclassified) | 8617170 |
| VRL | Вирусы (Viruses) | 1358516 |
| VRT | Другие позвоночные (Other Vertebrates) | 4446330 |
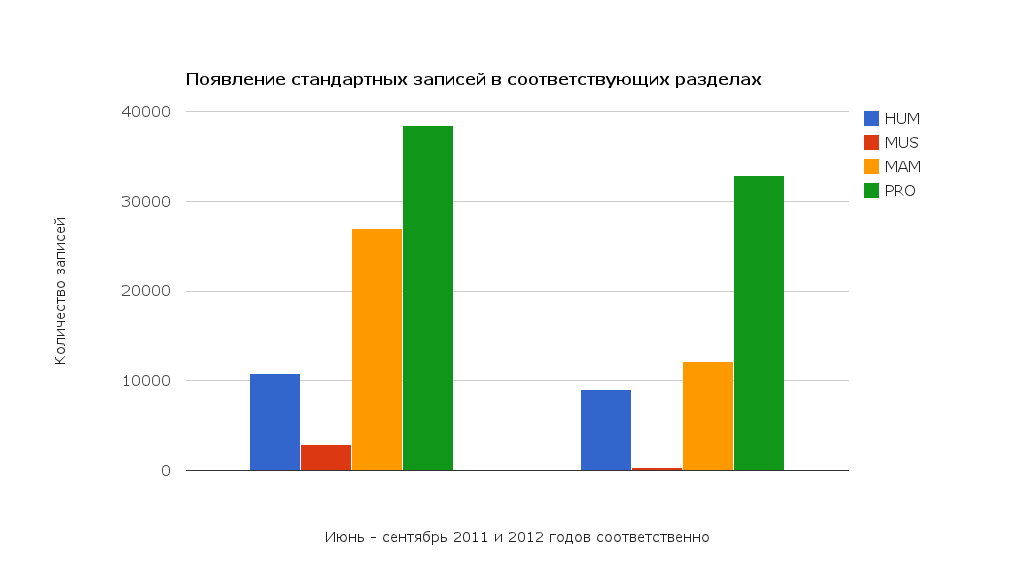
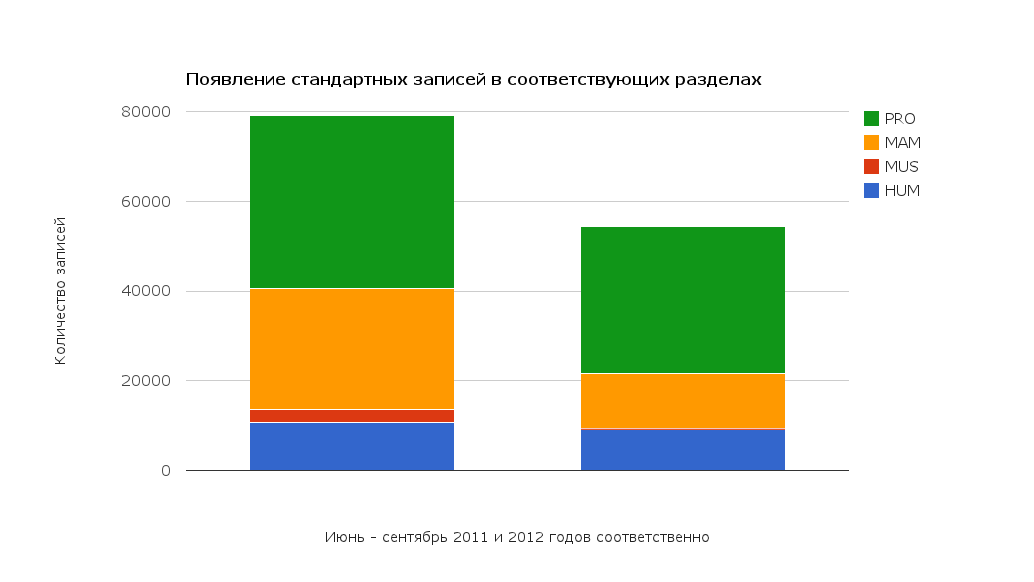
Выберем несколько разделов банка EMBL, например, HUM, MUS, MAM и PRO. Определим, сколько стандартных (класса STD) записей появилось в каждом из этих разделов за июнь - сентябрь 2012 года и за тот же период 2011 года (для этого можно воспользоваться расширенной формой поиска - Extended Query form). Результаты в виде столбчатой диаграммы и диграммы накопления приведены ниже.
Как мы видим, темп поступления стандартных записей для каждого из рассмотренных разделов снизился, при этом для разделов MUS и MAM весьма значительно. В данном случае можно говорить о замедлении поступления информации (записей класса STD) в указанные разделы.
Описание гена в записи банка EMBL
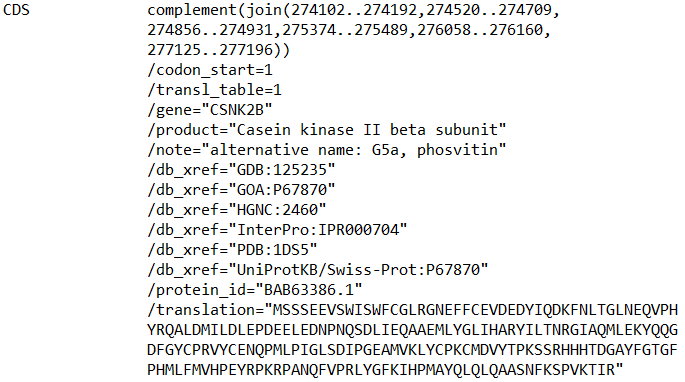
Рассмотрим ген CSNK2B, описанный в записи EMBL с кодом доступа BA000025 (см. файл BA000025.embl).
Для него можно привести следующую информацию:
| Направление гена относительно направления, выбранного для записи | обратное |
| Число кодирующих участков | 6 |
| Длина первого кодирующего участка | 91 |
| Длина последнего кодирующего участка | 72 |
| Длина первого интрона между кодирующими участками | 327 |
| Длина последнего интрона между кодирующими участками | 964 |
Нахождение белка по фрагменту гена
Самым длинным кодирующим участком является второй кодирующий участок (274520..274709). Вырежем его программой seqret, помня об обратном направлении гена относительно направления, выбранного для записи:
seqret BA000025.embl -sask
В результате получим файл csnk2b.fasta.
На домашней странице BLAST запустим blastx. Эта программа сравнивает вероятные продукты трансляции заданной нуклеотидной последовательности (6 вариантов, если учитывать две цепи и 3 возможных рамки считывания) с записями в базе данных белковых последовательностей.
В поле для ввода последовательности введём полученную последовательность кодирующего участка гена CSNK2B. Выберем необходимую базу данных: UniProtKB/Swiss-Prot(swissprot) в соответствующем поле для выбора базы данных для осуществления поиска. Так как исследуемым организмом является человек, то можно ограничить поиск только белками человека, выбрав human (taxid:9606) в поле Organism.
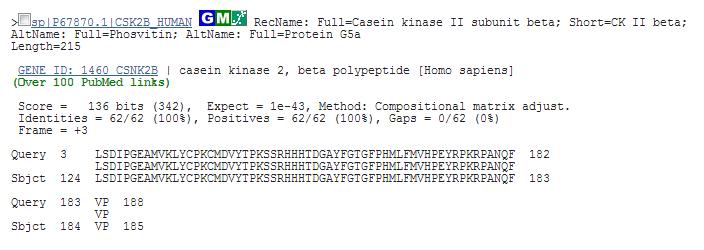
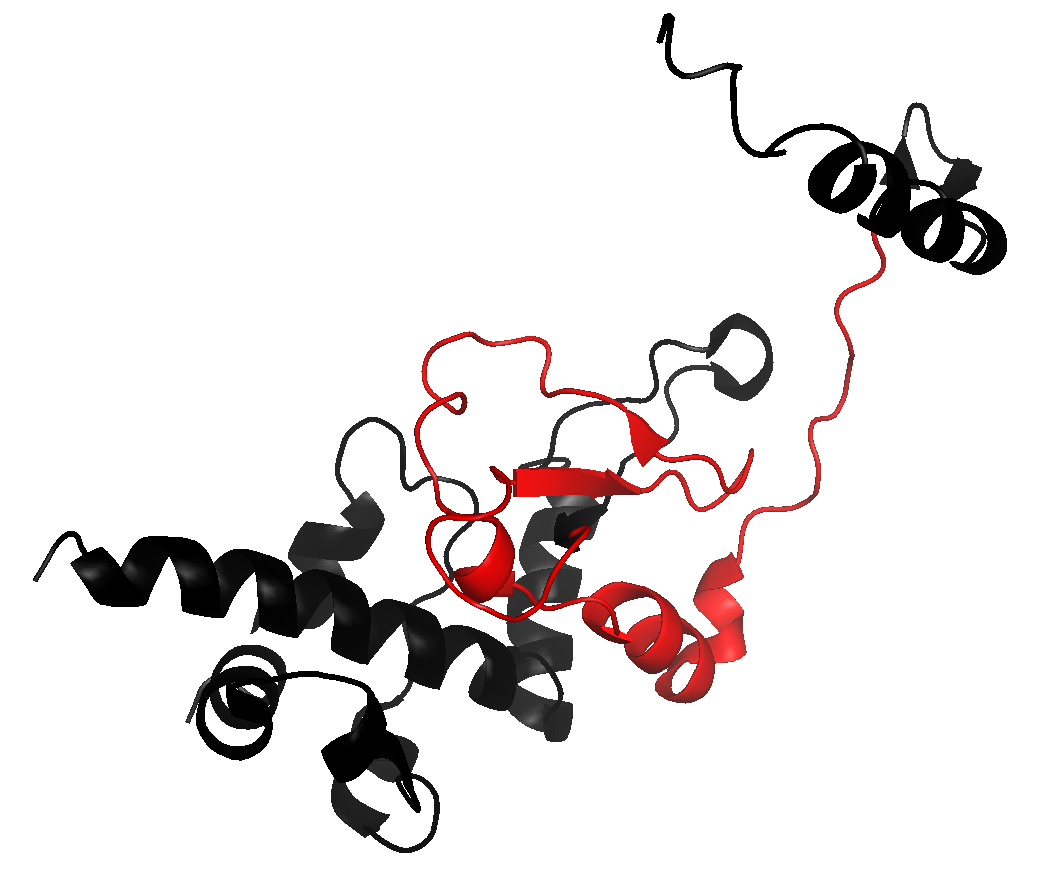
После завершения процесса поиска по последовательности кодирующего участка гена CSNK2B был найден соответствующий белок: Casein kinase II subunit beta (P67870). Как видно из приведённого ниже выравнивания, искомой последовательности кодирующего участка гена соответствует участок 124-185 белка. (В последовательности кодирующего участка предполагается сдвиг рамки считывания на 2 нуклеотидные позиции.)
С помощью программ визуализации пространственных структур макромолекул можно наглядно представить, какой участок белка соответствует рассматриваемому кодирующему участку:
Ссылки из записи банка Swiss-Prot на записи банка EMBL
Исследуемым белком является белок CDD_BACSU. Запись Swiss-Prot этого белка можно получить следующей командой:
entret sw:p19079
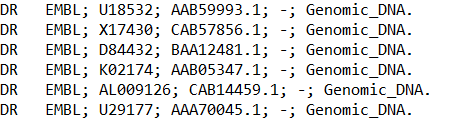
В результате получаем файл cdd_bacsu.entret. В записи содержатся следующие ссылки на банк EMBL:
С использованием SRS заполним таблицу:
Записи EMBL, в которых описан ген белка CDD_BACSU
| ID записи | Тип молекулы | Класс данных | Дата внесения в банк | Описание | Длина последовательности |
| U18532 | геномная ДНК | стандартные (STD) | 1 января 1995 | Ген цитидиндезаминазы (cdd) Сенной палочки и ген G-белка Bex, полный кодирующий участок. | 2089 |
| X17430 | геномная ДНК | стандартные (STD) | 9 декабря 1991 | ДНК Сенной палочки гена cdd, кодирующего цитидин/дезоксицитидин-дезаминазу. | 1170 |
| D84432 | геномная ДНК | стандартные (STD) | 9 мая 1996 | ДНК Сенной палочки, участок длиной 283 Kb, содержащий элемент покрова. | 282700 |
| K02174 | геномная ДНК | стандартные (STD) | 7 ноября 1985 | Фрагмент P43 Сенной палочки, содержащий 2 перекрывающихся промотора. | 476 |
| AL009126 | геномная ДНК | стандартные (STD) | 18 июля 2002 | Полный геном штамма 168 подвида subtilis Сенной палочки. | 4215606 |
| U29177 | геномная ДНК | стандартные (STD) | 19 июля 1995 | Ген PhoH Сенной палочки, фрагмент кодирующего участка; ген диацилглицерол-киназы (dgk), полный кодирующий участок; Ген цитидиндезаминазы (cdd), фрагмент кодирующего участка. | 3346 |
В ходе выполнения работы в SRS была создана таблица, содержащая следующие поля ID, Molecule, Data class, Sequence Length, Entry Creation Date и Description: файл cdd_bacsu_embl_srs.txt.
Ссылки
- Файл BA000025.embl.
- Файл csnk2b.fasta.
- Файл 4dgl.pdb.
- Файл cdd_bacsu.entret.
- Файл cdd_bacsu_embl_srs.txt.