Занятие 3: A- и В- формы ДНК. Структура РНК
- Содержание:
- Средства RasMol для работы со структурами нуклеиновых кислот
- Сравнение структур различных форм ДНК с помощью средств RasMol
- Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
- Исследование структуры тРНК
- Исследование ДНК-белковых взаимодействий в структуре комплекса белка и фрагмента ДНК
Построение модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA
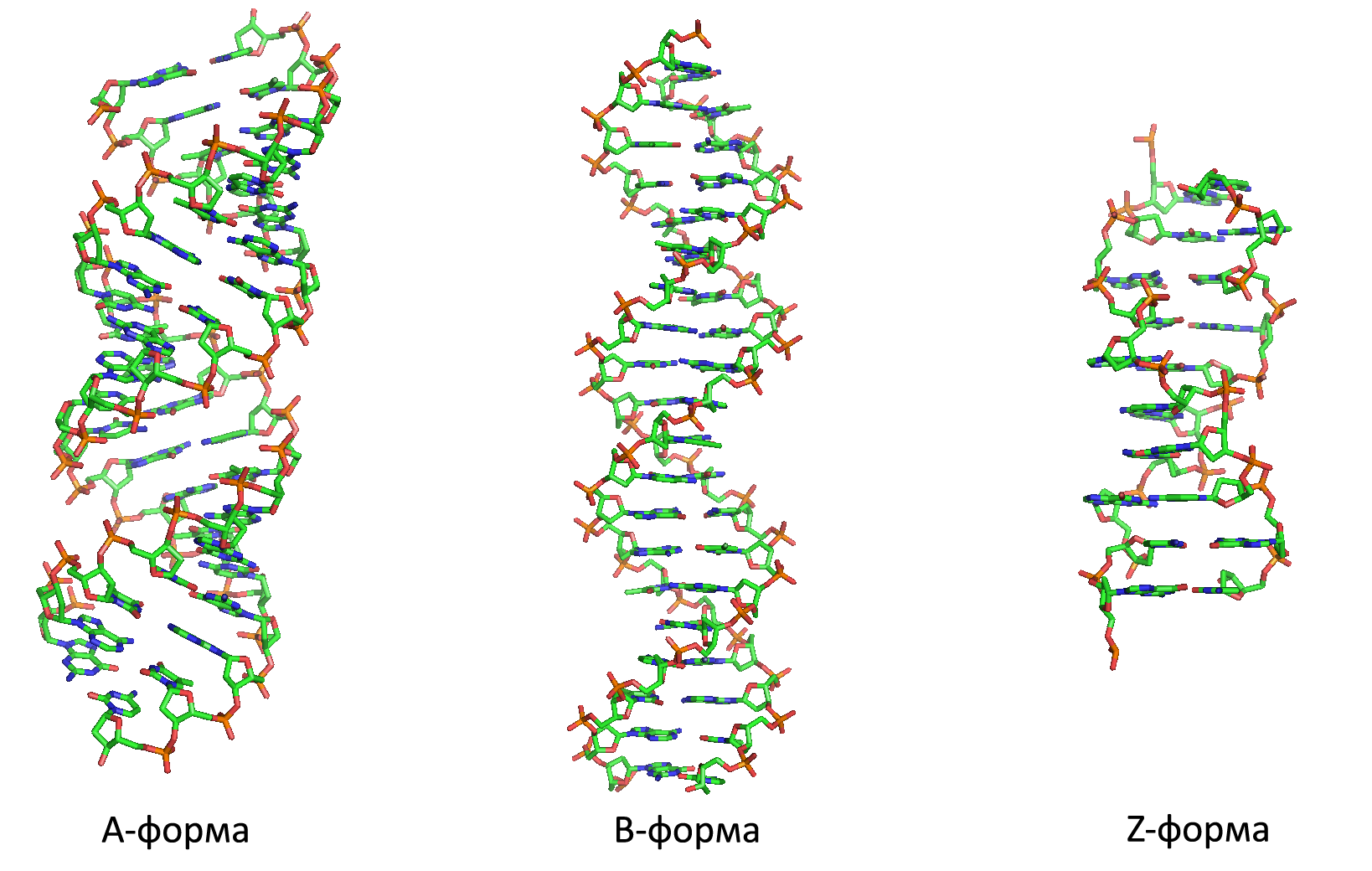
C использованием инструментов пакета 3DNA построим A-, B- и Z-форму дуплекса ДНК, последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность GATC. Структуры дуплексов сохраним соответственно в файлах gatc-a.pdb, gatc-b.pdb и gatc-z.pdb с помощью следующих команд:
fiber -a gatc-a.pdb
fiber -b gatc-b.pdb
fiber -z gatc-z.pdb
Созданные структуры показаны на изображении ниже (здесь и далее по нажатию на изображение доступен его просмотр в оригинальном размере):
Работа со структурами нуклеиновых кислот с помощью средств программы RasMol
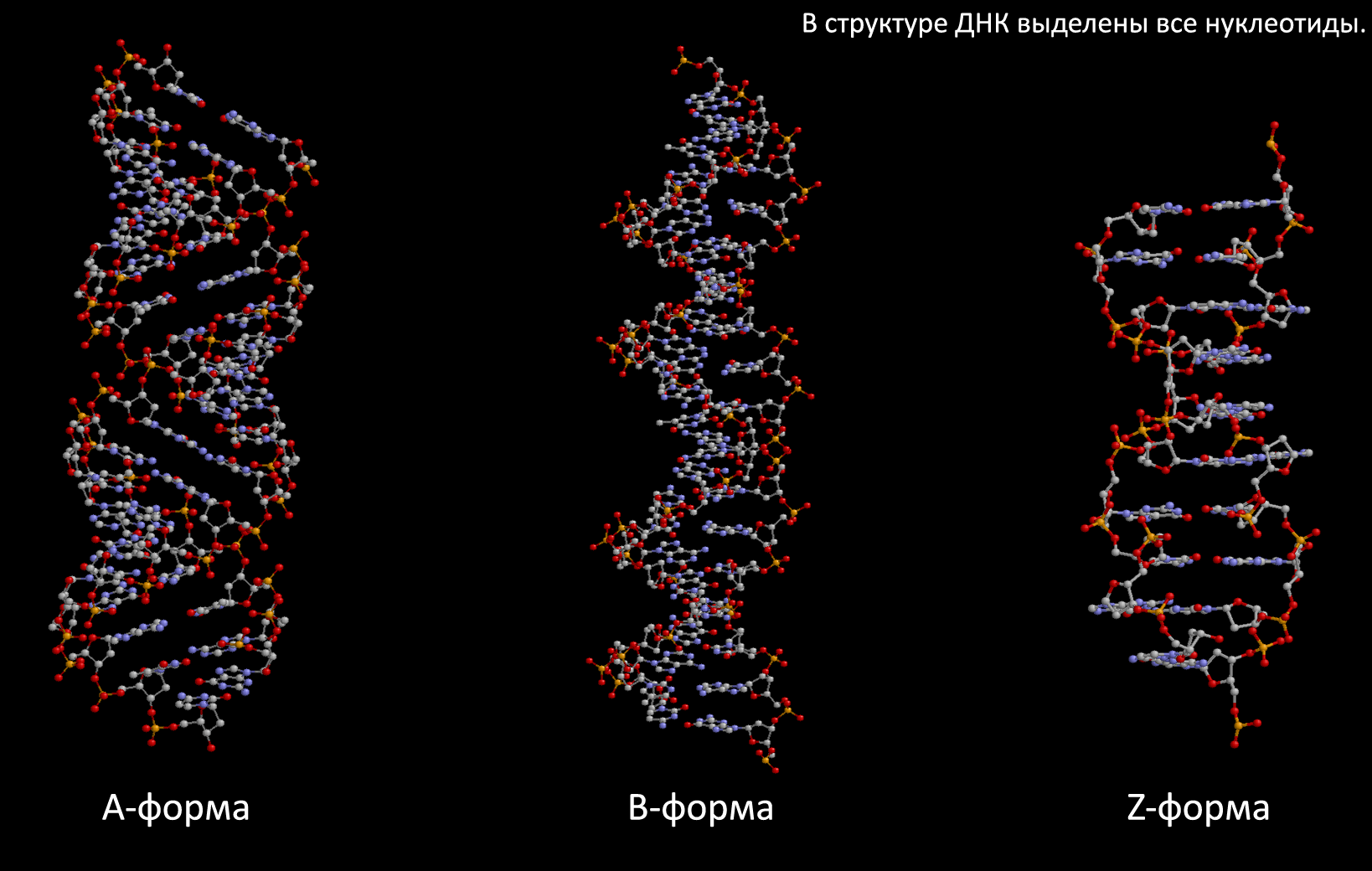
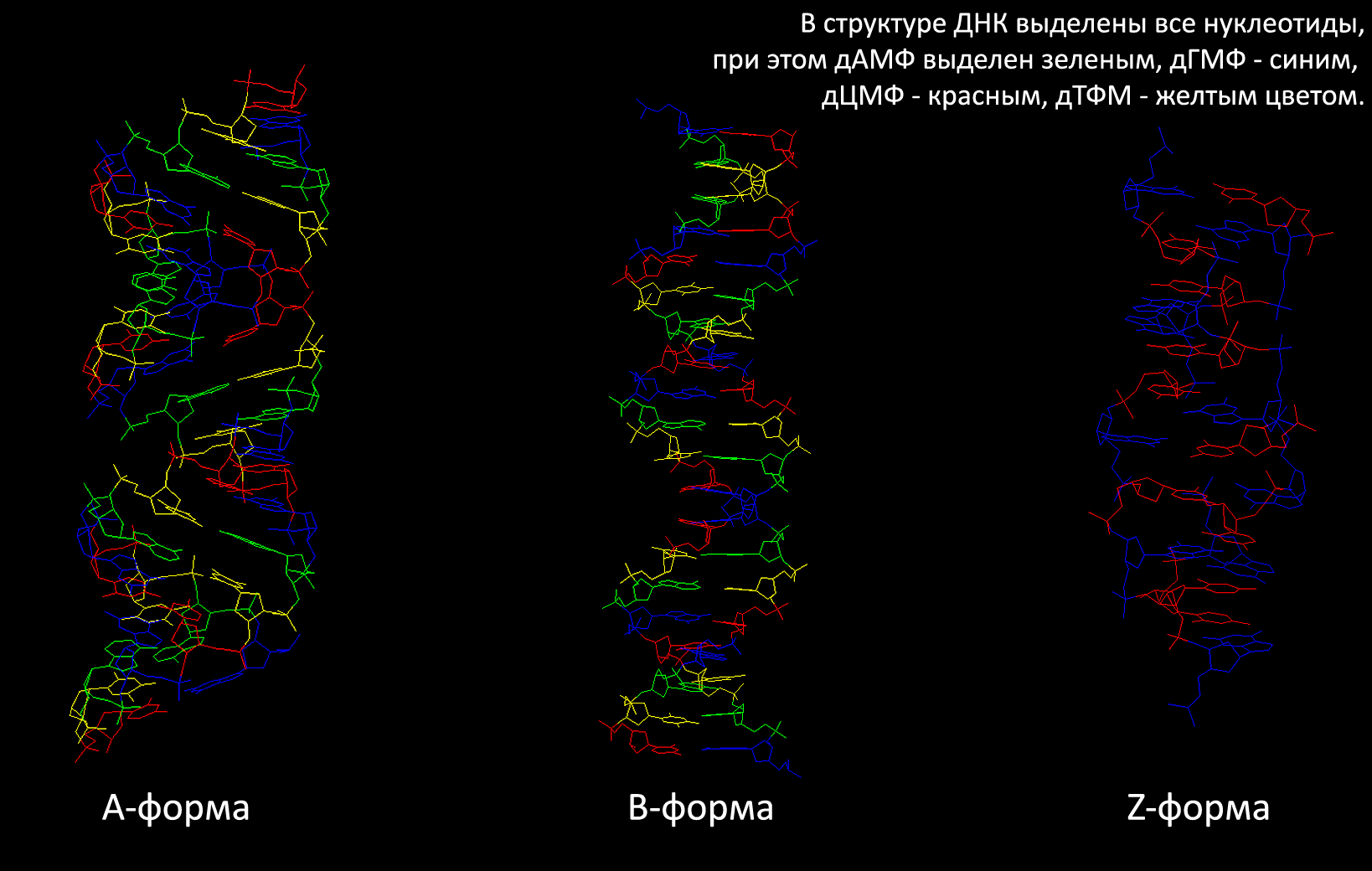
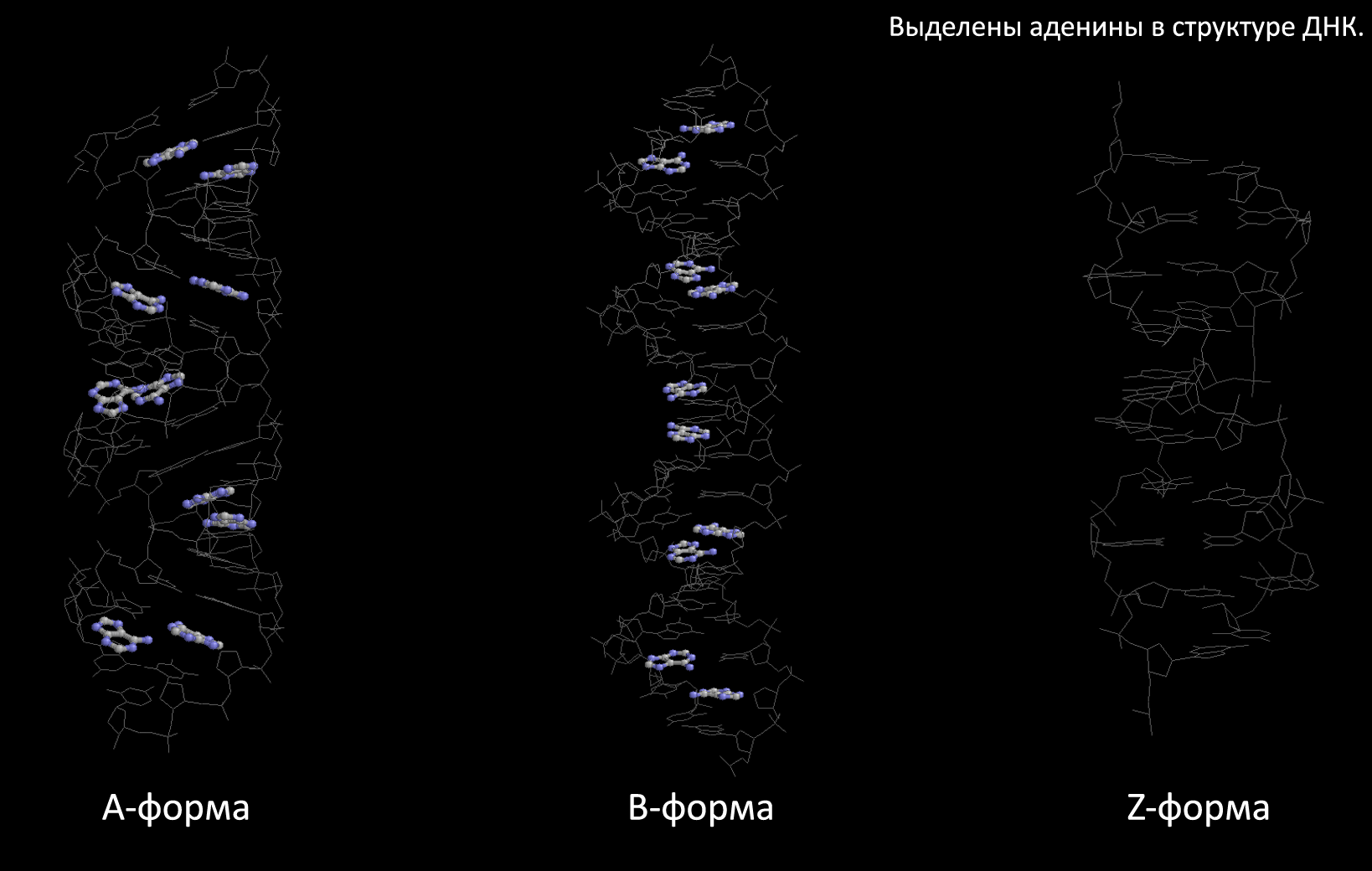
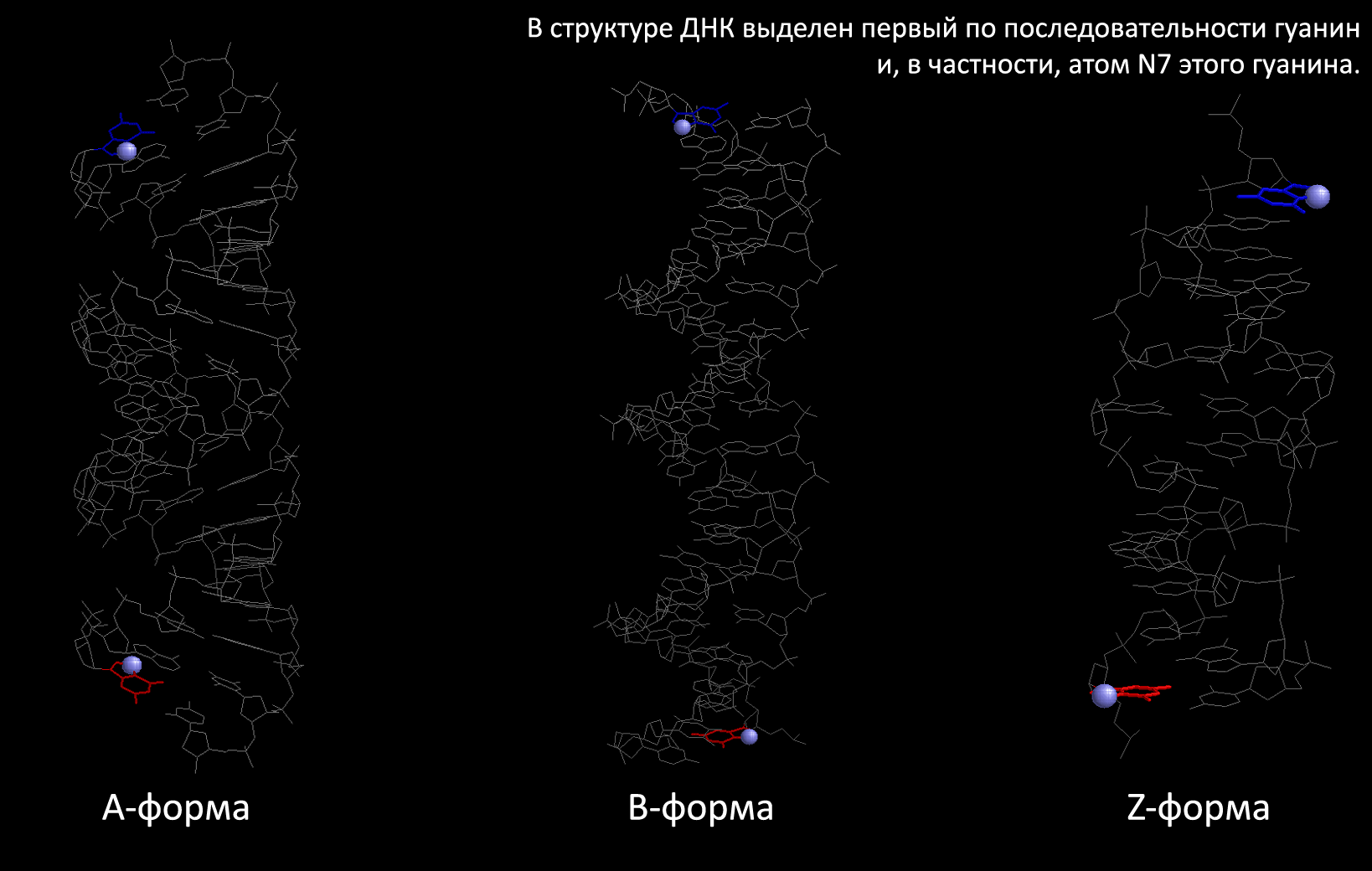
С помощью программы RasMol выделим в каждой из созданных структур дуплексов ДНК следующие атомы и химические группировки:
- сахарофосфатный остов ДНК;
- все нуклеотиды;
- все аденины;
- атом N7 во всех гуанинах;
- атом N7 в первом по последовательности гуанине.
(Т.к. нуклеотиды, по сути, составляют всю структуру ДНК, на изображении выше фактически выделена вся структура.)
(Как мы видим на изображении выше, в структуре Z-формы ДНК отсутствуют аденины. Это неудивительно, т.к. Z-форма ДНК содержит в своём составе лишь гуанины и цитозины.)
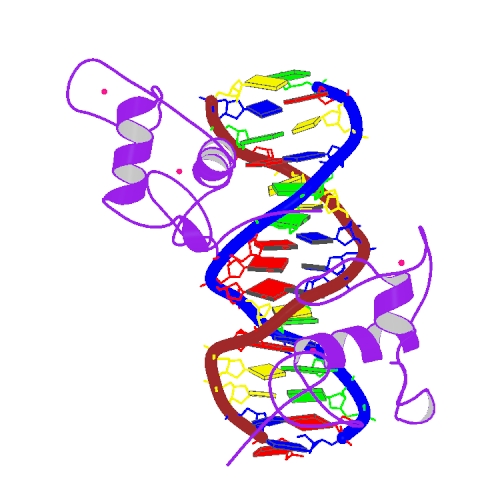
Получение файлов .pdb по идентификаторам 2dxi и 1by4
На сайте PDB выполним поиск по идентификаторам 2dxi и 1by4. Выберем подходящие варианты структур.
Выбранные варианты структур, представленные выше, сохранены в файлах 2dxi.pdb и 1by4.pdb соответственно.
Проверка заданных структур ДНК и РНК на наличие разрывов
Данные структуры можно рассмотреть в программе RasMol, изучив их на наличие разрывов.
В рассматриваемых структурах 2dxi и 1by4 разрывы отсутствуют. Ниже представлены изображения только нуклеиновой кислоты в проволочной модели для рассматриваемых структур.
В файлах 2dxi_rna_coords.txt и 1by4_dna_coords.txt сохранены координаты атомов только РНК и ДНК соответственно.
Сравнение структур различных форм ДНК с помощью средств RasMol
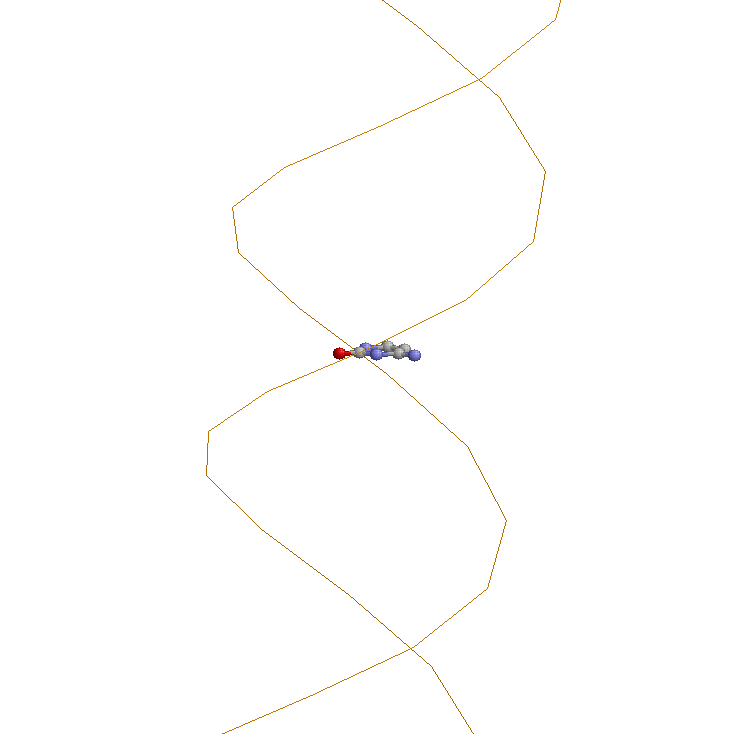
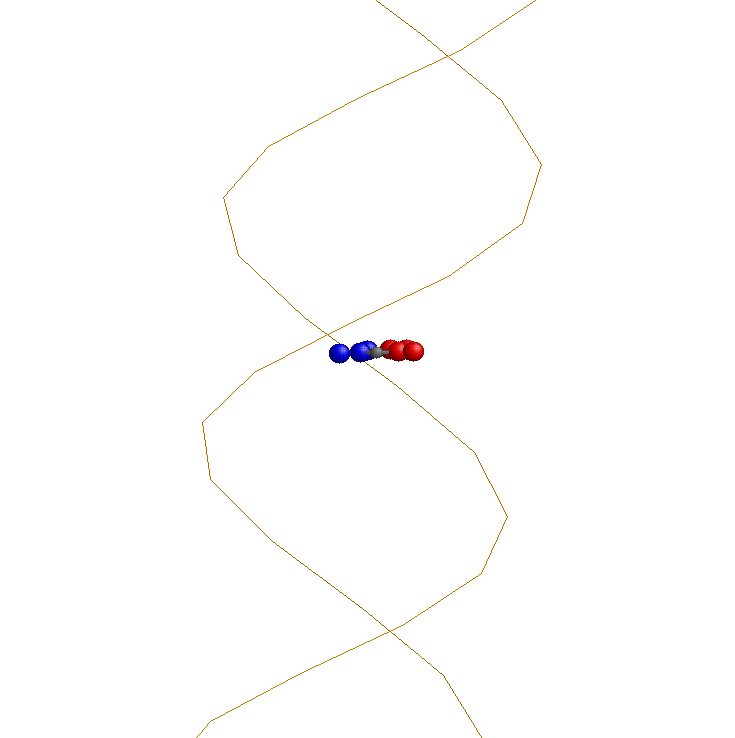
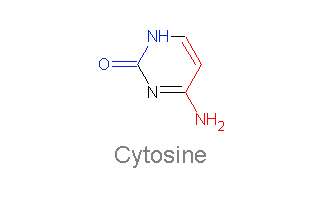
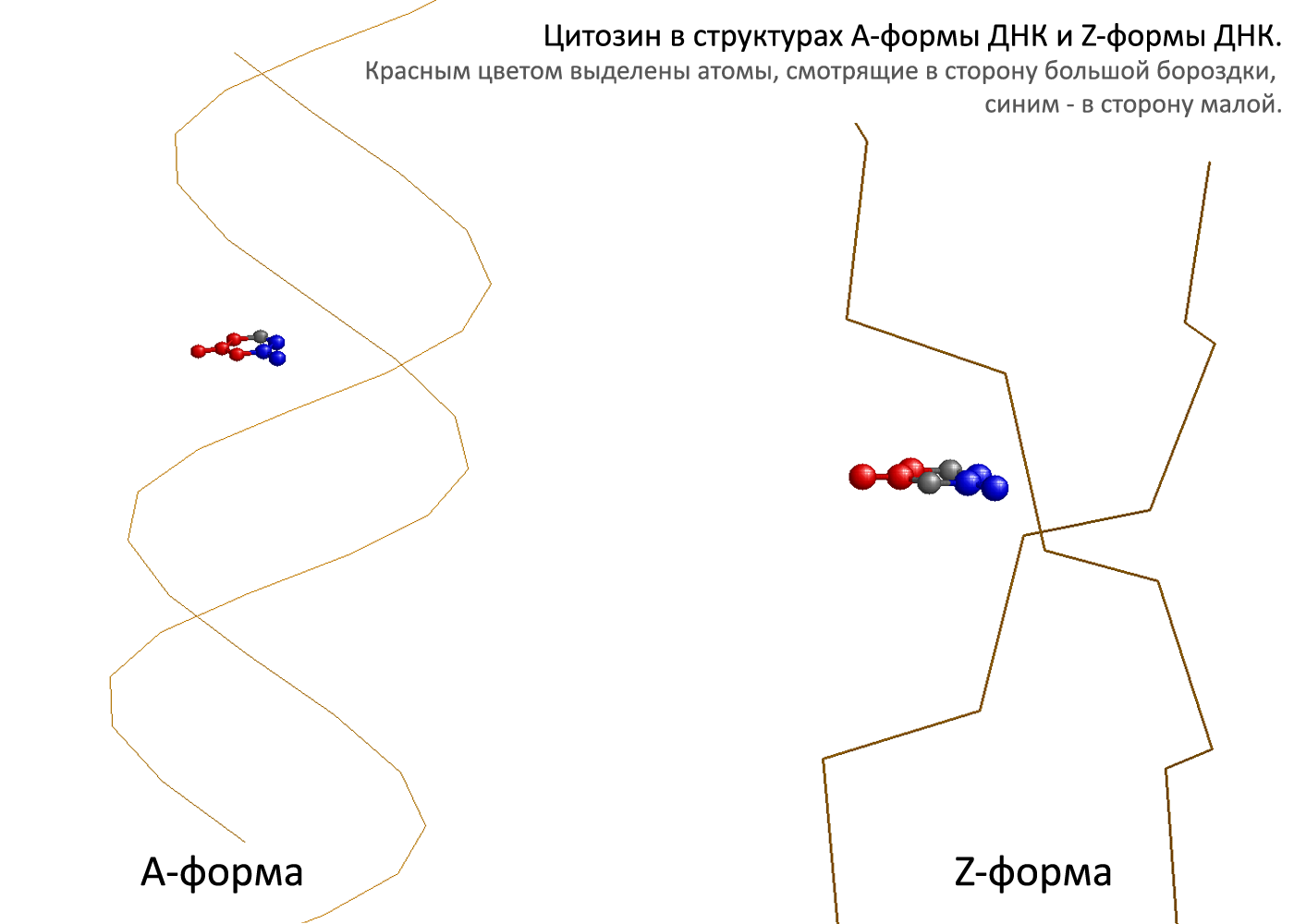
C помощью программы RasMol откроем файл gatc-b.pdb. Рассмотрите структуру B-формы ДНК и визуально определим большую и малую бороздку. Выберем в структуре цитозин, например, c12. Определим, какие атомы основания явно обращены в сторону большой бороздки, а какие в сторону малой:
С помощью программы ChemSketch получим изображение рассматриваемого основания. Выделим красным цветом атомы, смотрящие в сторону большой бороздки, синим - в сторону малой:
Аналогично рассмотрим цитозин в структуре A- и Z-форм ДНК:
| Форма ДНК | В сторону большой бороздки обращены атомы: | В сторону малой бороздки обращены атомы: | Остальные атомы основания: |
| A-форма | c12.n1, c12.c2, c12.o2, c12.n3 | c12.c5, c12.c4, c12.n4 | c12.c6 |
| B-форма | c12.c6, c12.c5, c12.c4, c12.n4 | c12.n1, c12.c2, c12.o2 | c12.n3 |
| Z-форма | c6.c5, c6.c4, c6.n4 | c6.n1, c6.c2, c6.o2 | c6.c6, c6.n3 |
Сравнение основных спиральных параметров разных форм ДНК
Результаты изучения структур из файлов gatc-a.pdb, gatc-b.pdb и gatc-z.pdb с помощью программы RasMol запишем в виде таблицы:
| A-форма | B-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Â) | 28.03 | 33.75 | 43.50 |
| Число оснований на виток | 11 | 10 | 12 |
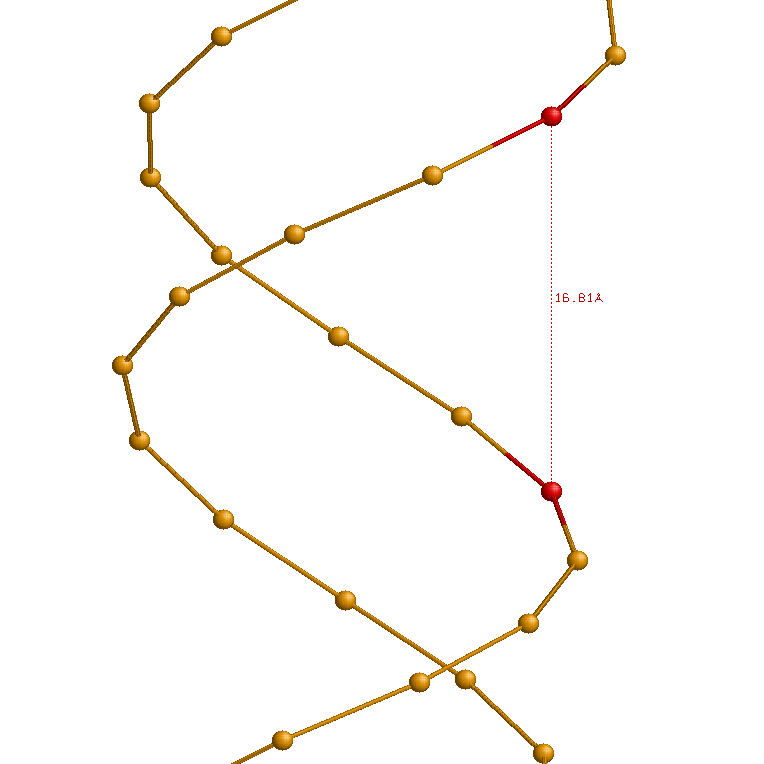
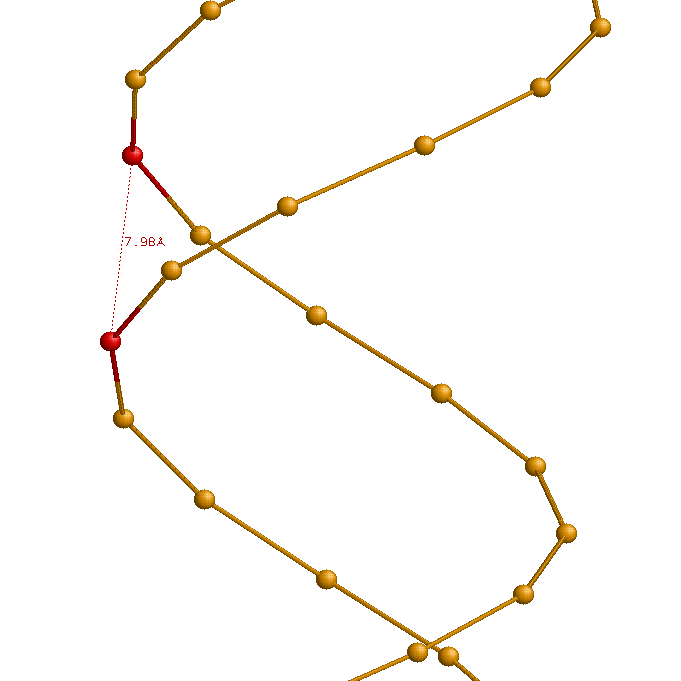
| Ширина большой бороздки (Â) | 16.81 (G13A.P - A30B.P) |
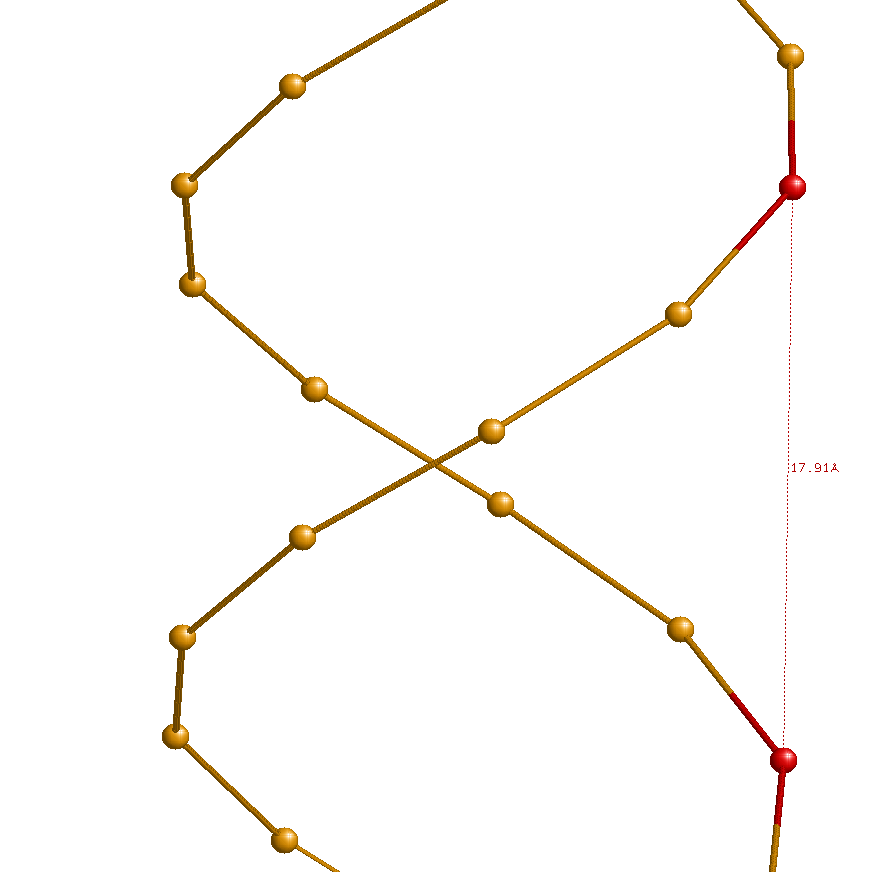
17.91 (A10A.P - T27B.P) |
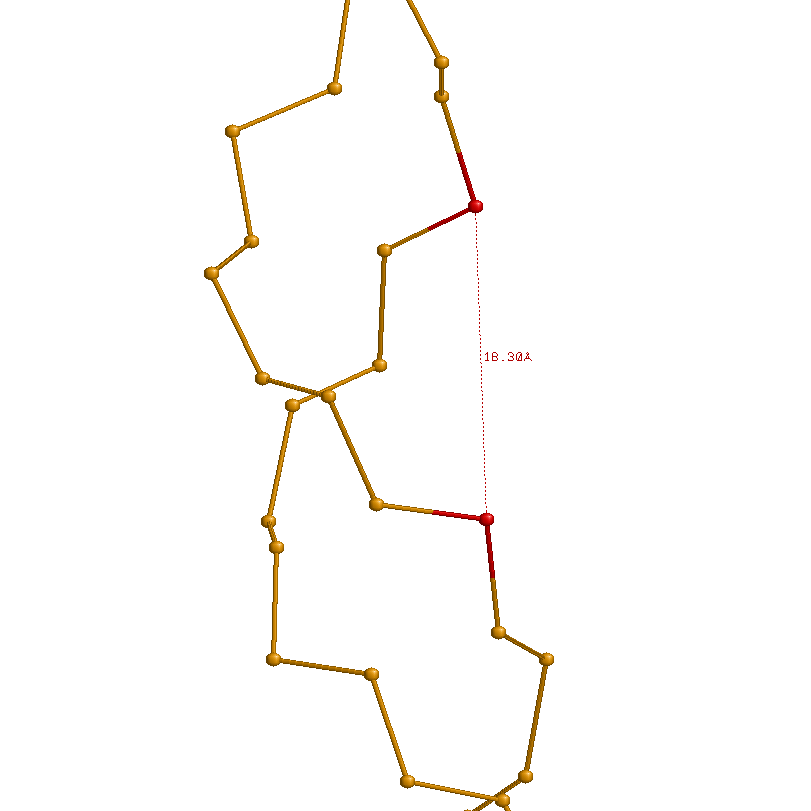
18.30 (G23A.P - G63B.P; см. файл gatc-z_20.pdb) |
| Ширина малой бороздки (Â) | 7.98 (G9A.P - A26B.P) |
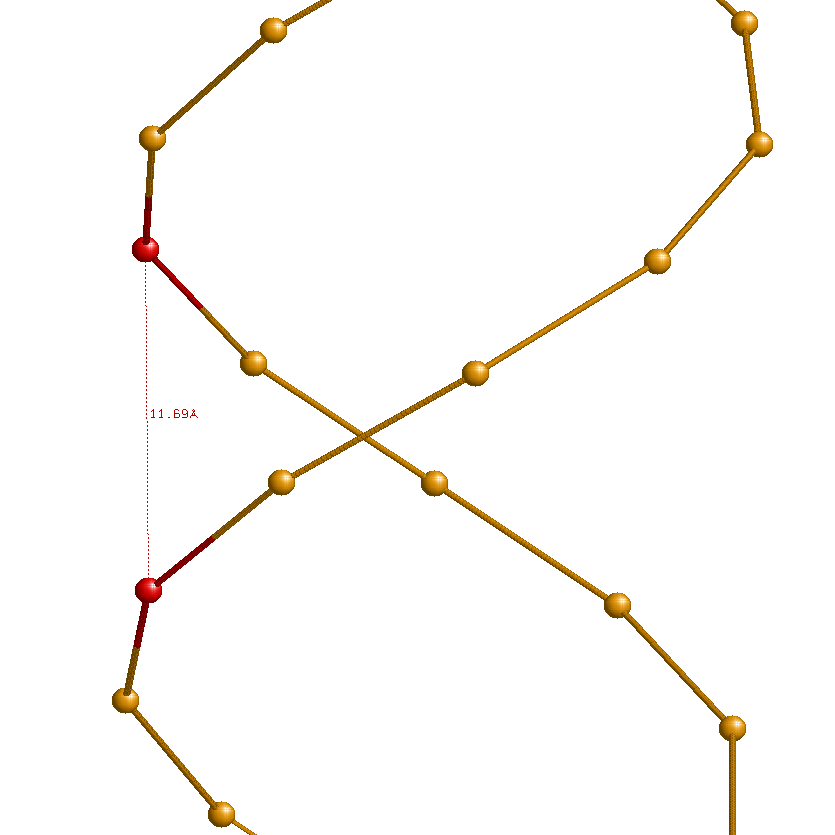
11.69 (A14A.P - T31B.P) |
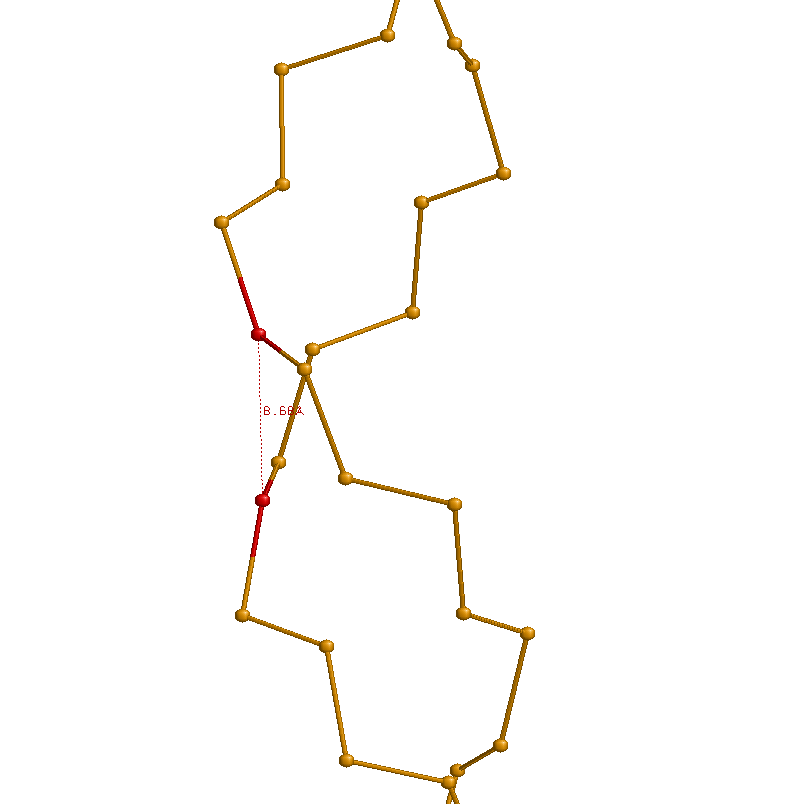
8.68 (C20A.P - C58B.P; см. файл gatc-z_20.pdb) |
Ниже приведены иллюстрации измерений ширины большой и малой бороздок в A-, B- и Z-формах ДНК:
Сравнение торсионных углов в структурах А- и В-форм ДНК
С помощью команды Settings -> Torsion в RasMol измерим торсионные углы цитозина. В таблице ниже приведены, кроме найденных величин, значения торсионных углов, данные для сравнения (см. в презентации).
| α (P - O5') | β (O5' - C5') | γ (C5' - C4') | δ (C4' - C3') | ε (C3' - O3') | ξ (O3' - P) | χ (C1' - N) | |
| A-ДНК | -51.67 | 174.79 | 41.73 | 79.11 | -147.75 | -75.15 | -157.18 |
| B-ДНК | -29.89 | 136.32 | 31.17 | 143.34 | -140.77 | -160.50 | -97.97 |
| Значения торсионных углов из презентации | |||||||
| A-ДНК | -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| B-ДНК | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA
Для каждого из файлов gatc-a.pdb, gatc-b.pdb и gatc-z.pdb была выполнена следующая команда:
find_pair -t gatc-x.pdb stdout | analyze
В результате для каждой структуры создан ряд файлов с описанием разных её параметров. Например, в файлах gatc-a.out, gatc-b.out и gatc-z.out можно найти значения торсионных углов. Для удобства значения торсионных углов для рассматриваемых структур приведены в файле gatc_torsion_angles.txt.
Если сравнить значения соответствующих торсионных углов в структурах A-, B- и Z-форм ДНК, то в наибольшей степени различаются значения следующих углов: у A- и B-форм - δ и χ; у A- и Z-форм - α; у A- и Z-форм - α, ξ и χ. (Впрочем, сравнения с торсионными углами Z-формы ДНК довольно условны.)
Так как пакет 3DNA работает только со старым форматом .pdb, используем программу remediator для перевода файлов 2dxi.pdb и 1by4.pdb в старый формат:
remediator --old "XXXX.pdb" > "XXXX_old.pdb"
Затем применим команды find_pair и analyze (см. выше).
Значения торсионных углов в структуре тРНК (2dxi_old.pdb) для удобства приведены в файле 2dxi_torsion_angles.txt. Данная структура больше всего похожа на A-форму ДНК, если рассматривать значения торсионных углов.
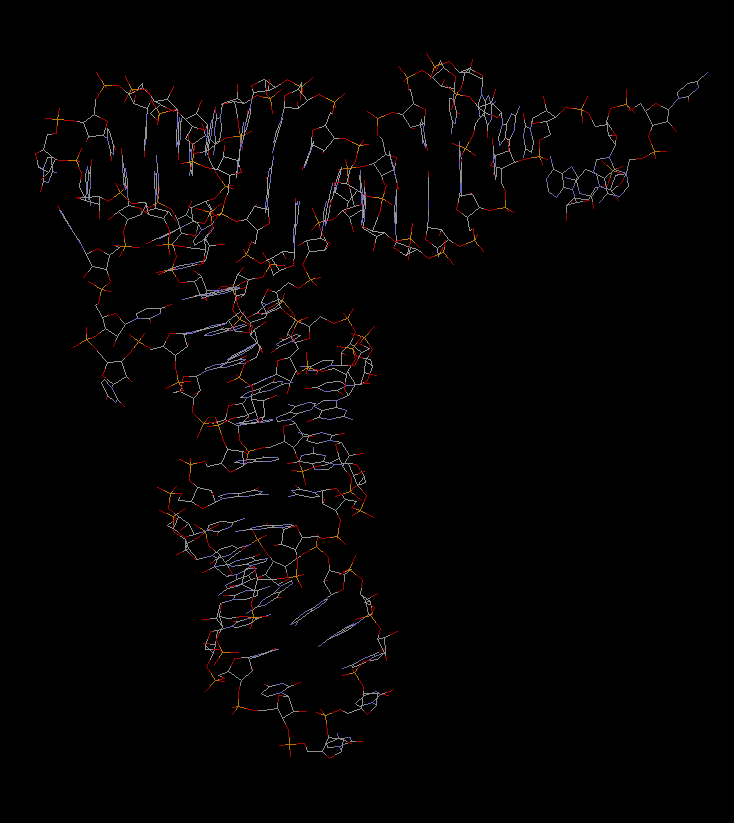
Исследование структуры тРНК
I. Краткое описание структуры в файле 2dxi.pdb
В файле приведены координаты атомов следующих молекул:
- тРНК (Synthetic construct, 1 молекула, только C-цепь);
- глутамил-тРНК синтетаза (Thermus thermophilus, 1 молекула, только A-цепь).
Для исследования была выбрана цепь C, представляющая тРНК (Glu) со следующей последовательностью:
[501] 5' - GGCCCCAUCG UCUAGCGGUU AGGACGCGGC CCUCUCAAGG CCGAAACGGG GGUUCGAUUC CCCCUGGGGU CACCA - 3' [576]
В записи выше 501 и 576 - номера первого и последнего нуклеотидов.
Как мы видим, на 3'-конце последовательности есть триплет CCA, к которому присоединяется аминокислота. Координаты его атомов приведены (их можно найти в файле 2dxi_rna_coords.txt, в котором приведены координаты атомов РНК).
II. Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями (см. файл 2dxi_hbonds.txt или файл 2dxi_old.out). В соответствии с полученными данными:
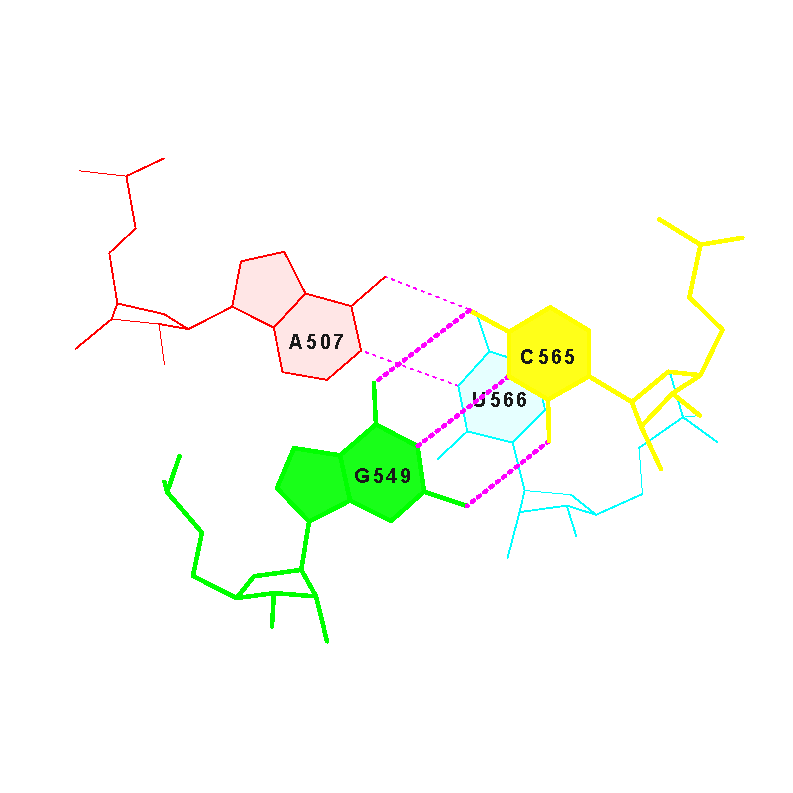
- акцепторный стебель состоит из участка 501 - 507 и комплементарного ему участка 566 - 572;
- Т-стебель состоит из участка 549 - 553 и комплементарного ему участка 561 - 565;
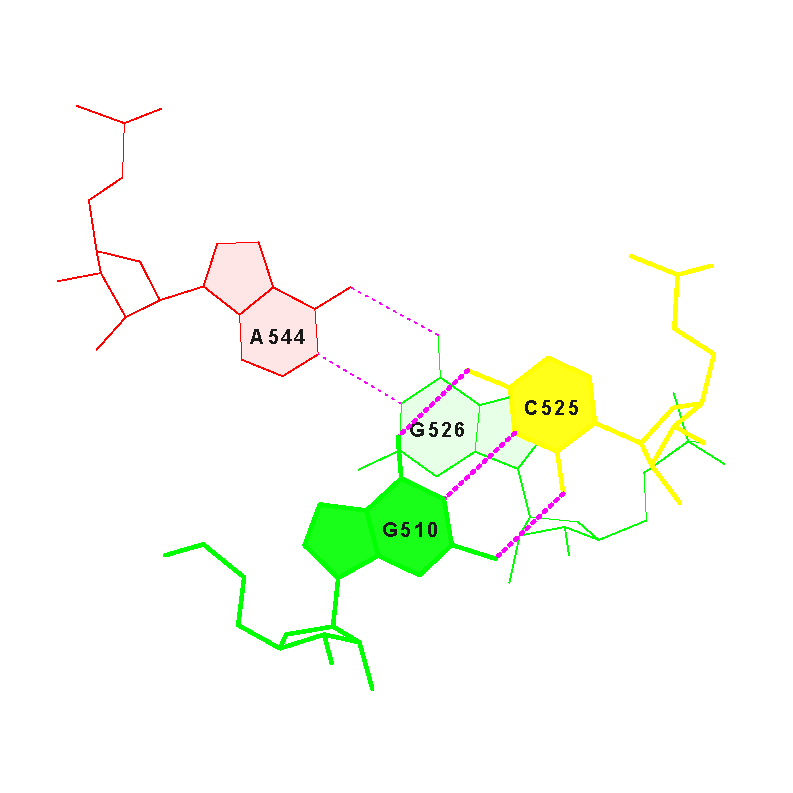
- D-стебель состоит из участка 510 - 513 и комплементарного ему участка 522 - 525;
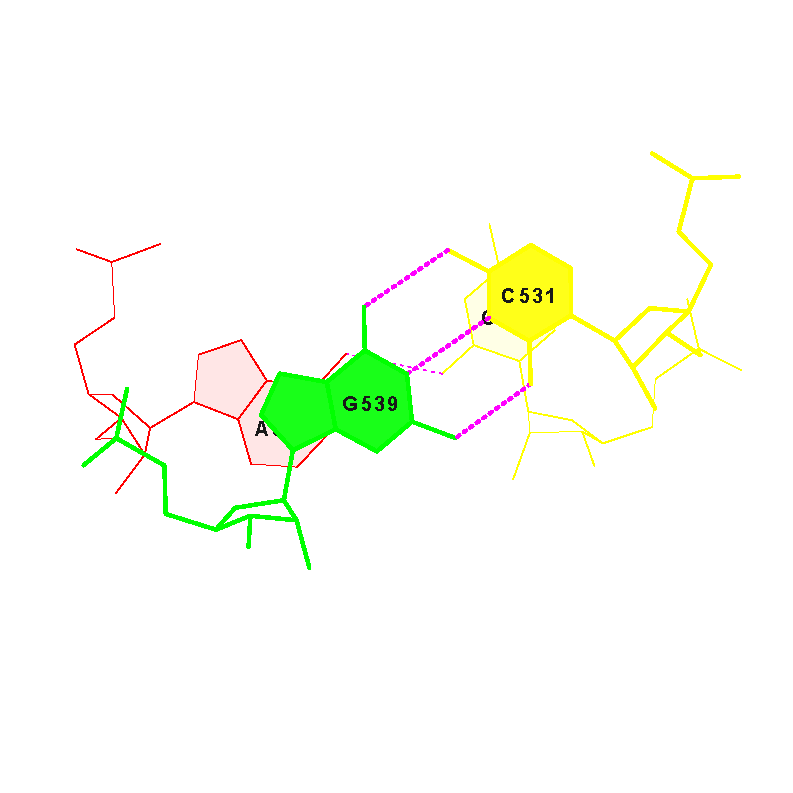
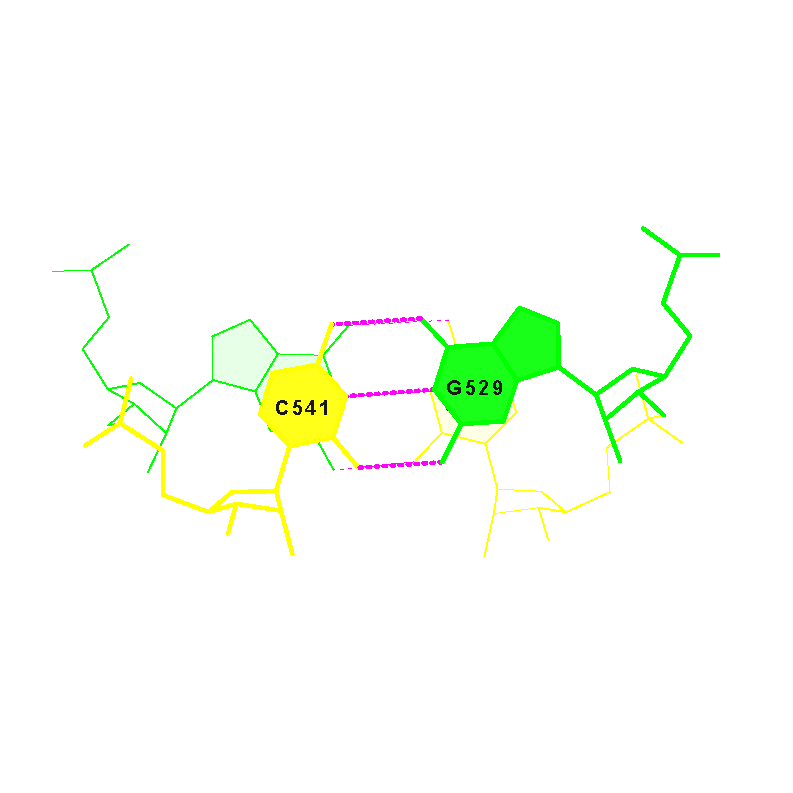
- антикодоновый стебель состоит из участка 526 - 532 и комплементарного ему участка 538 - 544.
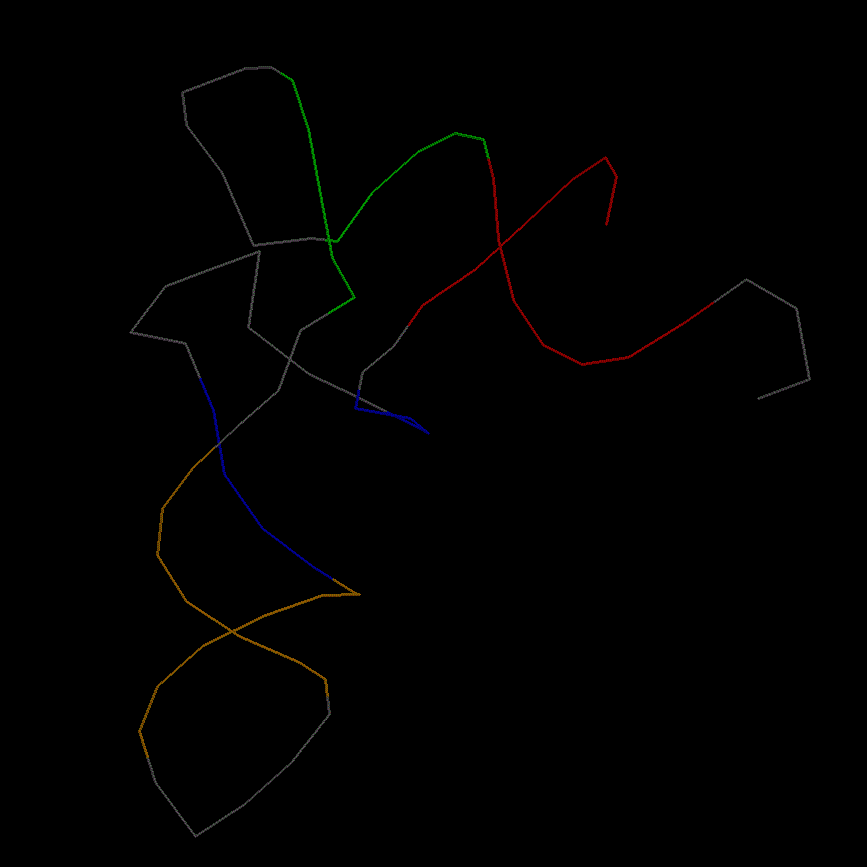
С помощью программы RasMol получим изображение остова исследуемой тРНК, где акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый стебель - оранжевым:
|
load 2dxi.pdb restrict rna wireframe off backbone 20 color gray select g501, g502, c503, c504, c505, c506, a507, u566, g567, g568, g569, g570, u571, c572 define acceptor selected color red select g549, g550, g551, g552, g553, c561, c562, c563, c564, c565 define tstrain selected color green select g510, u511, c512, u513, g522, g523, a524, c525 define dstrain selected color blue select g526, c527, g528, g529, c530, c531, c532, a538, g539, g540, c541, c542, g543, a544 define anticodon selected color orange |
(В скрипте выше подробное перечисление остатков (вместо команд вида «select 501-507 or 566-572») приведено исключительно с целью наглядности.)
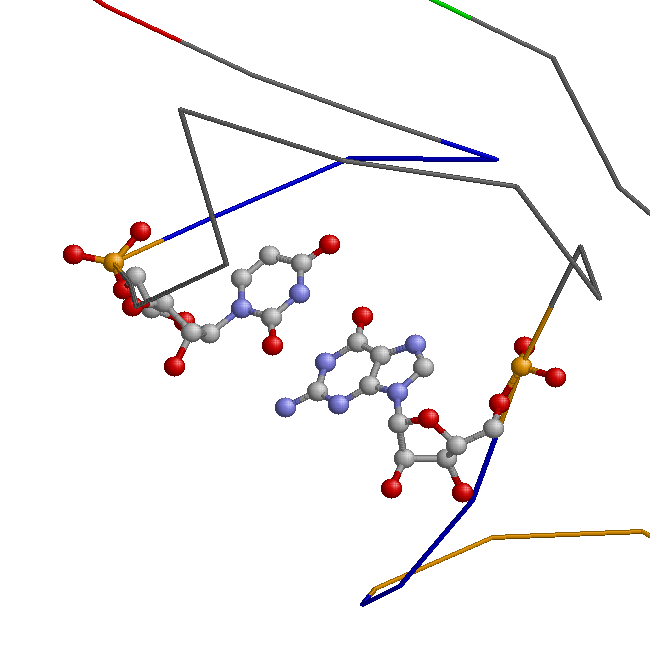
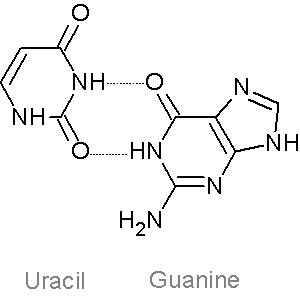
Структуру стеблевых дуплексов поддерживают 19 канонических пар оснований и 4 неканонические пары (G502-U571, U513-G522, G526-A544, C532-A538). Пример неканонической пары оснований (U513-G522) приведён на изображениях ниже.
[501] 5' - GGCCCCAUCGUCUAGCGGUUAGGACGCGGCCCUCUCAAGGCCGAAACGGGGG UUCGAUUCCCCCUGGGGUCACCA - 3' [576]
- Вариабельная петля присутствует и состоит всего из 3 нуклеотидов: A545, A546 и C548. Данные нуклеотиды выделены в последовательности выше более крупным шрифтом.
- В T-петле остаток тимидин отсутствует.
- Дигидроуридины в D-петле отсутствуют.
- Антикодон в антикодоновой петле - CUC (C534, U535, C536). На изображении ниже указанные нуклеотиды представлены в шарнирной модели:
III. Исследование третичной структуры
Исследуем возможность стекинг-взаимодействий.
В файле 2dxi_old.out с характеристикой структуры тРНК указаны данные о величине площади "перекрывания" двух последовательных пар азотистых оснований. Для пар с наибольшими значениями получим стандартное изображение стекинг-взаимодействия.
В файле stacking.pdb записаны преобразованные координаты всех динуклеотидных пар. С помощью следующих команд вырежем структуры, описывающие координаты искомых нуклеотидных остатков, между которыми предполагается стекинг-взаимодействие:
ex_str -2 stacking.pdb step2.pdb
ex_str -13 stacking.pdb step13.pdb
ex_str -16 stacking.pdb step16.pdb
ex_str -18 stacking.pdb step18.pdb
Построить изображения можно с помощью следующих команд:
stack2img -cdolt step2.pdb step2.ps
stack2img -cdolt step13.pdb step13.ps
stack2img -cdolt step16.pdb step16.ps
stack2img -cdolt step18.pdb step18.ps
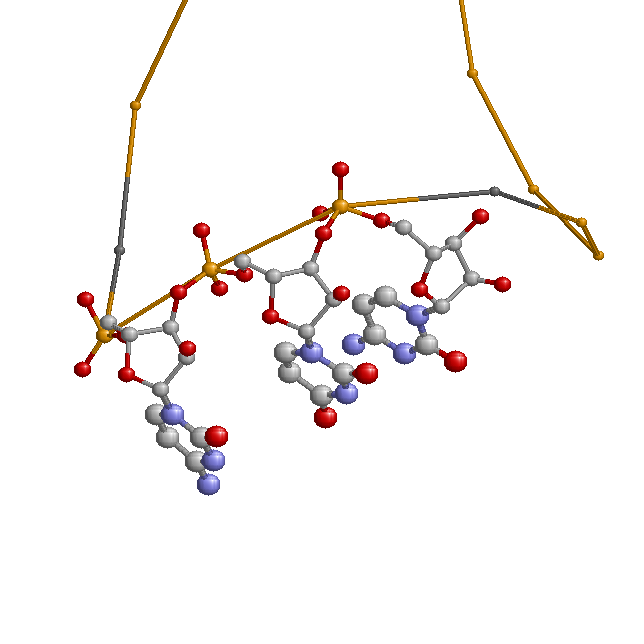
Теперь исследуем возможность стекинг-взаимодействия между основаниями конца акцепторного стебля и начала Т-стебля, а также между антикодоновым стеблем и D-стеблем. Величина площади "перекрывания" последовательных пар оснований невелика (см. файл 2dxi_old.out): 1.94 Â2 для AG/CU (Section#0007) и 2.20 Â2 для AG/CG (Section#0022). Аналогичным рассмотренному выше способу создадим следующие изображения, иллюстрирующие наложение оснований:
Как мы видим, "перекрывание" последовательных пар оснований очень мало. В данном случае говорить о сильном стекинг-взаимодействии не приходится.
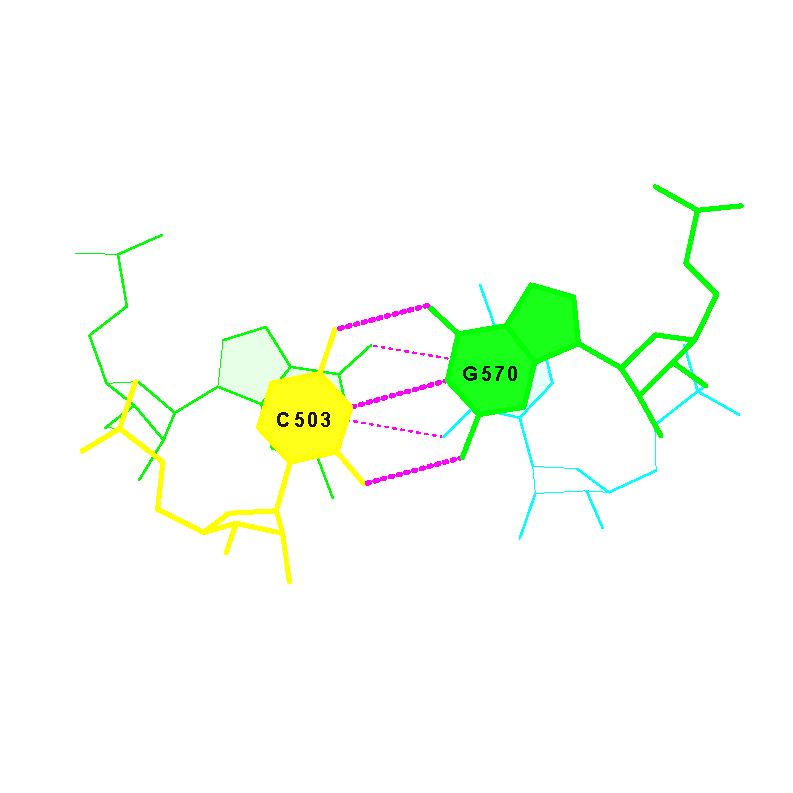
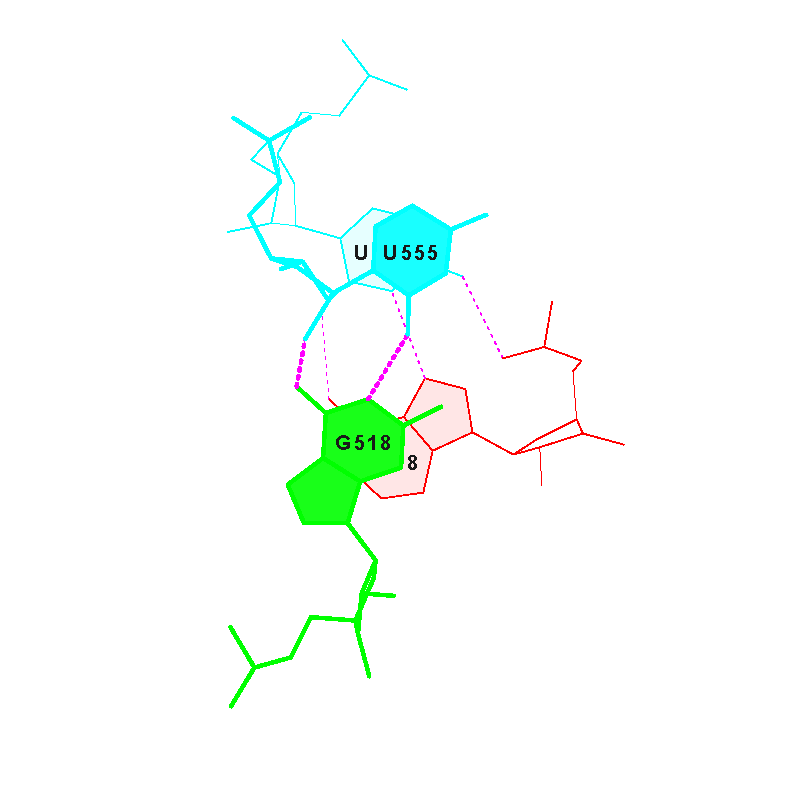
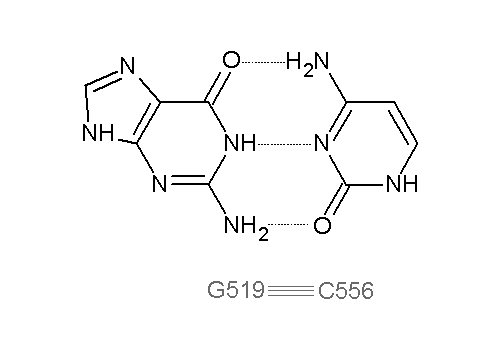
Между основаниями D- и Т-петель можно обнаружить (см. файл 2dxi_hbonds.txt) дополнительные водородные связи: G518 - U555, G519 - C556 (первая пара - неканоническая, вторая - каноническая). Имеется таже дополнительная водородная связь U520 - C548, стабилизирующая ее третичную структуру, однако C548 не расположен в T-петле. Ниже приведено изображение, иллюстрирующее водородные связи (показаны пунктирной линией) в паре G519 - C556:
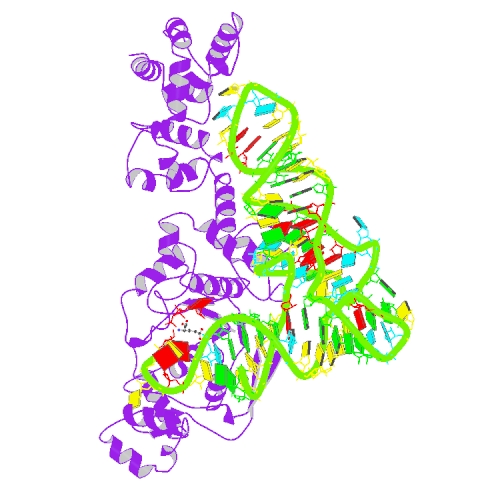
Исследование ДНК-белковых взаимодействий в структуре комплекса белка RXR и фрагмента ДНК
I. Краткое описание структуры в файле 1by4.pdb
В файле 1by4.pdb приведены координаты атомов молекулы белка RXR-alpha (рецептор ретиноевой кислоты; из homo sapiens; цепи A и B) и молекулы ДНК (synthetic, цепи E и F). Для исследования были выбраны цепи A и B белка и цепи E и F, представляющие ДНК со следующей последовательностью:
цепь E [1494] 5' - CTAGGTCAAAGGTCAG - 3' [1509] ||||||||||||||| цепь F [1531] 3' - ATCCAGTTTCCAGTC - 5' [1545]
II. Функции белка, структура которого представлена в файле 1by4.pdb
Белок рецептор ретиноевой кислоты RXR-alpha (UniProtKB: P19793) связывается с ответными элементами ретиноевой кислоты (RARE). В отсутствие лиганда RXR-RAR гетеродимеры связываются с мультибелковым комплексом, содержащим транскрипционные корепрессоры (ингибиторы экспрессии генов), которые вызывают ацетилирование гистонов, конденсацию хроматина и подавление транскрипции. При связывании лигандов корепрессоры отделяются от рецепторов и связываются с коактиваторами, ведущими к активации транскрипции. Данный белок имеет высокий уровень экспрессии в печени, найден также в легких, почках и сердце. Рецепторы ретиноевой кислоты являются регуляторами генетической экспрессии в различных биологических процессах.
III. Исследование структуры ДНК
Воспользуемся программами find_pair и analyze для файла 1by4_old.pdb:
find_pair -t 1by4_old.pdb stdout | analyze
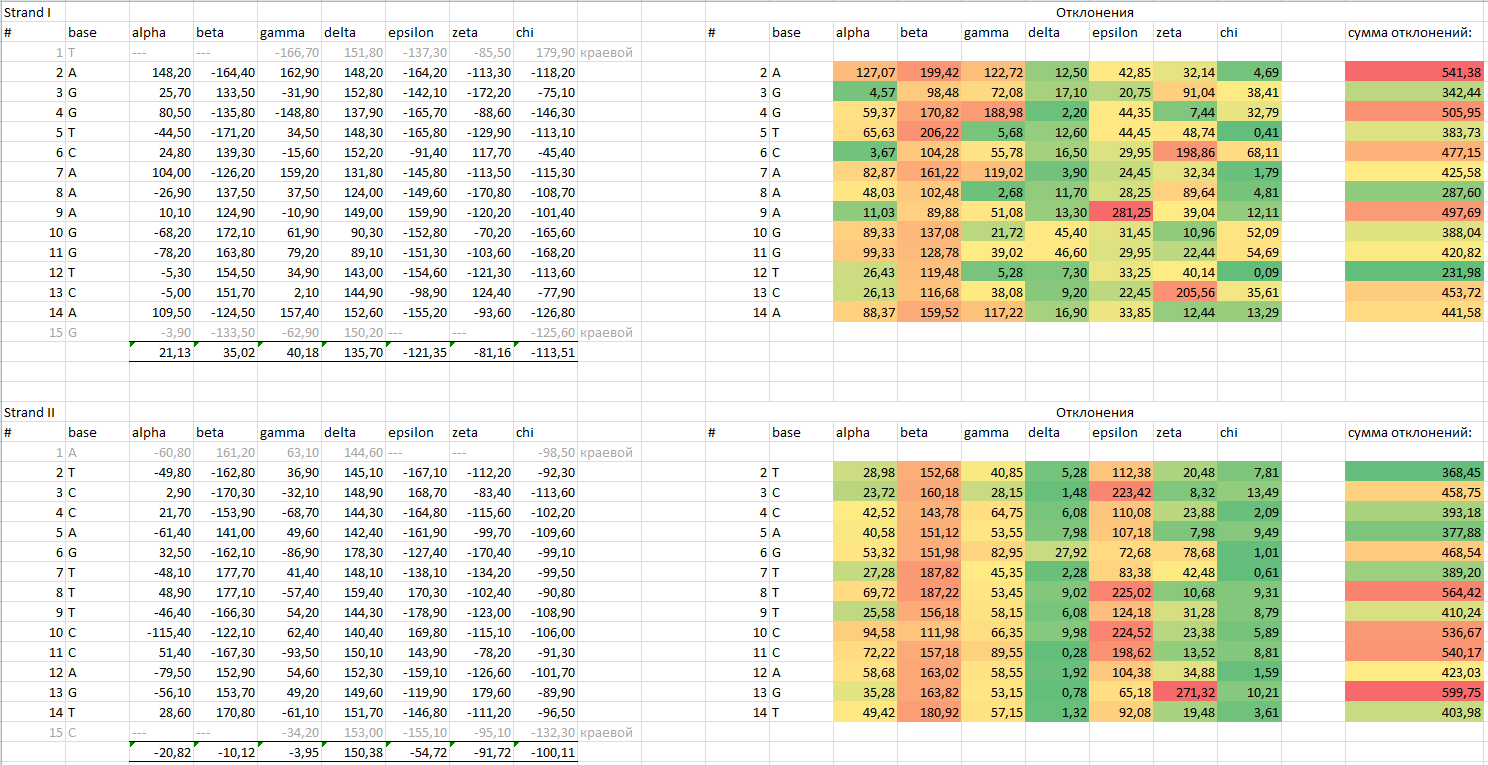
Файл 1by4_old.out содержит результаты анализа структуры, в том числе значения торсионных углов для нуклеотидов. Данная ДНК представляет собой B-форму ДНК. Для удобства значения торсионных углов рассматриваемой структуры приведены в файле 1by4_torsion_angles.txt. На основе анализа данных в программе Excel (см. файл файл 1by4_torsion.xlsx) сделаем следующие выводы:
- Наиболее "деформированным" нуклеотидом можно назвать нуклеотид G1533 (нуклеотид G, имеющий номер 13 во второй цепи).
- Как мы видим, связывание ДНК с белком приводит к деформациям в ДНК, в частности, мы можем наблюдать значительные отклонения в значениях торсионных углов изучаемой структуры по сравнению со свободной молекулой B-ДНК.
Ссылки
- Файл gatc-a.pdb.
- Файл gatc-b.pdb.
- Файл gatc-z.pdb.
- Файл gatc-a.out.
- Файл gatc-b.out.
- Файл gatc-z.out.
- Файл gatc_torsion_angles.txt.
- Файл 2dxi.pdb.
- Файл 2dxi_rna_coords.txt.
- Файл 2dxi_torsion_angles.txt.
- Файл 2dxi_old.pdb.
- Файл 2dxi_hbonds.txt.
- Файл stacking.pdb (для тРНК).
- Файл 1by4.pdb.
- Файл 1by4_dna_coords.txt.
- Файл 1by4_old.pdb.
- Файл 1by4_torsion_angles.txt.
- Файл 1by4_torsion.xlsx.