Занятие 7: Программа getorf. Поиск некодирующих последовательностей
Работа с программой getorf пакета EMBOSS
Для начала работы загрузим файл с записью D89965 банка EMBL, например, с помощью команды entret:
entret embl:d89965
В итоге получим файл d89965.entret. Затем, чтобы получить набор трансляций всех открытых рамок данной последовательности длиной более 30 нуклеотидов, считая открытой рамкой последовательность триплетов от старт-кодона до стоп-кодона, при использовании стандартного кода, выполним следующую команду:
getorf d89965.entret d89965.orf -find 1
В результате получим файл файл d89965.orf, содержащий 9 найденных открытых рамок. Если мы сравним найденные рамки с кодирующими последовательностями, приведёнными в записи D89965 на EMBL, то заметим, что пятая найденная открытая рамка (163 - 432) частично соответствует кодирующей последовательности с координатами 163 - 435.
Данная запись EMBL ссылается на запись P0A7B8 в Swiss-Prot. Загрузим последовательность этой записи с помощью следующей команды:
seqret sw:p0a7b8
Теперь выясним, какой из найденных ранее открытых рамок соответствует полученная последовательность (см. файл hslv_ecoli.fasta). Для этого можно воспользоваться программой blastp с соответствующими параметрами:
blastp -query hslv_ecoli.fasta -subject d89965.orf -out blastp.out
Исходя из информации в файле blastp.out, мы можем сказать, что последовательность записи P0A7B8 соответствует девятой найденной нами открытой рамке (294 - 1).
Заметим, что запись D89965 банка EMBL содержит последовательность мРНК для серой крысы, в то время как запись P0A7B8 банка Swiss-Prot, на которую она ссылается, содержит последовательность АТФ-зависимой субъединицы протеазы HslV кишечной палочки. Также обратим внимание на то, что в информации о кодирующей последовательности в записи D89965 указано: Rat Stomach Serotonin receptor-related gene. Поэтому вполне вероятно, что данное несоответствие возникло из-за использования крысы, заражённой кишечной палочкой, в работе по секвенированию.
Поиск гомологов некодирующих последовательностей программой BLASTN
Воспользуемся программой blastn, указав в качестве последовательностей для поиска файле trna_bacsu.fasta, в качестве банка - геном бактерии Streptococcus agalactiae; при этом установим табличный формат выдачи и порог на E-value 0.01:
blastn -query trna_bacsu.fasta -db sa -out trna_bacsu_sa_blastn.txt -evalue 0.01 -outfmt 6 -task blastn
Для подсчёта количества находок для каждой последовательности в файле trna_bacsu_sa_blastn.txt сначала необходимо составить список названий входных последовательностей. Выполним команду:
grep ">" trna_bacsu.fasta > trnas.txt
Полученный в файле trnas.txt список названий последовательностей импортируем в Excel. С помощью функции CONCATENATE (СЦЕПИТЬ) создадим строки для скрипта count_trnas_in_sa.scr следующего вида:
grep -c 't2xxxx' trna_bacsu_sa_blastn.txt >> trnas_in_sa_count.txt
Затем сделаем созданный файл со скриптом исполняемым и запустим его:
chmod +x count_trnas_in_sa.scr
./count_trnas_in_sa.scr
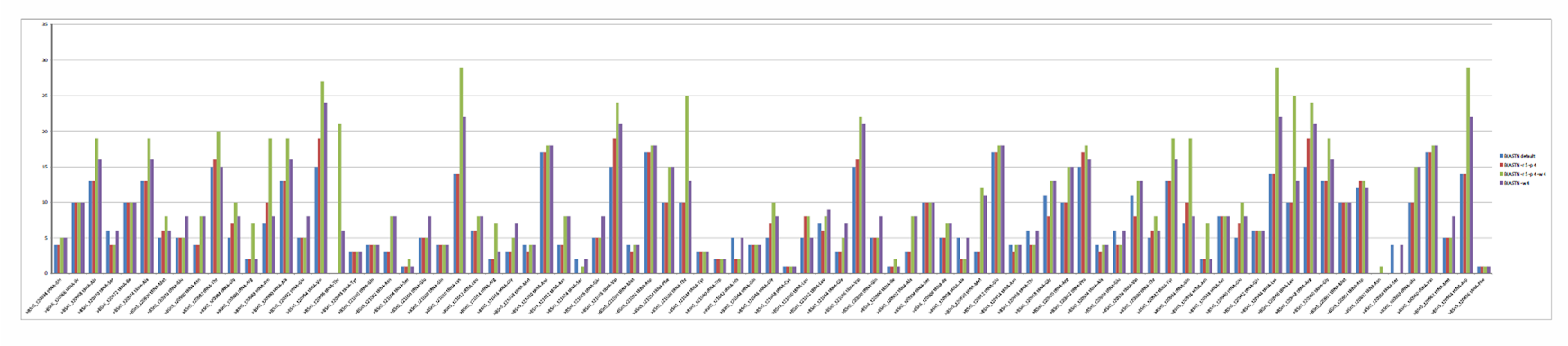
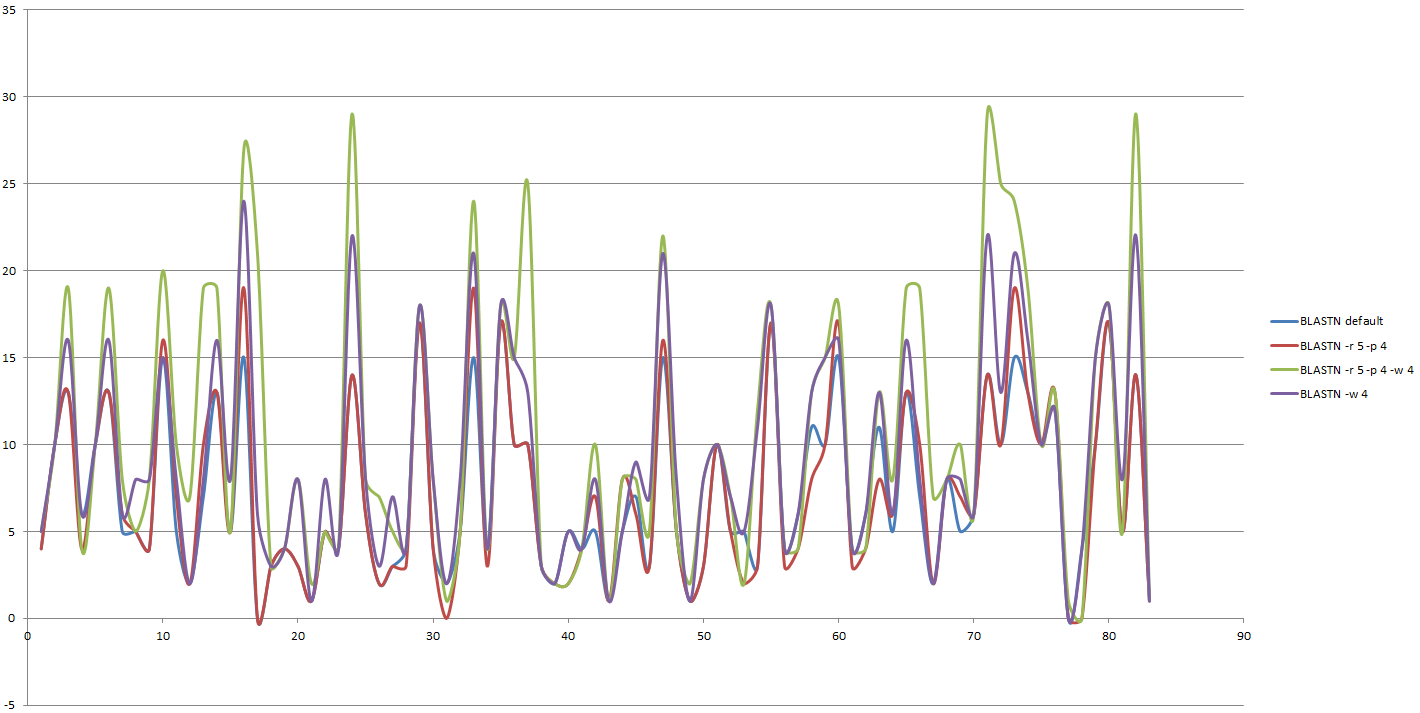
В результате в файле trnas_in_sa_count.txt мы получим столбец со значениями числа находок для каждой последовательности. Импортируем полученные данные в Excel (см. столбец BLASTN default в файле trna.xlsx).
Поиск гомологов при изменённых параметрах программы BLASTN
Повторим поиск гомологов с изменёнными значениями параметров программы blastn. Сначала изменим весовую матрицу: установим параметры -reward 5 и -penalty -4. Из предложенных программой изменений значений параметров -gapopen и -gapextent выберем равные 8 и 6 соответственно:
blastn -query trna_bacsu.fasta -db sa -out trna_bacsu_sa_blastn_1.txt -evalue 0.01 -outfmt 6 -reward 5 -penalty -4 -gapopen 8 -gapextend 6 -task blastn
Аналогичным описанному выше способом получим файл trnas_in_sa_count_1.txt.
Затем изменим, помимо значений параметров -reward, -penalty, -gapopen и -gapextend, значение параметра -word_size на минимально возможное, равное 4:
blastn -query trna_bacsu.fasta -db sa -out trna_bacsu_sa_blastn_1.txt -evalue 0.01 -outfmt 6 -reward 5 -penalty -4 -gapopen 8 -gapextend 6 -word_size 4 -task blastn
Аналогично получим файл trnas_in_sa_count_2.txt.
Полученные файлы импортируем в Excel (см. столбцы BLASTN -r 5 -p 4 и BLASTN -r 5 -p 4 -w 4 в файле trna.xlsx).
Можно выполнить поиск при минимальном значении -word_size и при значениях по умолчанию параметров вычисления веса выравнивания:
blastn -query trna_bacsu.fasta -db sa -out trna_bacsu_sa_blastn.txt -evalue 0.01 -outfmt 6 -word_size 4 -task blastn
В итоге мы получим файл trnas_in_sa_count_3.txt, информацию из которого можно использовать для заполнения ещё одного столбца (BLASTN -w 4) в таблице в файле trna.xlsx.
С помощью команды time, записываемой перед исполняемой командой, можно проследить за изменением времени работы программы при изменении параметров. Так, изменение значения параметра -word_size со стандартного (11) на равное 4 увеличило время выполнения команды в ~35 раз (время возросло с 0.432 секунд до 15.251 секунд).
Анализ результатов
Из результатов поиска гомологов видно, что наибольшего числа найденных гомологов удалось добиться при измененных значениях параметров вычисления веса выравнивания (-reward, -penalty, -gapopen и -gapextend) и значения параметра -word-size.
На "втором месте" по числу найденных гомологов - поиск при изменении значения параметра -word_size (и значениях по умолчанию параметров вычисления веса выравнивания). Наименьшее число гомологов было найдено, как правило, при выполнении поиска при значениях всех рассматриваемых параметров по умолчанию.
Выберем в одном из полученных файлов пару из тРНК кишечной палочки и найденного в геноме бактерии Streptococcus agalactiae гомологичного участка, например, тРНК BSn5_t20892 и участок 28016 - 28076 из AL766843. Эта пара находится только при изменении значений параметров вычисления веса выравнивания и -word_size. (В качестве одного из возможных объяснений этому можно привести то, что самый длинный участок выравнивания этой пары, выданного blastn, имеет длину 8 (bp). Поэтому при значении параметра -word_size, равном 11, этот участок не был найден программой blastn. При значениях по умолчанию параметров вычисления веса выравнивания, по-видимому, выравнивание с этим участком имело недостаточный вес и поэтому отсутствовало в выдаче blastn.)
Вырежем указанный участок с помощью команды seqret в отдельный файл (при этом учтём, что направление последовательности относительно записи EMBL прямое):
seqret embl:al766843 -sask
В результате получим файл al766843.fasta, содержащий найденный гомологичный участок. Исходную последовательность >BSn5_t20892 tRNA-Asn вырежем в отдельный файл t20892.fasta. С помощью программы needle выровняем две последовательности:
needle t20892.fasta al766843.fasta t20892_al766843.needle -auto
В полученном файле файле t20892_al766843.needle содержится выравнивание. Вес выравнивания равен 146, процент сходства (similarity) равен 59.5%, процент совпадений (identity) также равен 59.5%, длина выравнивания равна 74 bp. Данное выравнивание совпадает с выравниванием, выданным программой blastn при поиске гомологов с изменёнными значениями параметров вычисления веса выравнивания и -word_size.
В записи EMBL AL799843 найдём в поле FT участок последовательности генома Streptococcus agalactiae, содержащий найденный нами гомологичный участок:
FT tRNA 28010..28083 FT /product="transfert RNA-Met" FT /note="tRNAscan-SE vs 1.3 result - Cove score = 77.44"
Как мы видим, этот участок действительно является последовательностью тРНК, в частности, митионил-тРНК (tRNA-Met). (Для проверки этого можно воспользоваться, например, программой mfold, в результате чего мы получим изображение структуры, напоминающей тРНК, в которой, в частности, можно найти антикодоновую петлю и антикодон CAT.)
Ссылки
- Файл d89965.entret.
- Файл d89965.orf.
- Файл hslv_ecoli.fasta.
- Файл blastp.out.
- Файл trna_bacsu.fasta.
- Файл trna_bacsu_sa_blastn.txt.
- Файл trnas.txt.
- Файл count_trnas_in_sa.scr.
- Файл trnas_in_sa_count.txt.
- Файл trna.xlsx.
- Файл trna_bacsu_sa_blastn_1.txt.
- Файл trnas_in_sa_count_1.txt.
- Файл trna_bacsu_sa_blastn_2.txt.
- Файл trnas_in_sa_count_2.txt.
- Файл trna_bacsu_sa_blastn_3.txt.
- Файл trnas_in_sa_count_3.txt.
- Файл al766843.fasta.
- Файл t20892.fasta.
- Файл t20892_al766843.needle.