Занятие 9: Геномные браузеры
EnsEMBL
Портал EnsEMBL предназначен для визуализации известной информации о геномах человека и животных.
Для начала поищем информацию о гене человека CSNK2B (запись BA000025.embl). Начнём поиск, например, с экзона с координатами 274520 - 274709 (экзон под номером 6 в указанной записи) в человеческом геноме сервисом BLAST/BLAT. Количество результатов поиска равно одному. В разделе Alignment Locations vs. Karyotype можно увидеть расположение участка генома человека, который выровнялся с исходной последовательностью (искомый фрагмент в данном случае расположен, как мы видим, на малом плече шестой хромосомы):
На странице результатов поиска в разделе Alignment Locations vs. Query в графическом виде приведена информация о полученном выравнивании (HSP - это high-scoring segment pair):
В разделе Alignment Summary приведена таблица находок с указанием различной информации (доступен выбор строк таблицы, которые будут отображены). Среди приведённых ссылок в соответствующей графе таблицы можно найти, например, ссылку на выравнивание ([A] - Alignment) и узнать длину выравнивания (190), процент совпадений (100.00%), вес выравнивания (960), E-value (7.8e-162), направление искомой последовательности относительно записи в базе данных (обратное в данном случае) и другое. Раздел Genome Sequence (ссылка [G]) предоставляет инструменты для работы с находкой в геноме. ContigView (ссылка [C]) позволяет рассмотреть участок человеческого генома. Например, присутствует возможность интерактивной работы с участком хромосомы, приведена информация о генах на рассматриваемом участке хромосомы, а также о белок-кодирующих участках и интронах, при этом присутствует возможность настраивать отображаемую информацию (например, скрыть отображение %GC и отобразить транслируемые последовательности и старт-стоп кодоны).
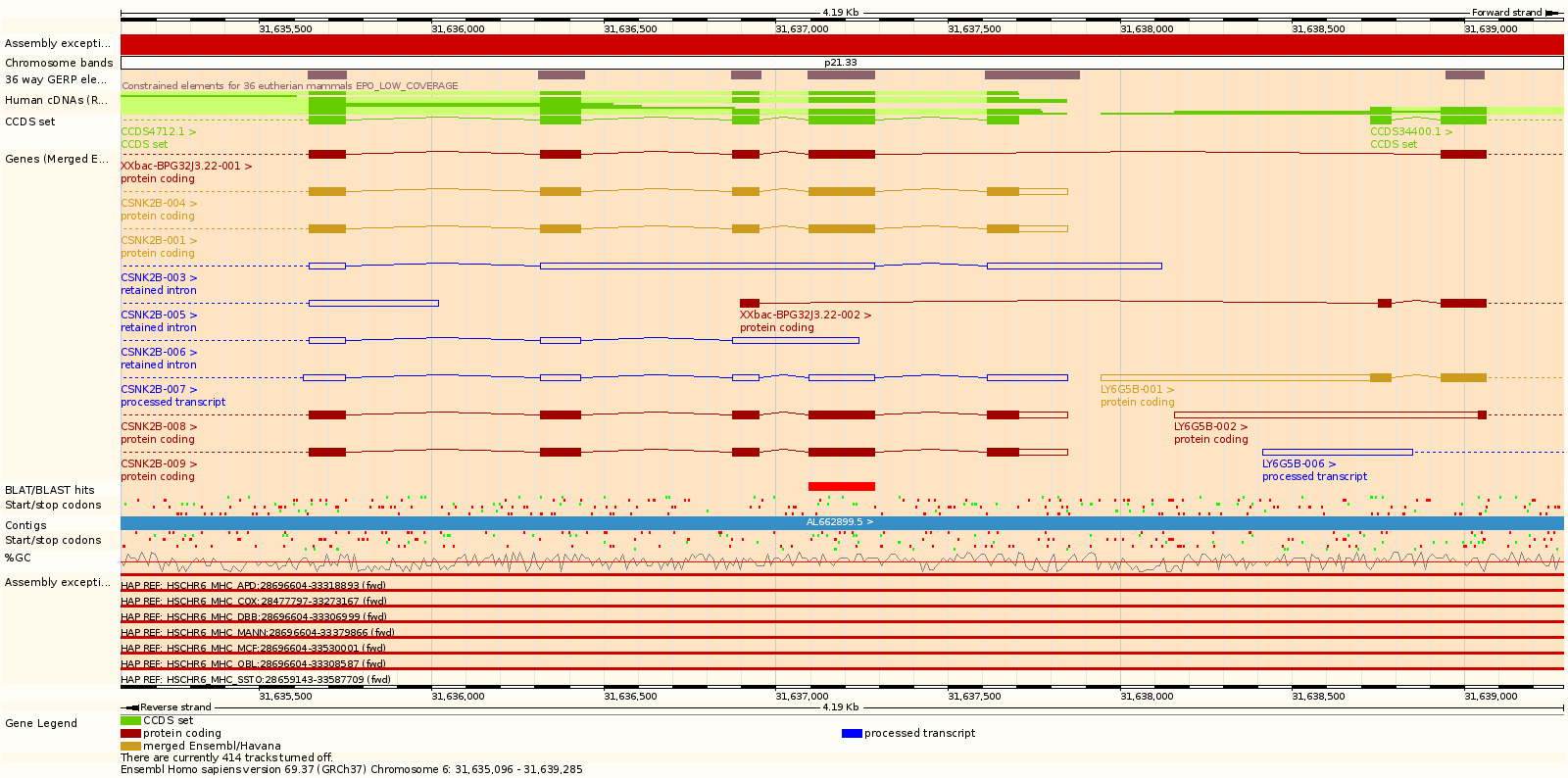
На странице Region in detail приведена различная информация о генах в интересующем нас отрезке генома. По умолчанию (см. изображение ниже) ширина отображаемого участка равна 1 Mb (миллион нуклеотидов); возможности для масштабирования присутствуют. Различными цветами (которые описаны в легенде к изображению) отмечены участки, кодирующие белок, контиги, псевдогены, а также гены, предсказанные Ensembl и Havana (информацию о группе Havana можно найти, например, на сайте The Sanger Institute):
На рассматриваемой странице геномного браузера также приведена ещё более подробная информация о фрагменте (см. изображение ниже). В данном случае фрагмент имеет координаты 6:31635096-31639285, где первая цифра 6 - номер хромосомы. Последовательность, которая была использована для поиска с помощью BLAT/BLAST, имеет стопроцентную находку, которая показана на изображении в поле BLAT/BLAST hits.
Все отображаемые поля имеют краткое описание (значок Info при наведении мышки), что помогает прояснить некоторые моменты при работе с геномным браузером. Однако не совсем ясно, например, как учитывалась рамка считывания при обозначении старт/стоп кодонов.
Можно найти ген по его названию. Для этого достаточно на домашней странице EnsEMBL ввести название гена в поисковую форму. Например, для гена CSNK2B было найдено 10 генов и 35 транскриптов в человеческом геноме.
В разделе Downloads для загрузки доступны базы данных различного формата (при этом можно выбирать отдельные участки последовательностей; доступны последовательности ДНК, кДНК, протеома и т.д. для различных животных).
Портал EnsEMBL предлагает также инструменты для работы со сравнительной геномикой, позволяя строить генетические деревья и изучать полногеномные выравнивания.
С помощью EnsEMBL можно узнать о регуляторных функциях генов, а также о уже изученных заболеваниях и других фенотипических проявлениях, например, о том, какие участки генома имеют связь с такой группой глазных заболеваний, как глаукома.
UCSC Genome Browser
Геномный браузер UCSC предоставляет широкий набор инструментов для работы с последовательностями и геномами.
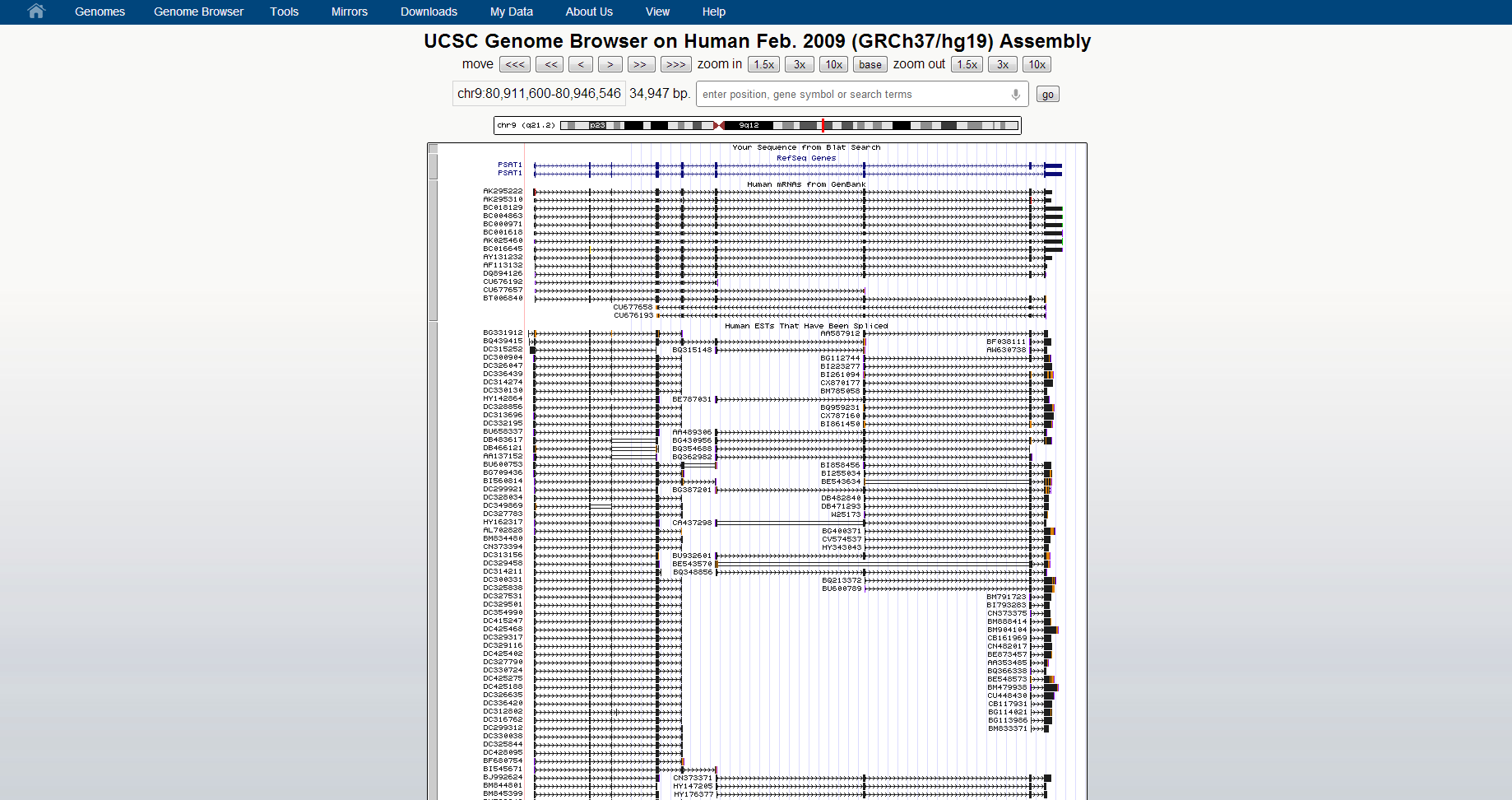
Интерфейс геномного браузера UCSC предоставляет возможности для изменения отображаемой информации в соответствии с целью его использования. На изображении ниже представлен фрагмент интерфейса геномного браузера; в соответствующем окне результатов мы можем видеть гены RefSeq, а также транскрипты (мРНК и EST человека) для указанного фрагмента генома человека. Анализ подобной информации может быть применён, например, при изучении альтернативного сплайсинга.
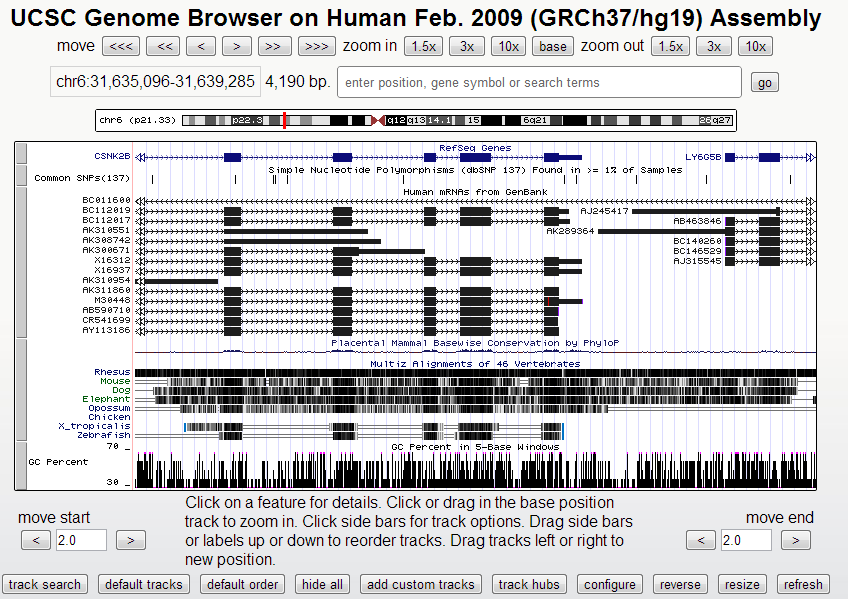
На изображении ниже приведена иллюстрация фрагмента интерфейса геномного браузера при работе с участком гена CSNK2B, который был использован для изучения особенностей геномного браузера EnsEMBL. для отображения в качестве примера были выбраны следующие поля информации: гены RefSeq (глядя на это поле, мы можем убедиться, что мы действительно осуществляли поиск по фрагменту гена CSNK2B), одиночные нуклеотидные полиморфизмы, мРНК человека (из GenBank), консервативность последовательности в различных животных, список которых можно изменить, кликнув по соответствующему полю, а также GC-содержание (в отличие от интерфейса EnsEMBL, здесь данный показатель представлен в виде отдельных пиков).
Если сравнивать геномный браузер UCSC с EnsEMBL, то можно отметить субъективно более удобную работу с участком генома (хромосомы), менее простую (однако, вероятно, не менее удобную) группировку настроек отображения информации, немного другой набор инструментов и предлагаемой смежной информации (например, есть инструмент Genome Graphs, имеются порталы с данными, полученными в ходе проектов ENCODE и Neandertal), субъективно более быструю работу и отзывчивость интерфейса.