2.
- с. Как определить характерный размер белка:
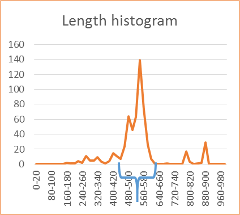
Мода в этом примере включает два пика. Намек но то, что две подгруппы. Но только намек, может случайность.
- d. Скопируйте таблицу на новый лист. Оставьте в таблице только белки с нужной архитектурой (два нужных домена) и с характерной длиной (т.к. слишком короткие последовательности вероятно фрагменты; слишком длинные - возможно содержат другие домены, еще не охарактеризованные в Pfam). Далее рекомендации, которым можно и не следовать. Из каждого семейства выберите несколько (2-5) представителей. В отчете опишите из каких отделов и семейств выбраны белки. Это будет важно при проверке профиля и обсуждении результата.
3.
- Скачайте, выровняйте последовательности выборки
получив список AC выборки составьте запрос к Uniprot. Длина строки запроса ограничена. Но 60-100 AC помещается: См образец запроса в Excel
- Ревизия выравнивания состоит в следующем:
Найти самый N-концевой консервативный блок слайды с примерами
- Начало поз 45 на слайде.
- Удалите все колонки до него (поз. 1-44)
- удалите очевидные фрагменты, те что без N-концевого конс. блока (посл-ти 11 и 13 на слайде; посл. 7 не знаю оставлять или нет
- Найти последний C-концевой консервативный блок (слайд 2). (На слайде нумерация поз. сбита,т.к. вырезал этот фрагмент для рисунка). Для решительных последняя консервативная позиция имеет номер 19. Для осторожных последняя конс. поз. 49. Я осторожный. При этом я удалил бы 16-ю последовательность, дающую длинную вставку. "консервативные" позиции начиная с 98й считаю ошибкой выравнивателя, т.к. их мало, они отделены от остльных вариабельными по длине участками. А их самих не хватит на структурный элемент белка.
- Удлить все колонки после последней колонки C-концевого консервативного блока. Слайды 3 и 4 - N-конец и C-конец после моей ревизии.
Программы пакета HMMER 2.3.2 (установлен на kodomo)
команда, вход, выход |
что делает |
полезные опции |
комментарии |
hmm2build <выходной файл с профилем> <входное выравнивание> |
Делает профиль по выравниванию |
-g <профиль для глобального выравнивания> |
--- |
hmm2calibrate <файл с профилем> |
добавляет в тот же файл-профиль строчку EDV с коэффициентами пересчета веса в нормализовнный |
--num <число случайных последовательностей, default=5000> |
Генерирует --num случайных последовательностей, строит выравнивание профиля с каждой, считает вес и рассчитывает коэффициенты пересчета |
hmm2search <профиль> <файл с последовательностями> |
находит домены в последовательностях |
-domE <порог E-value для доменов> -domT <порог веса T для доменов> |
b. Для построения профиля используйте пакет HMMER. Он установлен на kodomo. Подсказка ко всем трём программам даётся опцией -h. Более подробную информацию можно получить, выполнив команду man hmm2build (аналогично с hmm2calibrate и hmm2search).
Постройте профиль программой hmm2build.
Откалибруйте профиль программой hmm2calibrate
c. Проведите поиск по всем белкам Uniprot, включающим ОДИН ваш домен, программой hmm2search, используя откалиброванный профиль.
- Скачайте из Uniprot все бактериальные последовательности, содержащие ОДИН из двух ваших доменов в .fasta формате (в один файл). Выбирайте тот домен, который даст более разумное число последовательностей. Если один домен даёт 90 000 белков, а другой 20 000, то лучше взять другой. К чему стремиться. Такая выборка по построению будет содержать все белки с выбранной архитектурой. Но для проверки профиля важно, чтобы она содержала не только двух доменные белки с вашей архитектурой но и достаточное число других белков.
Понял, что замел под ковер непрочитанную лекцию. Исправляюсь см. презентацию о том, как проверять работу профиля
Извините за опоздание. Deadline продлил до ночи на 17 апр.
- В списке находок по профилю отметьте те, которые имеются в списке белков с выбранной доменной архитектурой.
- Подберите порог веса (или Е-value) для предсказания того, что находка имеет нужную доменную архитектуру. Для этого
- (1) постройте распределение весов находок (сортировка по убыванию веса; график весов)
- для каждого возможного порога - строчки в списке вычислите чувствительность и специфичность предсказания состоящего в том, что все строчки выше предсказываются имеющими нужную архитектуру доменов, ниже - не имеют.
- Постройте т.н. ROC кривую. Здесь будет ссылка на презентацию с объяснением.
- Подберите порог, дающий наименьшее значение параметра F1. Содержательно,этот параметр позволяет найти порог, при котором наиболее сбалансированы частоты ложно положительных и ложно отрицательных предсказаний. См презентацию.
Дополнительная информация
На kodomo, помимо пакета HMMER 2.3.2, установлен более новый пакет HMMER 3.0. Его программы отличаются отсутствием двойки в названии (например, hmmbuild вместо hmm2build). К сожалению, hmmbuild не принимает выравнивания в обычных форматах (fasta, aln, msf), поэтому с hmm2build работать проще. Впрочем, Jalview умеет сохранять выравнивания в стокгольмском формате, который hmmbuild понимает, поэтому можете работать с ним. Калибровка профиля в HMMER 3.0 не требуется.
В EMBOSS есть оболочка для пакета HMMER 2.3.2. Удобна тем, что стандартный EMBOSS интерфейс. Команды такие ehmmbuild и т.д.
4.
Coming soon


 2025
2025 2024
2024 2023
2023 2022
2022 2021
2021 2020
2020
 2019
2019 2017
2017