


 типа логотип |
Сайт | СТУДЕНТА | факультета | "Биоинженерии | & | Биоинформатики" |
Что?Белок Uracil-DNA glycosylase выполняет функцию вырезания остатков урацила из ДНК, которые могут появиться из-за ошибки включения dUMP-остатка * ДНК-полимеразы или вследствие дезаминирования** цитозина. * - встраивание в реплицирующуюся молекулу ДНК некомплементарного
матрице нуклеотида
** - реакция отщепления аминогруппы -NH2 от молекулы органического
соединения
С чем?На картинке расположенной слева вы можете видеть белок за работой. Как видим из картинки, на фрагменте молекулы ДНК сидят две молекулы белка. Именно две молекулы белка - они идентичны - мой белок встречается в виде мономера.Где?Как известно из школьного курса биологии E.Coli - это самая известная и самая изученная бактерия. Подчеркиваю - бактерия. А у бактерий ДНК располагается прямо в цитоплазме, т.е. место локализации белка - цитоплазма |
Ссылки используемые при исследовании: EcoCyc - энциклопедия по Escherichia coli штамма К12 PDBsum - сайт на котором предложена сумарная информация о третичной структуре известной на данное время (на мой взгляд он аналогичен информации на PDB банке) ENZYME - информация из базы данных ENZYME. К сожалению она не дала мне новой информации, т.к. среди предложенных ферментов семейства 3.2.2. нет моего белка. (см. результат поиска)
Расположен с 50 по 210 аминокислоту |
В последовательности моего белка обнаружен один домен: UNG-Uracil-DNA glycosylase (
EC:3.2.2)
[ банк данных Pfam ].
Про него известно, что благодаря этому домену белок P12295 является ферментом "ремонта" ДНК , который вырезает остатки урацила из ДНК путём разрушения N-гликозидных связей. Так же известно, что последовательность белка высоко конскрвативна. [перевод аннотации Pfam - http://www.sanger.ac.uk//cgi-bin/Pfam/getacc?acc=PF03167&pdb=2eug] |
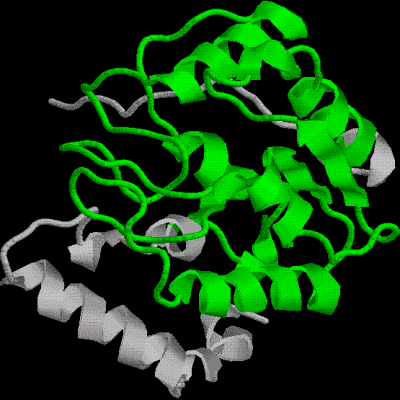
На рисунке расположенном справа домен выделен зелёным цветом. |
 |
||
| UNG_ECOLI | & | MUG_ECOLI | |
| 228 | Колличество остатков в белке | 168 | |
| Изображение | |||
| 150-210 | Расположение домена в белке | 5-161 | |
Если взглянуть на домены то видно что:
Эволюционное событие % cлучаев (белков) Комментарий Дупликация домена 0 Отличительной чертой данного семейства
являеться, то что они все имеют в
центральном полоожении домен UDG
И только егоСлияние доменов 0 Перестановка доменов 0 Задача исследования
Моя задача заключалась в том, чтобы найти в аминокислотных последовательностях особенности, отличающие подсемейства ДНК-гликозилаз, центральными белками в которых являются Урацил-ДНК-гликозилаза и mismatch-specific ДКН-гликозилаза и если возможно, охарактеризовать, в чем именно состоит функциональная роль найденных отличий.
Кроме того, предполагается создать правило, позволяющее распознавать по аминокислотной последовательности, принадлежит ли белок исследуемому функциональному подсемейству.Материалы и методы
1. Отбор "центральных белков" для составления выборок
В качестве центральных белков были выбраны белки MUG_ECOLI и UNG_ECOLI.
Оба центральных белка являются ДНК-гликозилазами, а отличие в специфике соответствующего белка , так UNG_ECOLI специализируется на урациле, а MUG_ECOLI на вырезание урацила из ошибочно сшитых вместе некомплиментарных оснований гуанина и урацила, в двухцепочечной молекуле ДНК (из-за ошибки в репликации или мутации). Он способен гидролизовать связи азот-углерод образующиеся между остатком фосфорной кислоты, присоединенным к сахарному остатку, и некомплементарно связанному урацилу.2. Составление обучающих выборок.
Обучающая выборка описана в низлежайшшей таблице. При её создании мы, сперва, выбрали два центральных белка, которые являются паралогами. Далее для каждого из них были найдены ортолог в базе данных KEGG. В состав выборок преимущественно
включались белки с id от 0.3 до 0.7, выравнивающиеся по всей длине не являющиеся белками из разных штаммов одного организма.(E.Coli).
Описание выборок
Название выборки Имя "центрального белка" Характерный признак Число ортологов в выборке Самый близкий ортолог Самый далекийортолог PDB Таксономия Set1 UNG_Ecoli Урацил ДНК гликозилаза 168 Q7UBX1 0.996 Q8F2A8 0.233 1LQJ От бактерий до человека Set2 MUG_Ecoli "mismatch-specific" ДНК гликозилаза 45 P43342 1.000 Q65R24 0.321 1LQG От бактерий до человека 3. Множественное выравнивание.
С помощью программы muscle (лежит на kodomo-count.cmm.msu.ru), было проведено множественное выравнивание двух выборок (файл со всеми последовательностями лежит в папке Credit - All.fasta; результат выравнивания вы можете найти здесь, а тут лежит файл в обработке GeneDoc). Кроме того, было получено выравнивание домена UMG для всех белков семейства из базы данных Pfam. На этом выравнивании были удалены все белки, не входящие в выборку. Так было создано выравнивание доменов белков выборки (Domain.msf - вы можете посмотреть результат в формате HTML На выравнивании домены выделены каждый в своеё группе зелёным и цветом cyan)
Предсказание аминокислотных остатков, определяющих функциональные особенности подсемейств
Для предсказания восспользовались сервером: http://math.belozersky.msu.ru/~psn/index.htm Я послал запрос на зтот сервер иполучил следующие СДП-позиции:
|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
На низлежащем рисунке изображен фрагмент выравнивания последовательности UNG_Ecoli на котором цветом Aqua (cyan) отмечены СДП-позиции (именно этот участок использовался для построения паттерна)
* 2 0 P 1 2 2 9 5 : - 5 8 - V I L G Q D P Y H G P G Q A H G L A F S V : 7 8 P 9 7 9 3 1 : - 1 4 2 - V I L G Q D P Y H G P N Q A H G L C F S V : 1 6 2 P 1 3 0 5 1 : - 1 4 9 - V I L G Q D P Y H G P N Q A H G L C F S V : 1 6 9 Q 7 V R Q 7 : - 5 8 - V I I G Q D P Y H G Y N Q A H G L A F S V : 7 8 Q 8 3 C W 4 : - 6 2 - V I L G Q D P Y H G P N Q A H G L A F S V : 8 2 Q 7 V M C 0 : - 5 6 - V I L G Q D P Y H G P N Q A H G L A F S V : 7 6 Q 9 J U C 4 : - 5 6 - V I L G Q D P Y H G A G Q A H G L A F S V : 7 6 Q 8 8 N 0 5 : - 6 5 - V I L G Q D P Y H G P G Q A H G L C F S V : 8 5 Q 8 E B 7 8 : - 5 5 - V L I G Q D P Y H G P D Q A H G L C F S V : 7 5 Q 8 P F Z 6 : - 6 6 - V I L G Q D P Y H G E G Q A H G L C F S V : 8 6 Q 8 P 4 D 7 : - 6 6 - V V L G Q D P Y H G E G Q A H G L C F S V : 8 6 V 6 6 G Q D P Y H G Q A H G L F S V
Полную картину выравнивания вы можете найти здесь. Причём SDP позиции отмечены снизу зелёными квадратиками.Создание и исследование паттерна для распознавания функционального подсемейства ДНК-гликозилаз.
Для построения паттерна выбран фрагмент c 58 по 78 выравнивания, приведенного выше на рис. 2. Этот фрагмент был выбран, потому что в нём часто располжены СДП позиции. Так-же потому что этот фрагмент участвует в образовании бетта-листа
Можно предложить следующий паттерн: V-I-L-G-Q-D-P-Y-H-G-x-x-Q-A-H-G-L-[AC]-F-S-V
По этому патерну был проведён поис по банкам данных Swiss-Prot и Trembl при условии, что неразрешено несовпадений с паттерном.При помощи программы fuzzpro(лежит на kodomo-count.cmm.msu.ru) Pезультат поиска при условии нет разрешённых несоответсвий приведен здесь: result of fuzzpro(Number of mismatches [0] )
В обучающей выборке фунциональное подсемейство UNG_Ecoli ыло представлено 31 последовательностью. Проведенный поиск позволил обнаружить 7 из 10 последовательностей, а 3 (идентификаторы) не найдены, см. таблицу II.2. Последовательности других функциональных подсемейств обучающей выборки при таком поиске не находятся В то же время, поиск позволил обнаружить новые, не вошедшие в обучающую выборку, последовательности, которые соответствуют паттерну не хуже, чем последовательности из обучающей выборки. Эти последовательности были исследованы подробнее.
Создание и исследование НММ-профиля для распознавания функционального подсемейства ДНК-гликозилаз.