Учебный сайт Фроловой Анастасии
Семестр 7. Биоинформатика в 3D.
Поиск структурных гомологов цепи А белка 4ep4 был проведен при помощи сервиса PDBeFold с параметрами RMSD между 0,8 и 2,5 и длиной выравнивания более 50% от длины белка. PDB ID отобранных структур: 4ep5(A), 4ld0(B), 1hjr(C) (рис.1).
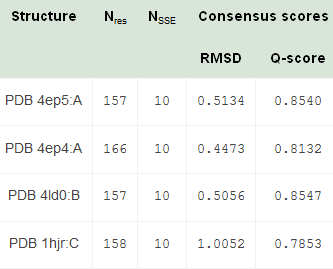
Рисунок 1. Параметры структурных гомологов 4ep4.
Выравнивание последовательностей с сервиса PDBeFold можно найти здесь, а файл с совмещенными структурами - здесь.

Рисунок 2. Cтруктурное выравнивание гомологов белка 4ep4. Зеленый - 4ep5(A), синиий - 4ep4(A), бирюзовый - 4ld0(B), желтый - 1hjr(C).
Необходимо сравнить выравнивание последовательностей по структуре (рис.4) с выравниванием последовательностей программой Muscle из Jalview (рис.3).

Рисунок 3. Выравнивание последовательностей через Muscle.

Рисунок 4. Выравнивание последовательностей по структуре.
Как видно, в позиции 40 выравнивания отличаются (рис.5). Так, в выравнивании по структуре в этой позиции стоит только лизин структуры 1hjr, а при выравнивании программой Muscle в позиции 40 стоят 3 серина и 1 лизин. Если посмотреть на структурное выравнивание белков (рис.6), то точно можно увидеть, что лизин не вписывается в группу серинов. В целом, выравнивания практически идентичны. Однако, мне кажется, что выравнивание по структуре все же правильнее.
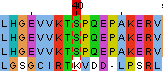
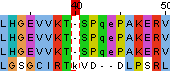
Рисунок 5. Позиция 40 в выравнивании по структуре (справа) и программой Muscle (слева).

Рисунок 6. Структурное выравнивание около позиции 40.
Для этого задания были использованы две структуры домена T-клеточного рецептора из цепи альфа (1bd2, 118-203, D) и цепи бета (1kgc, 119-247, E).
При помощи сервера SheeP были построена карты бета-листов доменов из альфа и бета цепей. Они представлены на рисунках 7 и 8.

Рисунок 7. Карта бета-листа для цепи альфа Т-клеточного рецептора.

Рисунок 8. Карта бета-листа для цепи бета Т-клеточного рецептора.
В каждой карте были найдены консервативные остатки цистеина: для альфа цепи это Cys139, для бета цепи - Cys148. Выравнивание структур домена будет проходит по этому консервативному остатку, который задаст выравнивание всего тяжа, и его соседним остаткам.
Структурное выравнивание было получено следующей командой в PyMOL:
select alpha, 1bd2 and resi 126-128+138-140+179-181 in chain D and name CA
select beta, 1kgc and resi 129-131+147-149+194-196 in chain E and name CA
pair_fit alpha, beta
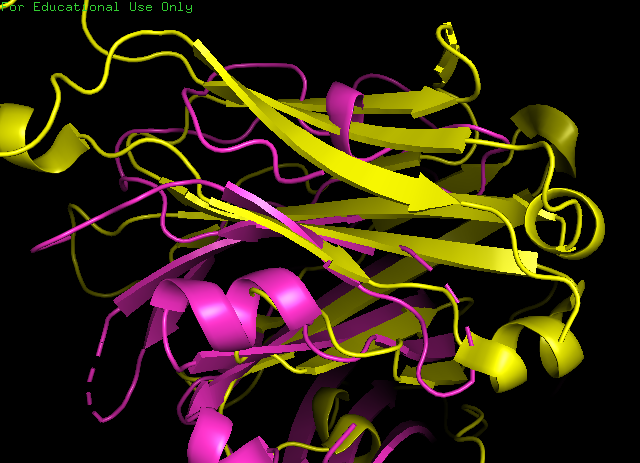
Рисунок 9. Структурное выравнивание цепи альфа(фиолетовый) и бета(желтый) Т-клеточного рецептора.
Файл с совмещением можно найти здесь. Общий ход полипептидной цепи совпадает, однако не все элементы структуры совпадают, поэтому делать выводы о сходстве топологий нельзя.