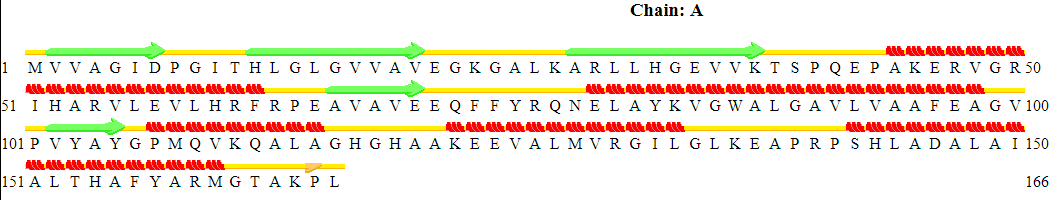Учебный сайт Фроловой Анастасии
Семестр 7. Биоинформатика в 3D.
В данном практикуме необходимо определить вторичную структура белка с помощью одной из программ (DSSP или Stride). Я решила взять уже знакомый мне белок с PDB ID: 4ep4, который точно содержит хотя бы одну альфа-спираль и хотя бы один бета-лист, а для определения вторичной структуры - программу Stride (сайт).
Из PDB файла можно определить количество вторичных структур, так в этом белке 10 альфа-спиралей и 10 бета-листов. После загрузки PDB файла, сервис выдал вот такой файл.
Необходимо сравнить границы четырех элементов вторичной структуры, полученные через сервис Stride с границами в PDB. Поэтому были выбраны 2 альфа-спирали и 2 бета-листа, информация о границах которых представлена в таблице ниже.
Таблица 1. Сравнение границ вторичных структур их PDB и Strike.
| № | Структура | Границы PDB (аминокислоты, цепь) | Границы Stride |
| 1 | Альфа-спираль | Pro141 - Met160, A | Ser142 - Met160, A |
| 2 | Альфа-спираль | Gly106 - Gly116, B | Pro107 - Ala115, B |
| 3 | Бета-лист | Ala28 - Lys37, A | Ala28 - Lys37, A |
| 4 | Бета-лист | Val102 - Tyr105, B | Val102 - Tyr105, B |
Сервис Stride так же может визуализировать полученные результаты (рис.1). Как можно заметить, одна из границ альфа-спирали под номером 1, полученная сервисом Stride, уменьшена в сравнении с границей в PDB на 1 аминокислоту (вместо pro141 - ser142). Однако во второй альфа-спирали границы уменьшены на 1 аминокислоту с обеих сторон в сравнении с PDB. Ситуация с бета-листами другая, границы с PDB и с Stride совпадают в обоих случаях.