Anna Zheltova
Fourth term (Четвертый семестр):
Molecular phylogeny (Молекулярная филогения)
Phylogeny reconstruction (реконструкция филогении)
Enzymes and metabolic pathways. KEGG database. (Ферменты и метаболические пути. База данных KEGG)
Membrane proteins(Мембранные белки)
Занятие 1
'Что такое филогенетическое дерево'
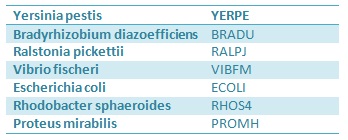
В таблице приведены отобранные бактерии.
Далее на основе дерева из задания было составлена скобочная формула и составлено дерево из части представленных в таблице бактерий.
Скобочная формула дерева:
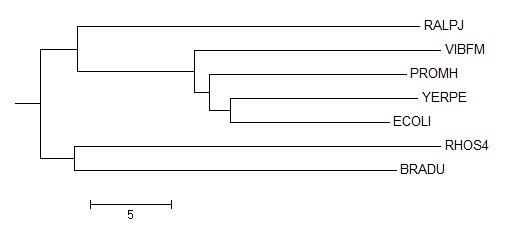
((BRADU, RHOS4),( RALPJ,(VIBFM,(PROMH,(ECOLI,YERPE)))))
Дерево из задания
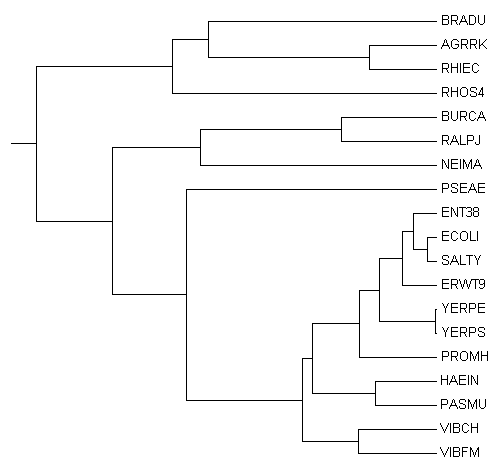
Дерево, составленное c помощью newick tree viewer online .
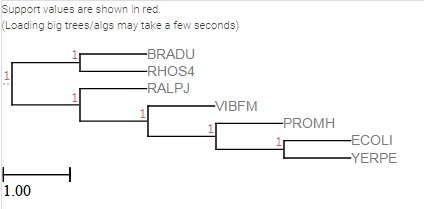
Рассмотрим нетривиальные ветви как разбиение множества листьев.
При этом будем называть ветвь нетривиальной, если она разбивает множество листьев на подмножества, в каждом из которых более одного элемента.
1) {ECOLI,YERPE,PROMH,VIBFM,RALPJ} vs {RHOS4,BRADU}
2) {ECOLI,YERPE,PROMH,VIBFM} vs {RALPJ,RHOS4,BRADU}
3) {ECOLI,YERPE,PROMH} vs {VIBFM,RALPJ,RHOS4,BRADU}
4) {ECOLI,YERPE } vs {PROMH,VIBFM,RALPJ,RHOS4,BRADU}
Занятие 2
'Реконструкция филогении'
Задание 1
Пользуясь таксономическим сервисом NCBI , было определено, к каким таксонам относятся отобранные бактерии.
Результат запроса: “Yersinia pestis”[All Names] OR “Bradyrhizobium diazoefficiens”[All Names] OR “Ralstonia pickettii”[All Names] OR “Vibrio fischeri”[All Names] OR “Escherichia coli”[All Names] OR “Rhodobacter sphaeroides”[All Names] OR “Proteus mirabilis”[All Names]

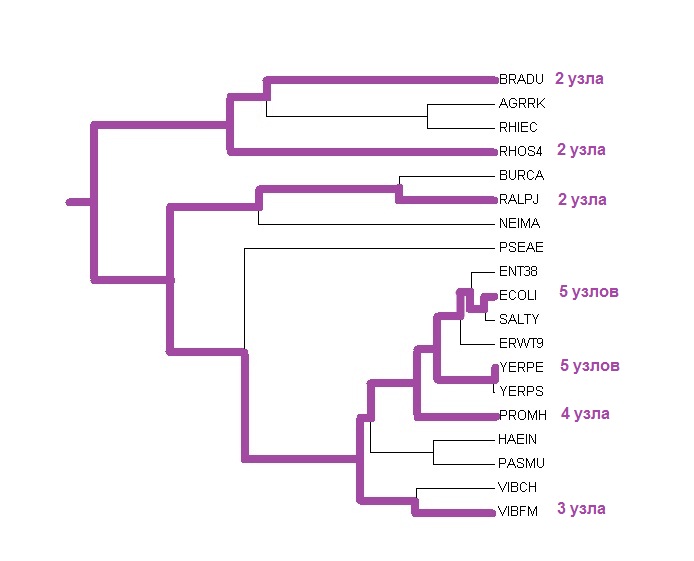
Нетривиальные ветви на дереве отобранных бактерий выделяют следующие таксоны:
ветвь 3 ({ECOLI,YERPE,PROMH} vs {VIBFM,RALPJ,RHOS4,BRADU} ) выделяет таксон Enterobacteriaceae и Enterobacteriales.
ветвь 2 ({ECOLI,YERPE,PROMH,VIBFM} vs {RALPJ,RHOS4,BRADU}) выделяет таксон Gammaproteobacteria
Можно заметить, что RALPJ относится к Betaproteobacteria, а BRADU и RHOS4 к Alphaproteobacteria.
Занятие 3
'Укоренение и бутстрэп'
Задание 1
В прошлом задании было построено филогенетическое дерево белка RSMH для семи выбранных бактерий.
Для его переукоренения в среднюю точку была использована программа retreeпакета PHYLIP. Полученное переукорененное дерево было открыто с помощью программы MEGА
Исходное филогенетическое дерево
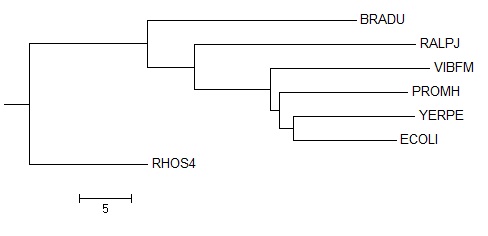
Переукорененное филогенетическое дерево