Anna Zheltova
Fourth term (Четвертый семестр):
Molecular phylogeny (Молекулярная филогения)
Phylogeny reconstruction (реконструкция филогении)
Enzymes and metabolic pathways. KEGG database. (Ферменты и метаболические пути. База данных KEGG)
Membrane proteins(Мембранные белки)
Задание 1. Построение дерева по нуклеотидным последовательностям
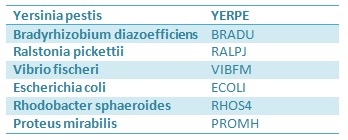
Были использованы последовательности 16S рибосомальной РНК бактерий (выбраны ранее в предыдущем практикуме) из базы полных геномов NCBI.
Последовательности 16S rRNA бактерий нужных видов бактерий были взяты из файлов с расширением .frn в записи, принадлежащей хромосоме каждой из бактерий, на сайте NCBI.
Таблица 1. Выбранные виды бактерий
Fasta-файл с последовательностями всех требуемых 16S rRNA .
Было получено выравнивание последовательностей c помощью программы Muscle в JalView.
Далее с помощью программы MEGA было получено дерево методом Neighbor-Joining (рис.1).

Рис.1. Полученное дерево
Дерево полностью совпадает с правильным (полученным в предыдущем задании)
Задание 2.Построение и анализ дерева, содержащего паралоги
Далее был проведен поиск гомологов белка CLPX_ECOLI. Для этого был создан общий файл с протеомами выбранных видов бактерий, взятый из папки P:\y14\term4\Proteomes, содержащей полные протеомы бактерий, скачанные из базы UniProt. Далее командой "cat file1 >> file2" они были сложены в один файл.
Затем был проведен поиск гомологов программой blastp с входной последовательностью белка CLPX_ECOLI по БД (файл с нужными протеомами). Порог e-value = 0.001.
Команды:
1. makeblastdb -in 3.fasta -dbtype prot -out out.fasta
2. blastp -query clpx_ecoli.fasta -evalue 0.001 -db out.fasta -outfmt 3 -out result.fasta
Как следует из выдачи, выдача дала 23 находки.Из них были отобраны только содержащие группы CLPX и HSLU (14 находок).
Было получено выравнивание последовательностей c помощью программы Muscle в JalView.
Далее с помощью программы MEGA было получено дерево методом Neighbor-Joining (рис.2).
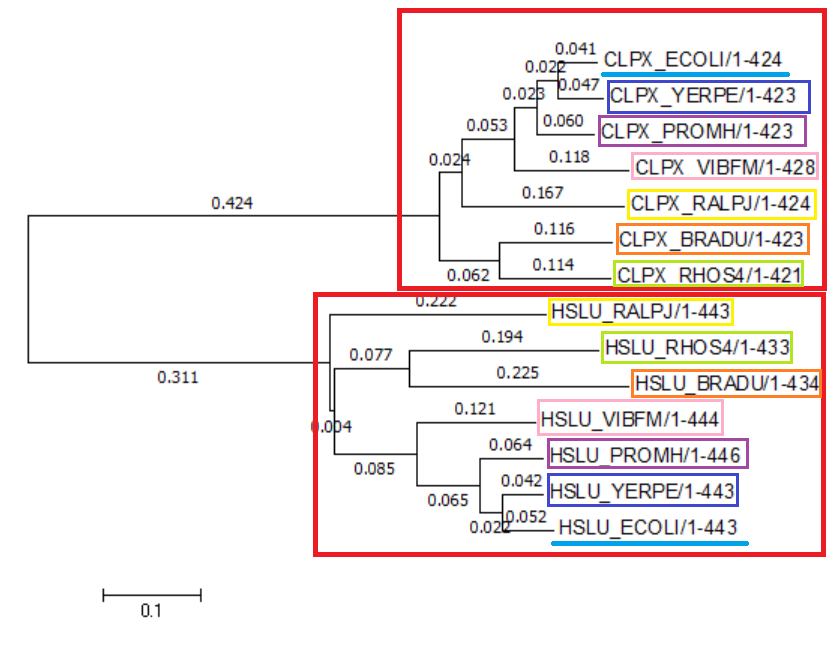
Рис.2. Полученное дерево. Красным отмечены ортологи, а синим, оранжевым, желтым, голубым, розовым, зеленым и фиолетовым - паралоги.
Паралоги - два гомологичных белка из одного организма (на примере ECOLI - подчеркнуто голубым цветом).
Ортологами называем два гомологичных белка, если они: а) из разных организмов; б) разделение их общего предка на линии, ведущие к ним, произошло в результате видообразования (красным выделены группы, внутри которых белки попарно являются ортологами)