Anna Zheltova
Fourth term (Четвертый семестр):
Molecular phylogeny (Молекулярная филогения)
Phylogeny reconstruction (реконструкция филогении)
Enzymes and metabolic pathways. KEGG database. (Ферменты и метаболические пути. База данных KEGG)
Membrane proteins(Мембранные белки)
Задание 1. Работа с KEGG ORTHOLOGY
Цель данного задания состояла в том, чтобы проверить, являются ли члены разных ортологических рядов KEGG гомологичными белками, и проанализировать их филогенетические отношения.
Для этого в метаболическом пути биосинтеза инозитолфосфата была выбрана реакция гидролиза Фосфатидилинозитол-3,4,5-трифосфата до 1-Фосфатидил-1D-мио-инозитол-3,4-бифосфата (EC: 3.1.3.86; БД KEGG PATHWAY).,которую катализирует два ортологических ряда белков. Данная реакция представлена на рис.1. (выделена красным цветом) и рис.2.
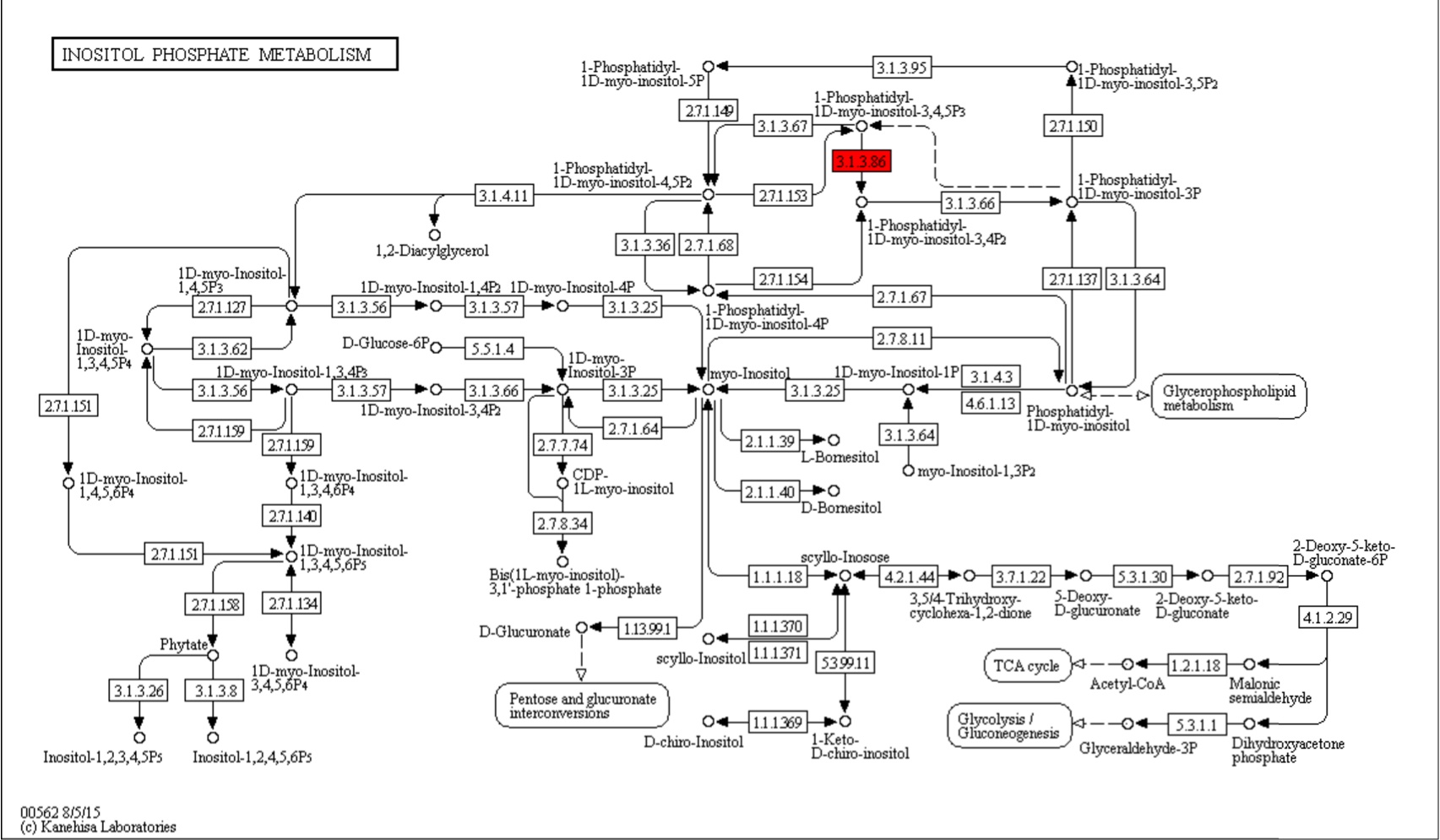
Рис.1.Метаболический путь биосинтеза инозитолфосфата.
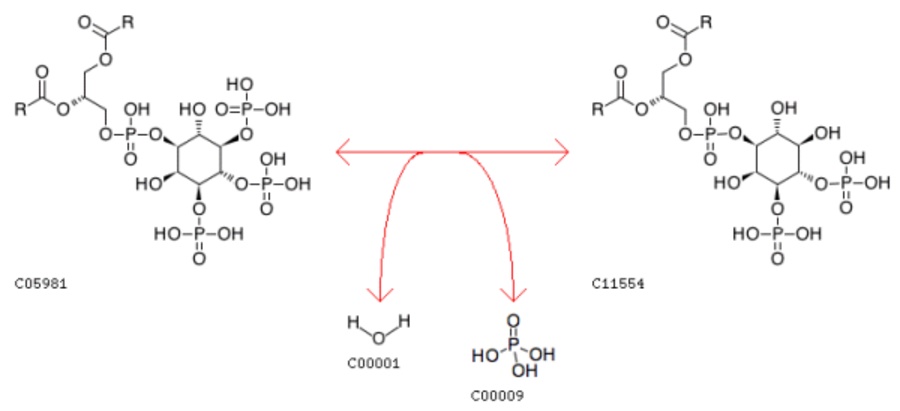
Рис.2. реакция гидролиза Фосфатидилинозитол-3,4,5-трифосфата до 1-Фосфатидил-1D-мио-инозитол-3,4-бифосфата (EC: 3.1.3.86; БД KEGG PATHWAY)
В таблице представлена информация об ортологических рядах белков, катализирующих реакцию 3.1.3.86 и ссылки на запись в БД KEGG PATHWAY.
Таблица 1. Информация об ортологических рядах белков, катализирующих реакцию 3.1.3.86.
| Идентификатор | Количество белков в ряду | Ссылка |
| K03084 | 14 (10 в UniProt, 4 в SWISS-PROT) | K03084 |
| K15909 | 20 (16 в UniProt, 4 в SWISS-PROT) | K15909 |
Для дальнейшей работы были выбраны только последовательности из UniProt
Далее были получены последовательности белков для каждого ортологического ряда с помощью сервиса "Retrieve/ID mapping" из БД Uniprot.
После этого были модифицированы идентификаторы каждого белка так, что они содержали информацию об ортологическом ряде. Идентификаторы модифицировали следующим образом: ZZZZZZ_ZZZZZ|K03084, где K03084 – идентификатор ортологического ряда в БД KEGG PATHWAY.
Полученные последовательности были объединены в один файл и выровнены с помощью метода Muscle.
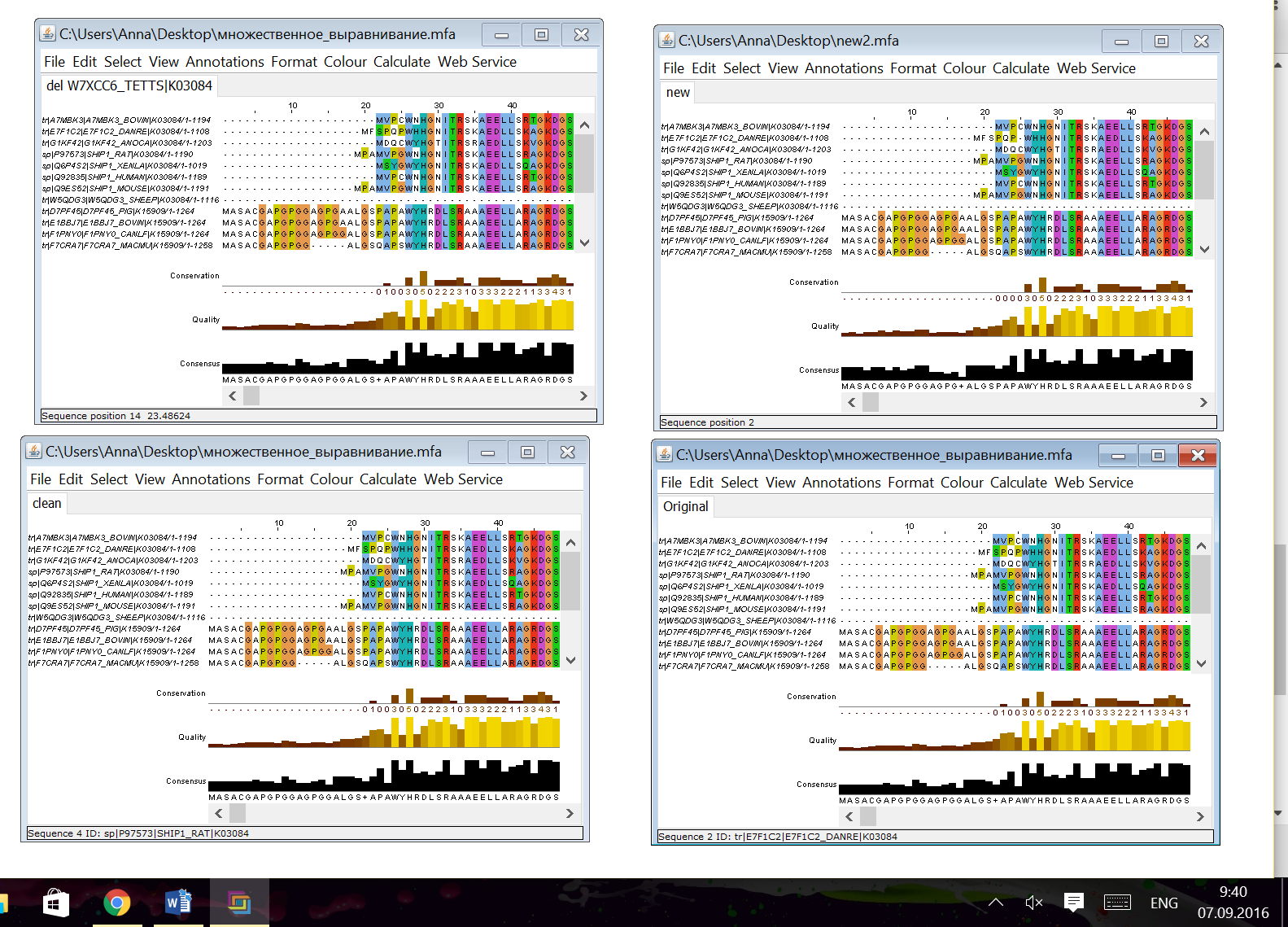
Рис.3.Окна, содержащиеся в проекте jalview.
Гомологичность белков в выравнивании
После построения выравнивания можно заключить, что вероятно белки данных ортологических рядов гомологичны между собой. В выравнивании имеется достаточно много функционально консервативных колонок. При этом присутствуют абсолютно консервативные колонки. Полученное выравнивание приведено в окне Original, раскраска ClustalX.
Проверка выравнивания
Был обнаружен белок W7XCC6_TETTS|K03084, который был плохо выравнен с остальными. Данный белок был удален из выравнивания (окно del W7XCC6_TETTS|K03084).
Также были обнаружены короткие белки, имеющие много гэпов в тех колонках, где в других последовательностях выравнивания консервативные колонки. Данные белки (F4Q584_DICFS|K03084, T1FTN2_HELRO|K15909 ) были также удалены. Стоит отметить, что были удалены белки L8H092_ACACA|K15909, L8H4T3_ACACA|K15909, L8HGG6_ACACA|K15909 (отмечу, что не смотря на то, что они принадлежат одному организму, они были плохо выровнены между собой). (окно clean).
В окне new представлено получившееся множественное выравнивание после обработки последовательностей из окна clean методом Muscle.
Построение дерева
Было построено филогенетическое дерево (программой MEGA, методом Neighbor-Joining, со 100 бутстреп-репликами).

Рис.4. Построенное дерево с уазанием количества бутстреп-реплик.

Видно, что дерево распадается на клады, которые соответствуют отдельным ортологическим рядам. Отметим, что бутстреп-поддержка ветви, разделяющей дерево на эти клады, равна 100. Таким образом, можно сделать вывод, что данное разделение является достоверным.
Если рассматривать длины ветвей, то у больших групп белков данные ветви почти одинаковы. Однако несколько выделяется ветвь M4AFT3 _XIPMA|K15909. Если рассматривать выравнивание, то можно отметить, что данная последовательность выравнена несколько хуже, чем другие, однако, это почти не заметное отличие.