Anna Zheltova
Fourth term (Четвертый семестр):
Molecular phylogeny (Молекулярная филогения)
Phylogeny reconstruction (реконструкция филогении)
Enzymes and metabolic pathways. KEGG database. (Ферменты и метаболические пути. База данных KEGG)
Membrane proteins(Мембранные белки)
Задание 1. Построить выравнивание представителей домена Pfam белков с разной доменной архитектурой
Для выполнения данного задания случайным образом был выбран домен PF00340. Этот домен представляет собой IL-1. Домен в БД Pfam
Немного истории:
IL-1 (ИЛ-1) - медиатор острого и хронического воспаления [ Braquet, P., Paubert-Braquet, M.,ea., (1989) ]. ИЛ-1 выполняет много важных функций: воздействуя на гипоталамус, вызывает лихорадку; стимулирует выход нейтрофилов из костного мозга ; активирует лимфоциты и нейтрофилы.
Интерлейкин-1 был открыт в 1972 г., когда было показано, что фитогемагглютинин или липополисахарид в культуре прилипающих клеток способствуют выделению фактора, стимулирующего пролиферацию лимфоцитов [ Gery ea 1972 ].
ИЛ-1 осуществляет различные функции: индуцирует хемотаксис полиморфноядерных лейкоцитов , хемотаксис макрофагов [ Durum ea 1985 ], пролиферацию эндотелиальных клеток и остеобластов [ Burke ea 1993 ], стимулирует дифференцировку и пролиферацию B-клеток [ Oppenheim ea 1986 ], высвобождение факторов, связанных с ростом и дифференцировкой миелоидной и лимфоидной клеточных линий [ Oppenheim ea 1986 ], играет роль в регуляции и транскрипции гена ИЛ-2 и гена ИЛ-3 в определенных Т-клеточных линиях [ Hagiwara ea 1987 ].
Согласно БД Pfam, данный домен содержит три архитектуры

Рис.1. Архитектуры домена ИЛ-1
Третья архитектура содержит всего 4 последовательности, поэтому работа проводилась с первыми двумя архитектурами.
Второй домен содержит перед собой пропептид (PF02394).
С помощью скрипта swisspfam-to-xls.py (python swisspfam-to-xls.py -z /srv/databases/pfam/swisspfam.gz -o 1.txt.xls -p PF00340) были получена информация о всех доменах в последовательностях, имеющих наш домен.
Затем с помощью сервиса Retrieve / Uniprot ID Mapping был получен файл, содержащий информацию о всех последовательностях.
С помощью скрипта tab.py из этих данных были составлены доменные архитектуры для каждой последовательности. Для каждого домена (столбцы) показана встречаемость в каждой последовательности (строки). Колонка, соответсвующая исходному домену, выделена синим.
С помощью скрипта get_length.py посчитали длину выбранного домена в каждой последовательности
С помощью скрипта uniprot-to-taxonomy.py была получена таксономия для каждой последовательности .
Все полученные данные были объединены в одну таблицу. На отдельном листе, выборкa представителей с указанием доменной архитектуры и подтаксона.
Был выбран таксон позвоночные Vertebrata. Подтаксоны: Mammalia и Actinopterygii
С помощью скрипта filter.py из общего выравнивания были выбраны соответствующие выбранным такстону и подтаксонам последовательности.
Выравнивание отобранных последовательностей домена , построенное с помощью MUSCLE, разбитое на группы по доменным архитектурам и раскрашенное внутри групп по схеме ClustalX с порогом консервативности 15%. Выравнивание обработано редактором JalView.
Подтаксоны представлены примерно порoвну, как и архитектуры. Все выбранные последовательности содержат домены примерно равной длины.
Задание 2. Построить филогенетическое дерево последовательностей ДОМЕНА
Перед именами всех последовательностей выборки были добавлены через знак "_" коды:
1 - первая архитектура, содержащая один домен
2 - вторая архитектура, содержащая два домена
A - подтаксон Actinopterygii
M - подтаксон Маmmalia
Далее было построено дерево в программе MEGA (метод Maximum Likelihood).
Скобочная формула дерева:
С помощью программы ITOL был создайн рисунок дерева с использованием методов выделения (раскраски и т.п.) ветвей и/или клад для наглядной демонстрации результата
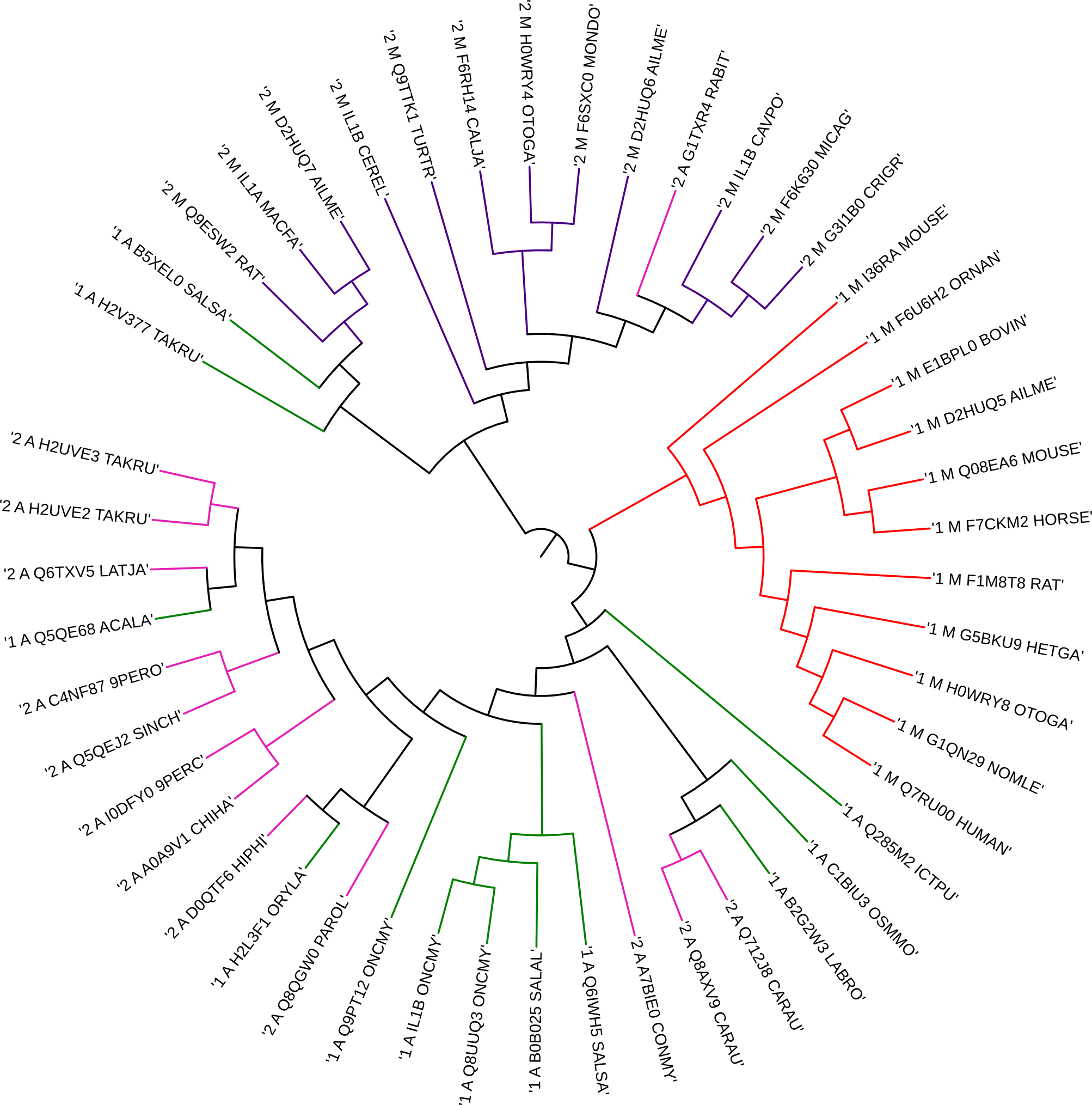
1_А - зеленый;
1_М - красный;
2_А - розовый;
2_M - фиолетовый.
На дереве видно разделение на таксоны, которое произошло раньше разделения архитектур. В пределах подтаксона Mammalia наблюдается разделение на архитектуры, которое, вероятно произошло вскоре после отделения таксона. Наличие трех последовательностей Actinopterygii среди таксона Mammalia - скорее всего случайность. Эволюцию этого подтаксона проследить сложно.
Задание 3. Построить профиль подсемейства и охарактеризовать качество его работы
В качестве подсемейства последовательностей я выбрала 1_M, так как они все входят в одну ветвь. Выравнивание всех этих последовательностей.
Далее с помощью программы hmm2build (hmm2build build_out 1.fasta) был построен профиль последовательностей и затем он был откалиброван программой hmm2calibrate (hmm2calibrate build_out). Результат.
С помощью сервиса Retrieve в Uniprot были получены все последовательности (по списку AC) , содержащие ИЛ-1 (AC последовательностей получены из сводной таблицы ).
По этому профилю был проведен поиск по всем белкам, включающим семейство доменов ИЛ-1 с помощью программы hmm2search при E-value 1E-12. Результат.
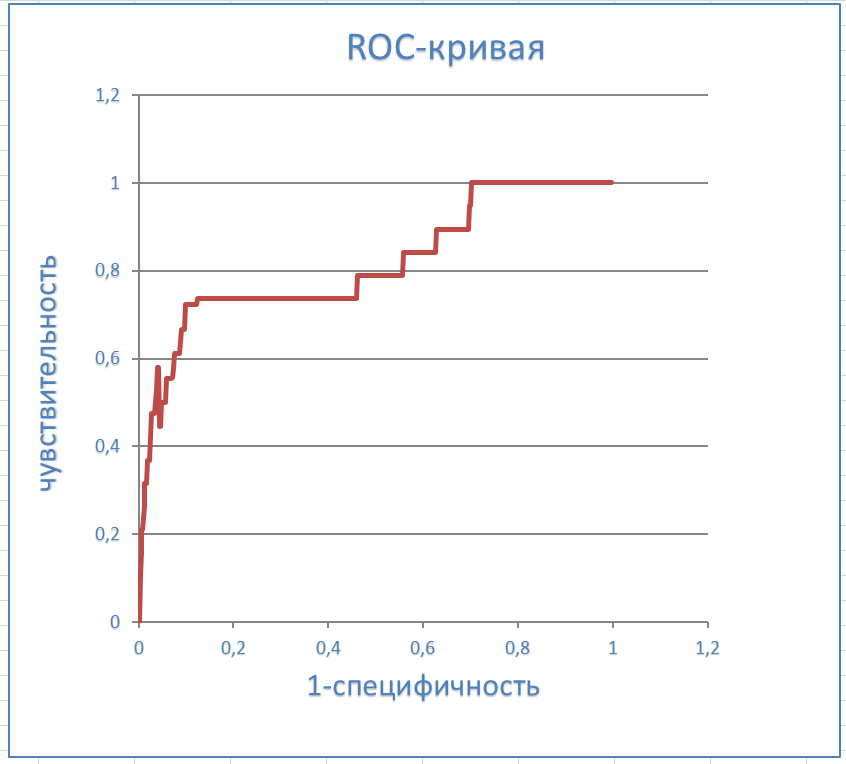
Для определения порога, по которому стоит относить последовательности к подсемейству, была построена ROC-кривая с помощью скрипта и Excel.
Далее были рассчитаны параметры чувствительности и специфичности поиска. Для их расчёта находили TP, TN, FP, FN. Для каждого значения веса предполагали, что именно оно является порогом, и посчитаем 4 переменных: 1) количество последовательностей, которое выше этого порога и достоверно содержит искомый домен (переменная TP - TruePositive) , 2) количество последовательностей, которое ниже порога и достоверно не содержит домен (TN - TrueNegative), 3) количество последовательностей, которое выше порога, но на самом деле не содержит домена (FP - FalsePositive), и 4) количество последовательностей, которое ниже порога, но достоверно содержит домен (FN - FalseNegative).

Была построена ROC-кривая: