Мембранные белки
Целью данного задания было проверить корректность предсказания сервиса TMHMM и проверить консервативность различных частей трансмембранных белков.
Для этого было составлена выборка из белков, являющихся предположительными гомологами белка Protease GlpGProtease GlpG(идентификатор PDB - 4QO2)
Поиск осуществлялся с помощью blastp в базе данных nr. Поиск производился по трем суперсемейсвтам - Archaea, Eukaryotes и Bacteria (в случае данного суперсемйства поиск производился по самым крупным бактериальным филумам - Proteobacteria, Firmicutes, Actinobacteria).
В результате было выбрано 4 белка архей, 3 белка эукариот, 21 белок протеобактерий, 4 белка фирмикут и 1 белко актинобактерий. Затем на основании этих последовательностей и последовательности исходного белка было построено множественное выравнивание с помощью сервиса Muscle. Полученное выравнивание можно скачать по этой ссылке. Изображение полученного выравнивания приведено на рис. 1.
С последовательностью белка 4QO2 была ассоциирована его трехмерная структура, на основе которой в строке аннотации TM_REAL были отмечены трансмембранные участки белка 4X32. Также принималась во внимание информация, полученная с помощью сервера предсказания PPM базы данных ОРМ.
С помощью программы TMHMM для гомолога intramembrane serine protease GlpG из Microbacterium sp. было получено предсказание трансмембранных участков (визуализация представлена на ):
# gi|661515477|gb|KEP72094.1| Length: 180 # gi|661515477|gb|KEP72094.1| Number of predicted TMHs: 5 # gi|661515477|gb|KEP72094.1| Exp number of AAs in TMHs: 106.59024 # gi|661515477|gb|KEP72094.1| Exp number, first 60 AAs: 22.59981 # gi|661515477|gb|KEP72094.1| Total prob of N-in: 0.93306 # gi|661515477|gb|KEP72094.1| POSSIBLE N-term signal sequence gi|661515477|gb|KEP72094.1| TMHMM2.0 inside 1 19 gi|661515477|gb|KEP72094.1| TMHMM2.0 TMhelix 20 42 gi|661515477|gb|KEP72094.1| TMHMM2.0 outside 43 61 gi|661515477|gb|KEP72094.1| TMHMM2.0 TMhelix 62 84 gi|661515477|gb|KEP72094.1| TMHMM2.0 inside 85 95 gi|661515477|gb|KEP72094.1| TMHMM2.0 TMhelix 96 115 gi|661515477|gb|KEP72094.1| TMHMM2.0 outside 116 119 gi|661515477|gb|KEP72094.1| TMHMM2.0 TMhelix 120 142 gi|661515477|gb|KEP72094.1| TMHMM2.0 inside 143 154 gi|661515477|gb|KEP72094.1| TMHMM2.0 TMhelix 155 177 gi|661515477|gb|KEP72094.1| TMHMM2.0 outside 178 180
В строке аннотации TM_PREDICTED были отмечены предсказанные трансмембранные участки. Проект JalView можно скачать по этой ссылке. Визуализация выравнивания с аннотацией представлена на рис. 3.
Изображение белка 4QO2, покрашеного в соответствии с выравниванием можно увидеть на рис. 4.
Исходя из выравнивания, участки, относящиеся к трансмембранным спиралям высоко консервативны, в них чаще всего встречаются неполярные остатки. Участки же между спиралями менее консервативны. Скорее всего, это обосновано тем, что практически любая мутация в участке, находящемся в мембранне, вызовет невозможность правильного расположения белка и, следовательно, выполнения им его функции. Несмотря на преобладание неполярных остатков в структуре трансмембранных участков, в них встречаются и консервативные полярные остатки (к примеру, Asn68, Lys87 (сильно положительно заряжен!), Hys168). Их консервативность можно объяснить несколькими эффектами. Во-первых, они могут участвовать в закреплении белка в мембранне. Во-вторых, возможно, они необходимы для "протаскивания" синтезируемого белка сквозь мембранну, затем же они изолируются в гидрофобных ядрах, образуемых гидрофобными остатками. Также, они могут быть нужны для предания строго определенной формы трансмембранному участку.
Предсказание трансмембранных участков с помощью TMHMM и реальная информация, полученная на основании выравнивания, в моем случае отличаются незначительно. Отсутствие в предсказании последней спирали связано с делецией соответсвующего участка у использованного гомолога. В остальных случаях участки определены приблизительно верно. Тем не менее в случае второй трансмембранной спирали для гомолога ее участок явно больше. При условии, что в выравнии "лишний участок" присутствует как и в гомологе, так и взятом изначально белке, можно утверждать, что в данном случае программа ошибается. Это, вероятнее всего, связано с тем, что "излишек" также состоит из гидрофобных аминокислот и ничто не свидетельствует о его непричастности к находящемуся рядом трансмембранному участку. В данном случае, можно предположить, что программа не может учесть всех особенностей процесса протаскивания белка через мембранну, который может регулироваться большим количеством дополнительных факторов, предсказание которых из последовательности затрднительно.

Рис. 1. Выравнивание репрезентативной выборки. Раскраска Hydrophobicity (красный цвет соответствует гидрофобным остаткам, синий - гидрофильным) + Above Identity Threshold 70%.
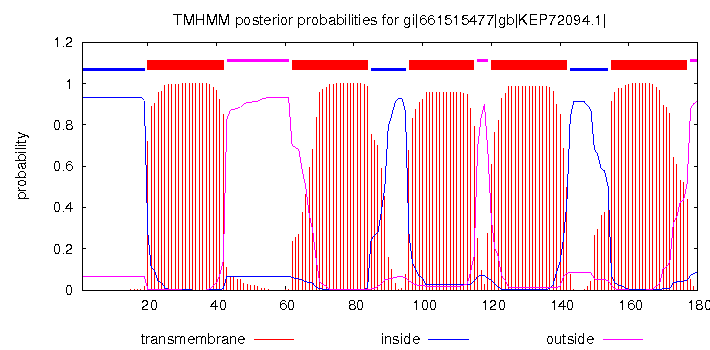
Рис. 2. Предсказание трансмембранных участков с помощью TMHMM

Рис. 3. Выравнивание гомологов исследуемого белка с аннотациями. Раскраска Hydrophobicity (красный цвет соответствует гидрофобным остаткам, синий - гидрофильным) + Above Identity Threshold 70%.
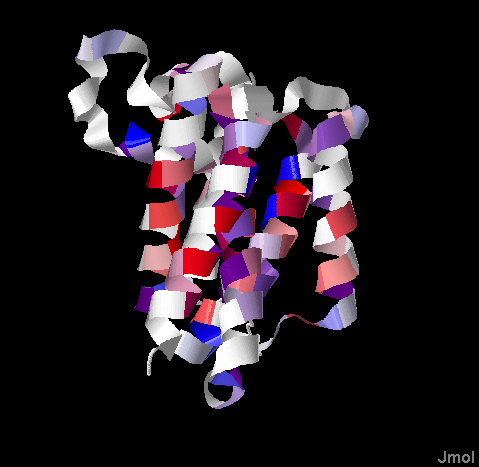
Рис. 4. Изображение трёхмерной структуры белка, покрашенного в соответствии с выравниванием. n-сторона мембраны сверху, ориентированная в p-сторону - снизу
