Ферменты. База KEGG. Работа с KEGG ORTHOLOGY
Целью данного задания было проверить, являются ли члены разных ортологических рядов KEGG гомологичными белками.
Объектом исследования была выбрана реакция с EC-номером 2.7.1.74 из биохимического пути метаболизма пуринов, карту которого можно увидеть здесь, ее изображение приведено на рис. 1
Изображение реакции приведено на рис.2. Данную реакцию катализируют два ортологических ряда белков - K00893 (дезоксицитидин киназы), K15519 (дезоксиаденозин/ дезоксицитидин киназы)
Были получены последовательности белков для каждого ортологического ряда. Все они были переименованы с целью содержать информацию об ортологическом ряде, к которому принадлежат с помощью скрипта на Python. Далее они были слиты в единый fasta-файл, который можно по этой ссылке.
Далее с помощью сервиса MUSCLE было получно выравнивание последовательностей белков, которое можно скачать по этой ссылке.
Полученное выравнивание было открыто в JalView и покрашено раскраской Clustalx. Изображение выравнивания приведено на рис.3. Проект JalView - all_prot_aln.jar
С помощью программы MEGA методом Neighbour-Joining с 100 бустреп-репликами было построено филогенетическое дерево выбранных белков. Файл полученного дерева - all_prot.nwk. Изображение полученного дерева - на рис.4. Стоит отметить, что дерево распадается на клады, соответсвуюшие ортологическим рядам за исключением клады из четырех белков, состоящей из трех белков из ряда K15519 и одного из K00893. Поддержка этой клады мала (21), а для клад, точно разделяющиз остальные белки двух рядов поддержка равна 88.
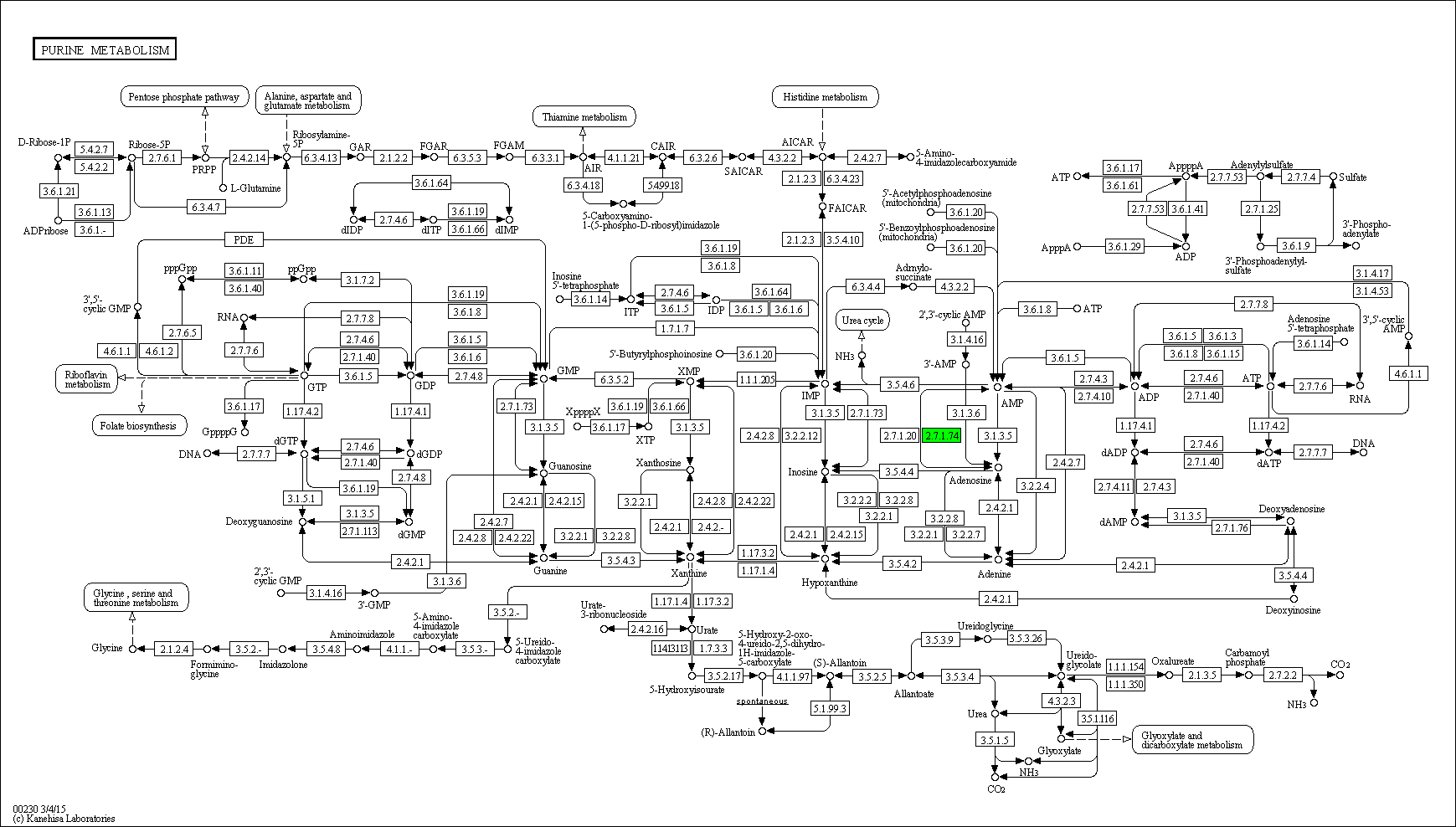
Рис. 1. Карта метаболического пути пуринов. Изображение взято из базы данных KEGG. Выбранная реакция покрашена в зеленый цвет.
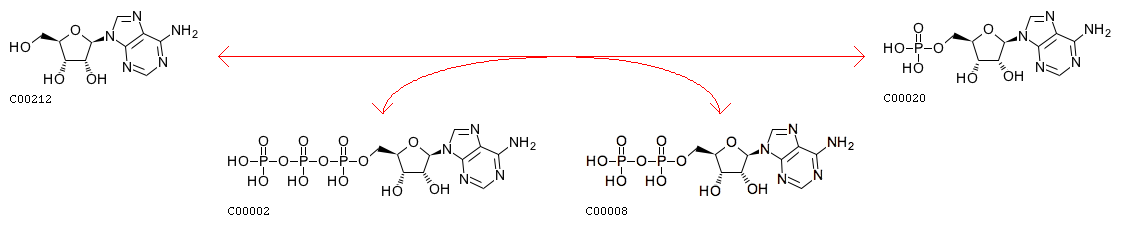
Рис. 2. Реакция с EC-номером 2.7.1.74

Рис. 3. Выравнивание белков из ортологических рядов
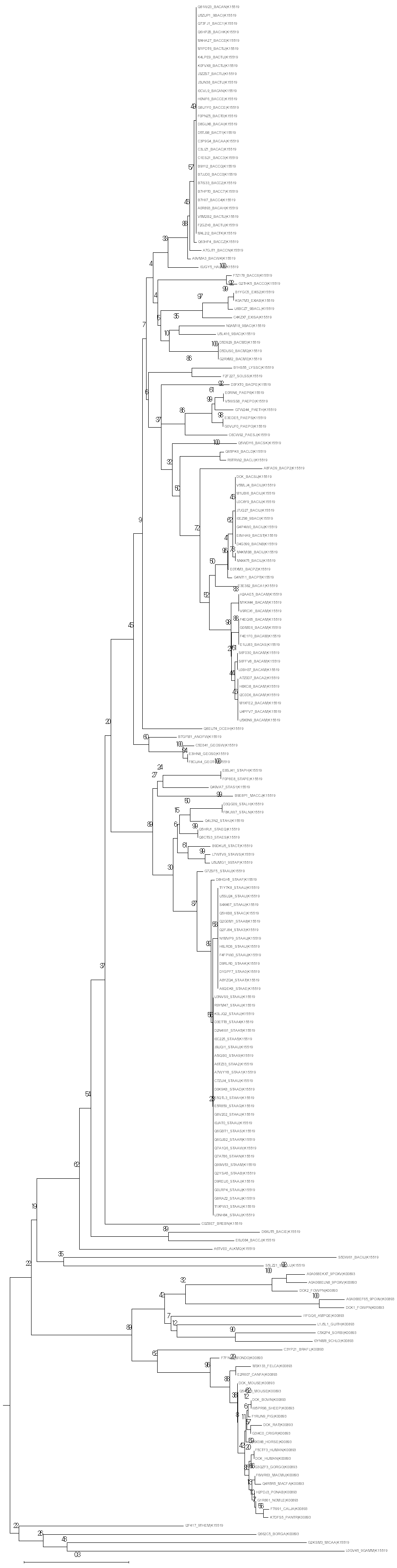
Рис. 4. Филогенетическое дерево белков.
