Сравнение деревьев, построенных разными методами
Для сравнения на основании файла множественного выравнивания с помощью программ JalView и MEGA были построены деревья с помощью различных методов, включая neighbour-joining с по-разному посчитанными матрицами расстояния, UPGMA(Average Distance) и метода наибольшего правдоподобия. Ссылка на архив с файлами, содержащими формулы деревьев и их изображениями. Названия составлялись из имени программы, в которой получали деревья, имени метода и используемой (если есть) матрицы расстояний. Файл natural_tree.nwk содержит дерево, полученное в результате предыдущего практикума. В выравнивании использовались последовательности Пептидил-тРНК гидролазаПептидил-тРНК гидролазы бактерий.
Для сравнения полученных деревьев использовался скрипт на Python, написанный с использованием библиотеки ETE2 для работы с филогенетическими деревьями. Сравнение производилось на основании метрики Робинсона-Фоулдса (за расстояние между деревьями принимается сумма A+B , где А - количество разбиений первого дерева, которые не присутствуют во втором, B - количество разбиений второго дерева, которые не присутствуют в первом). Скрипт принимает на вход имена файлов деревьев, которые надо сравнить и имя выходного файла. На выходе получаем таблицу, содеражщую информацию о расстояниях между деревьями.
Скрипт был запущен в папке с файлами деревьев при помощи команды:
python compare_trees.py -tf *.nwk -ref compare_trees.txt.xls
На выходе была получена таблица результатов, которую можно скачать по этой ссылке. Результаты сравнения каждого дерева с "настоящим" показаны в таблице 1.. Можно заметить, что наиболее точное дерево дают методы 'Average Distance using BLOSUM62' и 'Average Distance using PAM250'. На рис.1 представлено "настоящее" дерево, на рис.2 - построенное 'Average Distance using BLOSUM62', на рис.3 - методом 'Neighbour-Joining' в программе MEGA
Таблица 1. Сравнение деревьев, построенных различными алгоритмами с "настоящим" деревом.
| Имя первого дерева | Имя второго дерева | Расстояние|Максимальное расстояние | Разбиения, которые есть в первом дереве, но нет во втором | Разбиения, которые есть во втором дереве, но нет в первом |
| natural_tree.nwk | jal_tree_average_blosum.nwk | 0|14 | - | - |
| natural_tree.nwk | jal_tree_average_identity.nwk | 4|16 | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} ||| {'CLOB1', 'GEOKA', 'STAA1'} vs {'ENTFA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} ||| {'CLOB1', 'GEOKA', 'STAA1'} vs {'ENTFA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} |
| natural_tree.nwk | jal_tree_average_pam.nwk | 0|14 | - | - |
| natural_tree.nwk | jal_tree_neighbour_blosum.nwk | 4|16 | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} ||| {'CLOB1', 'GEOKA', 'STAA1'} vs {'ENTFA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} ||| {'CLOB1', 'GEOKA', 'STAA1'} vs {'ENTFA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} |
| natural_tree.nwk | jal_tree_neighbour_identity.nwk | 4|16 | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} ||| {'CLOB1', 'GEOKA', 'STAA1'} vs {'ENTFA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} ||| {'CLOB1', 'GEOKA', 'STAA1'} vs {'ENTFA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} |
| natural_tree.nwk | jal_tree_neighbour_pam.nwk | 4|16 | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} ||| {'CLOB1', 'GEOKA', 'STAA1'} vs {'ENTFA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} ||| {'CLOB1', 'GEOKA', 'STAA1'} vs {'ENTFA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} |
| natural_tree.nwk | mega_max_prob.nwk | 2|15 | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} |
| natural_tree.nwk | mega_neighbour.nwk | 2|15 | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} | {'CLOB1', 'STAA1'} vs {'ENTFA', 'GEOKA', 'LACLM', 'LISMO', 'STRP1', 'STRPN'} |
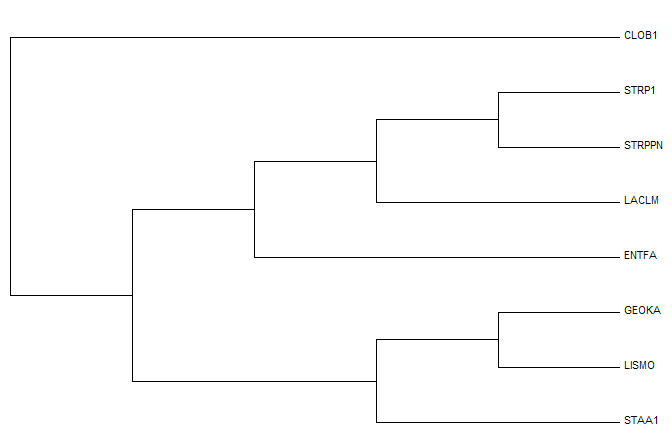
Рис. 1. "Настоящее" дерево.
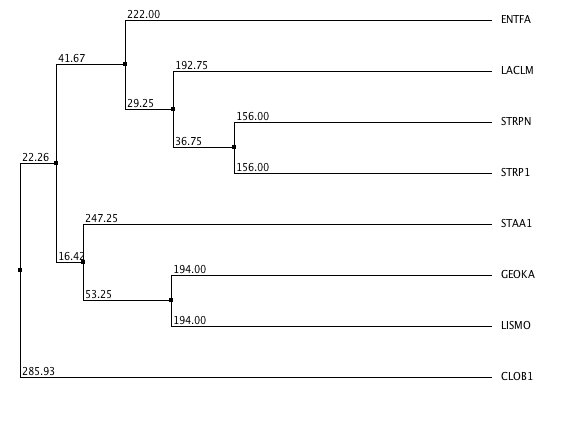
Рис.2. Дерево, построенное методом 'Average Distance using BLOSUM62'
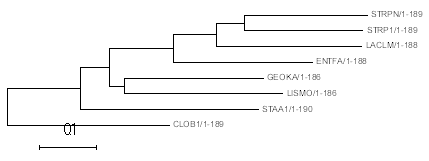
Рис.3. Дерево, построенное методом 'Neighbour-Joining' в программе MEGA
