Укоренение филогенетических деревьев
Для построения филогенетического гена использовалось множественное выравнивание пептидил-тРНК гидролаз бактерий, выбранных в предыдущем практикуме. Скачать его можно по этой ссылке.
Укоренение в среднюю точку
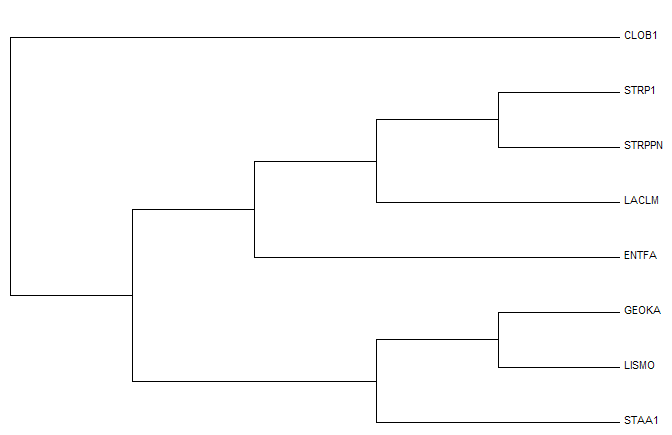
Дерево было построено в программе JalView с использованием метода 'Neighbor-Joining using % identity'. Скачать файл с формулой дерева можно по этой ссылке. Изображение дерева приведено на рис.1Для укоренения дерева в среднюю точку использовалась программа retree пакета PHYLIP. Была использована опция 'M' ("Midpoint root the tree"). Выдачу непосредственно программы можно увидеть на рис. 2. Далее дерево было сохранено с помощью опции 'W'("Write tree to a file"). Изображение дерева в программе MEGA можно увидеть на рис. 3. Файл в .nwk-формате можно скачать по этой ссылке Укоренение произошло в ветвь {CLOB1, STAA1} vs {GEOKA, LISMO, STRP1, STRPN, LACLM, ENTFA}. Если сравнить полученное дерево с "правильным" (рис.4), то можно заметить, что укоренение произошло неверно.
Рис. 1. Неукорененное дерево, полученное методом 'Neighbor-Joining using % identity'.
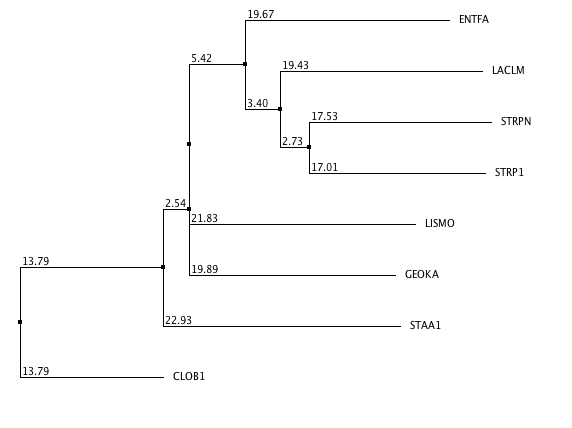
Рис.2. Вид укоренненого в среднюю точку дерева в программе PHYLIP
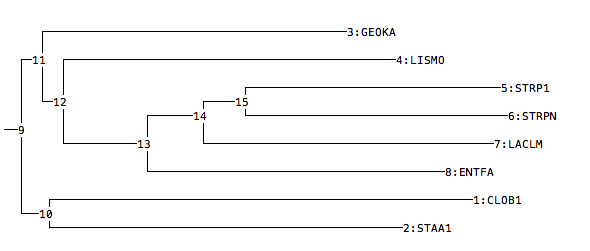
Рис.3 Вид укоренненного в среднюю точку дерева в программе MEGA
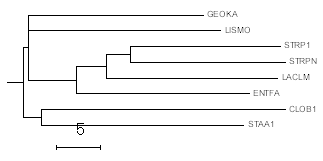
Рис.4. "Правильное" филогенетическое дерево.