Проверка дерева цитохромов b
В данном задании необходимо было проверить дерево цитохромов, построенное в статье Early Evolution of Cytochrome bc Complexes (Schutz et al).
В статье авторы используют белки архей в качестве out-группы и производят укоренение с ее помощью. Возникает вопрос, имеют ли авторы право, вообще говоря, проводить укоренение подобным образом. С целью проверить данный факт, были скачаны последовательности белков цитохромов b, которые, вероятнее всего, использовались авторами. Далее на основания выравнивания этих белков программой Muscle было построено разными способами несколько укорененных деревьев.
В первом случае использовался метод Neighbour-joining с использованием метрики Kimura.
Для построения дерева таким образом использовался пакет Philip
- Были переименованы все последовательности в выравнивании, для того, чтобы имя каждой последовательности было не более 10 символов (требования формата Phylip). Переименование осуществлялось с помощью скрипта на Python
- Далее с помощью команды "seqret article_aln.fasta phylip::article_aln.phylip" файл выравнивания был приведен к формату Phylip.
- Было получено 500 boot выравниваний (в 10 раз меньше, чем указано авторами, хотя, скорее всего, и это количество излишне) с помощью команды seqboot пакета Phylip.
- С помощью скрипта на Python полученный файл с выравниваниями был разделен на отдельные выравнивания.
- С помощью скрипта на Bash был получен файл с деревьями, полученными на основании этих 500 последовательностей.
- Программой consense пакета Phylip было получено консенсусное дерево.
- С помощью скрипта на Python полученное консенсусное дерево было модифицировано (нормализована поддержка каждой ветви, осуществлено обратное переименование последовательностей)(итоговый файл).
- C помощью онлайн-сервиса Phyfi было получено изображение консенсусное дерево. Его изображение приведено на рис. 1.
- На основе исходного выравнивания также было построено дерево с использованием той же метрики. Файл дерева в формате .nwk. Изображение укорененного в среднюю точку дерева в программе MEGA приведено на рис.2. Видно, что выделение архей в отдельную группу подтверждается (возникает, правда, вопрос о справедливости использования в данном случае укоренения в среднюю точку). При сравнении с консенсусным деревом можно заметить, что деревья по ветвям не отличаются.
Также были построены деревья с помощью программы MEGA с использованием Neighbor-Joining (рис. 3) и Maximum Likelihood(рис. 4). Полученные деревья также были укоренены в среднюю точку. В данных случаях археи также попадали в отдельную кладу.
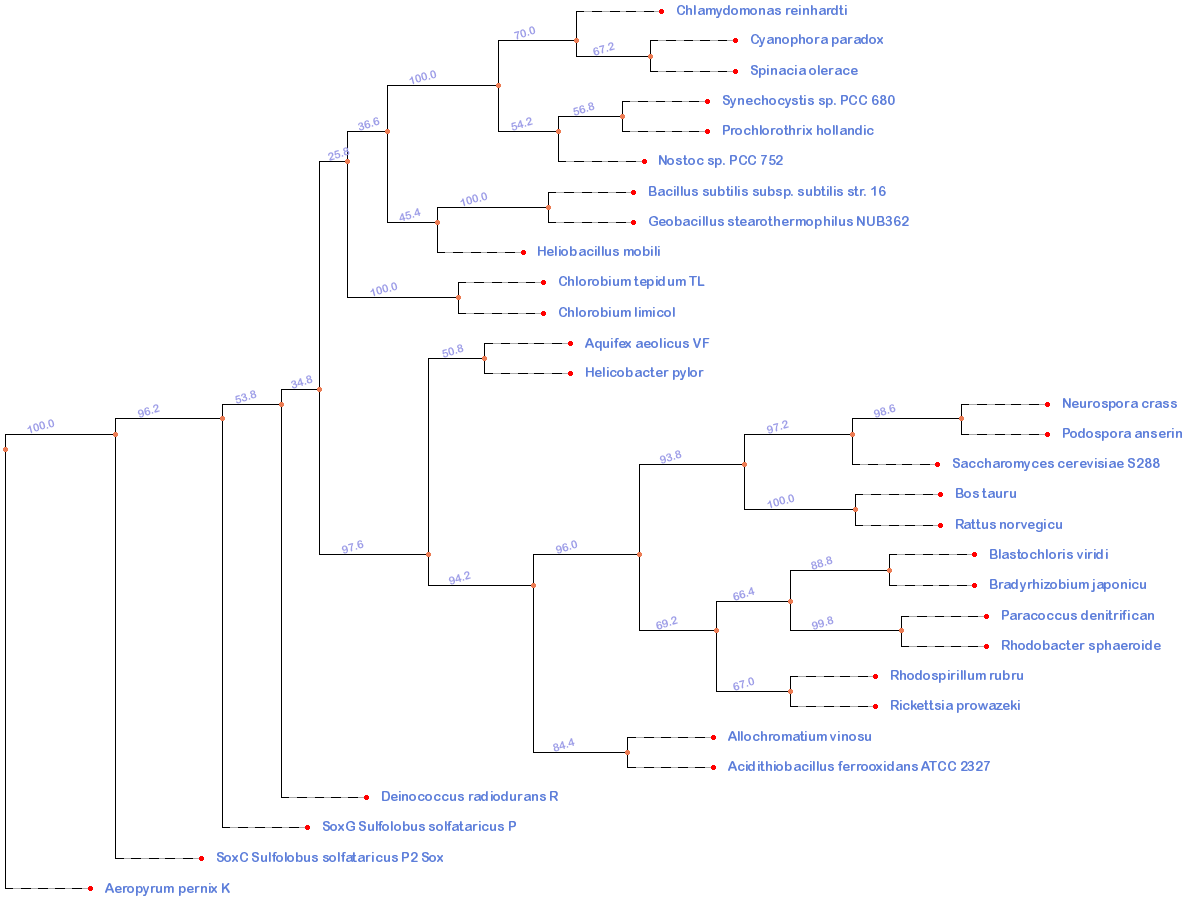
Рис. 1. Консенсусное дерево, полученное на основании 500 бутстрепов.
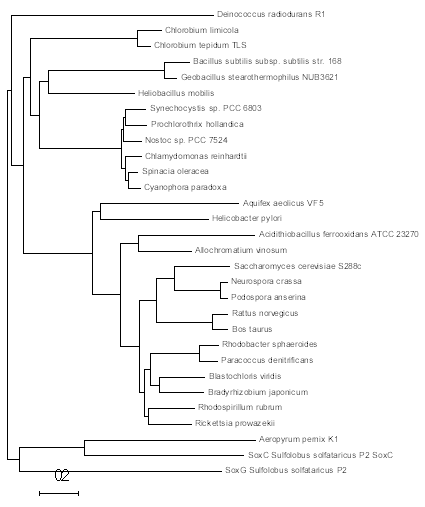
Рис. 2. Дерево, построенное на основании метрики Кимуро

Рис. 3. Дерево, полученное в программе MEGA с помощью метода Neighbor-Joining
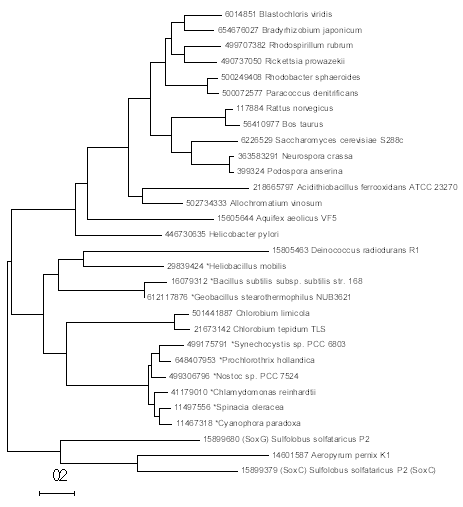
Рис. 4. Дерево, полученное в программе MEGA с помощью метода Maximum Likelihood
