Филогенетические деревья
В данном занятии нами изучались филогенетические деревья. Введем несколько терминов (большая их часть взята из презентации Дарьи Дибровой):
- Дерево – связный ациклический граф: каждая вершина имеет 0 или более дочерних вершин и не более одной родительской вершины.
- Филогенетическое дерево - дерево, отражающая гипотетическое взаимное происхождение видов. Так как обычно из одного вида образуется два (редкие исключения - когда сразу несколько (>2) популяций оказываются в разных средах обитания, к примеру.), то дерево теоретически бинарное. Несмотря на это дерево, построенное программой может не являться бинарным из-за недостаточности данных о происхождении видов.
- Ветвь - Связь между узлами графа (как внутри, так и между внутренними узлами и листами)
- Нетривиальная ветвь дерева - ветвь, разбивающая множество листьев на два подмножества с мощностью >1
Мною было отобрано 8 видов бактерий из отдела Firmicutes. Предлагалось выбрать их из более длинного списка видов, размещенного по следующему адресу.
Далее, на основании филогенетического дерева для всех видов списка предлагалось создать дерево для выбранных мною видов. Таблица с названием вида и соответствующей мнемоникой (Таблица 1) и изображение дерева (Рис. 1.) для всех видов приведено ниже.
Сначала была создана скобочная формула дерева:
(CLOB1,((((STRP1, STRPPN),LACLM),ENTFA),((GEOKA, LISMO), STAA1)))
и помещена в файл my_tree.nwk. Далее с помощью программы MEGA было получено изображение филогенетического дерева видов, которое приведено на рис. 2
Следующим шагом стало выписывание всех нетривиальных ветвей в моем дереве:
- {STRP1, STRPPN} против {LACLM, ENTFA, GEOKA, LISMO, STAA1, CLOB1}
- {STRP1, STRPPN, LACLM} против { ENTFA, GEOKA, LISMO, STAA1, CLOB1}
- {STRP1, STRPPN, LACLM, ENTFA} против { GEOKA, LISMO, STAA1, CLOB1}
- {STRP1, STRPPN, LACLM, ENTFA, STAA1, CLOB1} против { GEOKA, LISMO}
- {STRP1, STRPPN, LACLM, ENTFA, CLOB1} против { GEOKA, LISMO, STAA1}
Далее была получена полная таксономия видов таксономического сервиса NCBI
. Данные приведены в таблице 2. Путем сравнения филогенетического дерева и таксономии получил следующие соответствия, результаты также отображены на рис. 3:- множество {GEOKA, LISMO, STAA1} соответствует порядку Bacillales
- множество {STRP1, STRPPN} соответсвует роду Streptococcus
- множество {STRP1, STRPPN, LACLM} соответствует семейству Streptococcaceae
- множество {STRP1, STRPPN, LACLM, ENTFA} соответствует порядку Lactobacillales
- множество {GEOKA, LISMO, STAA1} соответствует порядку Bacillales
- множество {STRP1, STRPPN, LACLM, ENTFA, STAA1, GEOKA, LISMO, STAA1} соответствует классу Bacilli
- Все бактерии находятся в отделе Firmicutes
Таблица 1. Название и мнемоника выбранных видов
| Название | Мнемоника |
|---|---|
| Clostridium botulinum | CLOB1 |
| Enterococcus faecalis | ENTFA |
| Geobacillus kaustophilus | GEOKA |
| Lactococcus lactis | LACLM |
| Listeria monocytogenes | LISMO |
| Staphylococcus aureus | STAA1 |
| Streptococcus pneumoniae | STRPN |
| Streptococcus pyogenes | STRP1 |
Таблица 2. Название вида и его таксономия
| Название вида | Таксономия |
| Clostridium botulinum | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Enterococcus faecali | Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Geobacillus kaustophilus | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Lactococcus lactis | Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Lactococcus |
| Listeria monocytogenes | Bacteria; Firmicutes; Bacilli; Bacillales; Listeriaceae; Listeria |
| Staphylococcus aureus | Bacteria; Firmicutes; Bacilli; Bacillales; Staphylococcaceae; Staphylococcus |
| Streptococcus pneumoniae | Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
| Streptococcus pyogenes | Bacteria; Firmicutes; Bacilli; Lactobacillales; Streptococcaceae; Streptococcus |
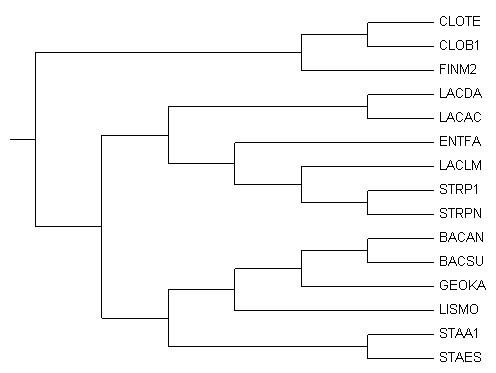
Рис. 1. Филогенетическое дерево всех видов из списка.
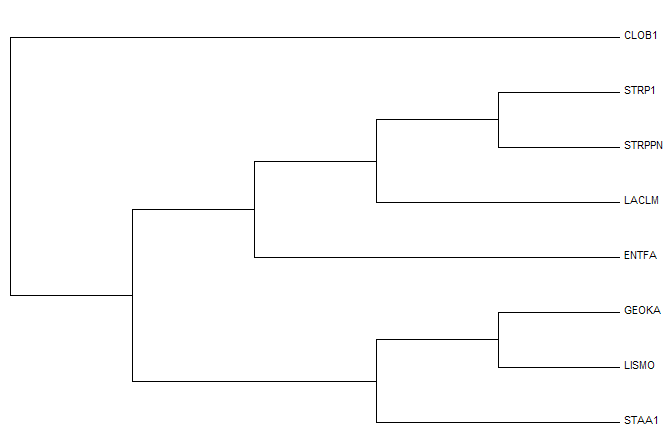
Рис. 2. Филогенетическое дерево для выбранных мною видов (приведены выше). Получено с помощью программы MEGA.
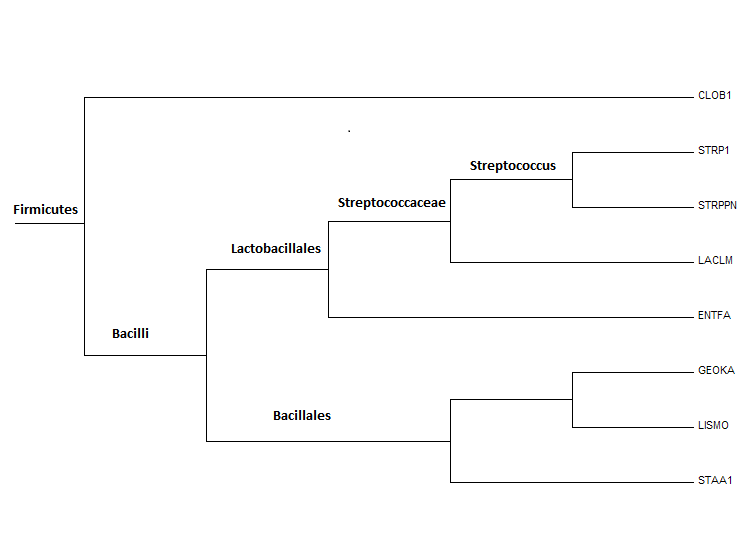
Рис. 3. Филогенетическое дерево для выбранных мною видов (приведены выше). Отмечены соответсвующие разбиению таксоны. Получено с помощью программы MEGA.
