| Учебный сайт Саши Погорельской |

|
|
||||||
| Главная | Семестры | Скрипты | Обо мне | Ссылки | ||
|
|
Представления филогенетического дерева Для выполнения этого практикума было дано филогенетическое дерево нескольких бактерий отдела Firmicutes. Нужно было выбрать несколько организмов (таблица 1) и составить дерево для них (рисунок 1). Таблица 1.Выбранные бактерии
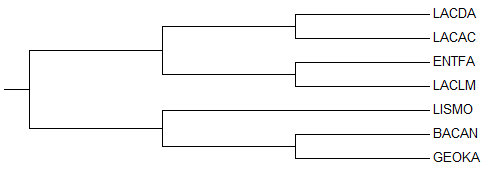
Рисунок 1. Филогенетическое дерево для бактерий из таблицы 1. Получено с помощью программы MEGA Скобочная интерпретация дерева, изображенного на рисунке 1:
Полученное дерево содержит несколько нетривиальных ветвей:
С помощью сервиса NCBI я посмотрела таксономические группы бактерий из таблицы 1 и их соответсвие дереву. Все эти бактерии принадлежат классу Bacilli. Первое разветвление дерева соответствует делению на порядки: бактерии из порядка Lactobacillales (LACDA,LACAC,ENTFA,LACLM) противопоставляются порядку Bacillales (LISMO,BACAN,GEOKA). Следующее разветвление - деление на семейства, при чем LACDA и LACAC относятся к одному семейству Lactobacillaceae, а ENTFA и LACLM - к разным (Enterococcaceae и Streptococcaceae соответственно). В нижней половине дерева (на рисунке 1) деление проходит строго аналогично таксономическому делению: BACAN и GEOKA принадлежат семейству Bacillaceae, а LISMO - другому - Listeriaceae. Укоренение в среднюю точку Для реконструкции дерева бактерий я использовала последовательности белков - факторов элонгации EFTS. Выравнивание этих белков можно скачать здесь. Дерево, реконструированное из этого выравнивания методом "Neighbor Joining Using % Identity" неукорененное, один из методов укоренения - укоренение в среднюю точку (середина самой длинной ветви). Изображение такого дерева представлено на рисунке 2. 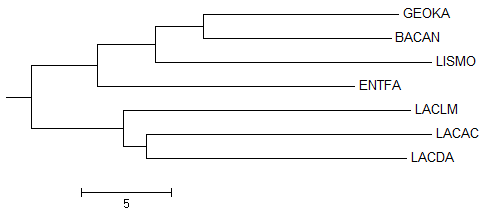
Рисунок 2. Укорененное в среднюю точку дерево. Получено с помощью программы MEGA Корень разделяет ветви {BACAN,GEOKA,LISMO,ENTFA} против {LACDA,LACAC,LACLM}, что неправильно с точки зрения филогении (ошибка в порядке). Построение дерева по нуклеотидным последовательностям Для работы были взяты последовательности РНК 16S субъединицы рибосомы бактерий из таблицы 1. Fasta-файл с ними можно скачать здесь. Последовательности были выровнены программой muscle, был получен этот файл. По нему с помощью программы MEGA было построено дерево методом максимального правдоподобия. Полученное дерево представлено на рисунке 3. 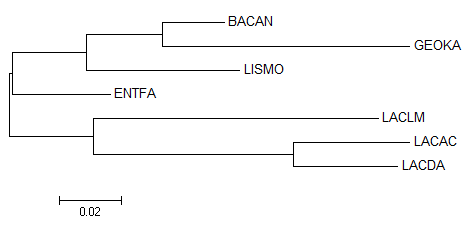
Рисунок 3. Дерево, построенное по последовательностям 16S субъединицы рибосом. Получено с помощью программы MEGA Ветви представленного на рисунке 3 дерева полностью соответствуют полученным ранее (из белковых последовательностей). Построение и анализ дерева, содержащего паралоги С помощью программы blast были найдены все гомологи белка CLPX_BACSU бактерии Bacillus subtilis в протеомах бактерий, указанных в таблице 1. Получилось 33 гомолога. Выравнивание последовательностей этих белков можно скачать тут. По выравниванию методом максимального правдоподобия было построено дерево. Оно представлено на рисунке 4. 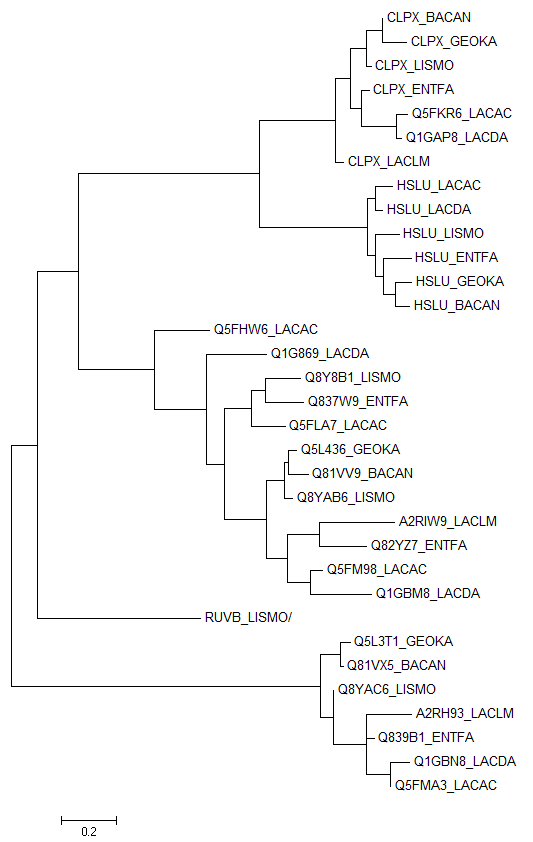
Рисунок 4. Дерево, построенное по гомологам белка CLPX_BACSU. Получено с помощью программы MEGA Паралогами называются белки, являющиеся гомологичными и принадлежащие одному организму. Паралоги образуются в результате внутригеномных мутаций одного вида. Было найдено несколько паралогов, например, 4 белка организма Bacillus anthracis (исходный CLPX_BACAN, HSLU_BACAN, Q81VV9_BACAN и Q81VX5_BACAN). Ортологичные белки - гомологичные белки, разошедшиеся в результате видообразования. Группой таких белков является, например вся верхняя ветвь рисунка 4, включающая 7 белков (CLPX_GEOKA, CLPX_BACAN, CLPX_LISMO, CLPX_ENTFA, CLPX_LACLM, Q5FKR6_LACAC, Q1GAP8_LACDA). |
||||||||||||||||||||||||||||||||
| © Pogorelskaya Sasha | Last modification date: 09.05.15 | |||||||||||||||||||||||||||||||||