| Учебный сайт Саши Погорельской |

|
|
||||||
| Главная | Семестры | Скрипты | Обо мне | Ссылки | ||
|
|
Восстановление предкового состояния доменной архитектуры Эволюционные домены являются единицами непрерывной эволюции белков, то есть в процессе эволюции с ними просходят только локальные изменения: небольшие вставки и делеции, мутации отдельных аминокислотных остатков. Для работы в этом задании был выбрано семейство доменов D-ser_dehydrat - предполагаемый домен серин-дегидразы (идентификатор PF14031). Он был найден в С-концевой области D-серин дегидразы дрожжей, являющейся цинк-зависимым ферментом. Он катализирует реакцию превращения D-серина в пируват и аммиак. На рисунке 1 представлены все типы доменных архитектур, включающие в себя анализируемый домен D-ser_dehydrat. Для дальнейшей работы я выбрала первые два их них, так как они довольно хорошо представлены в различных последовательностях (включают 1012 и 84 последовательности соответсенно). Одна из них включает второй домен - аланиновую рацемазу (фермент, содержащий такой домен способен катализировать реакцию превращения D-аланина в L-аланин). Другая архитектура однодоменная. 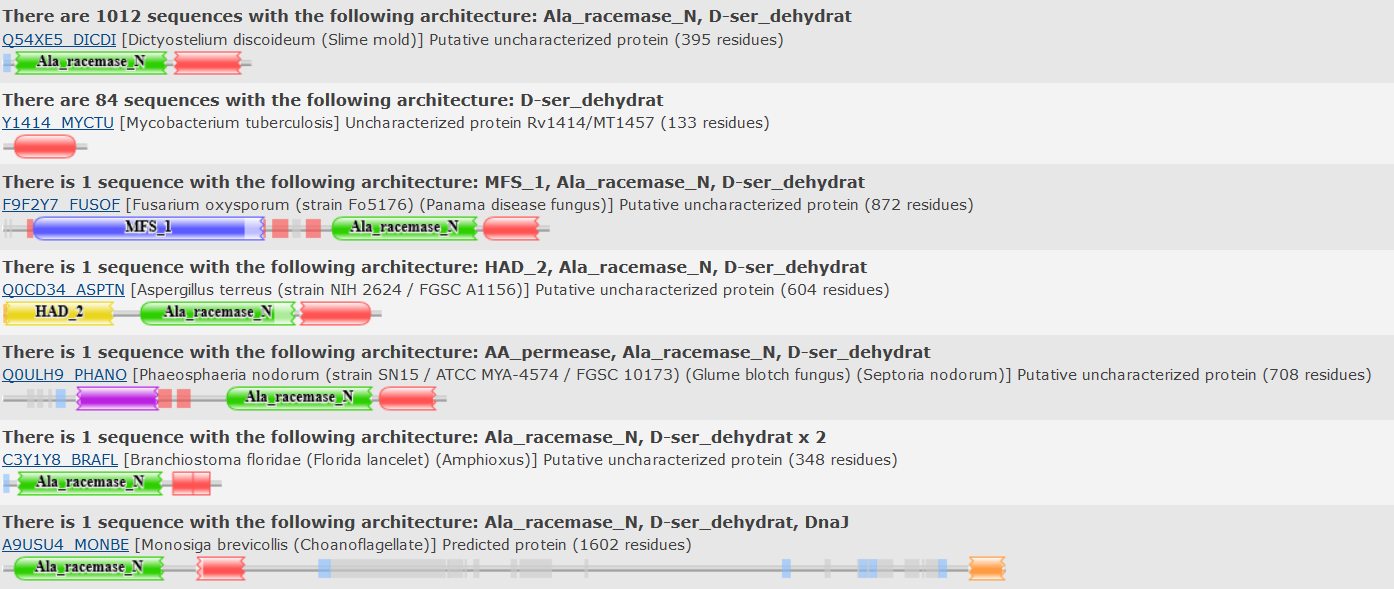
Рисунок 1. Архитектуры, образуемые доменом D-ser_dehydrat. Получено с сайта Pfam В качестве таксонов, в которых представлены домены этих белков я выбрала два царства (Bacteria, Eukaryota), общий для них таксон - Cellular organisms. Информация об архитектурах всех последовательностей, включающих выбранный домен представлена на первом листе (Domains) таблицы Excel. Данные были получены с помощью первого скрипта (swisspfam-to-xls.py), из данных по заданию. С помощью Python из этих данных были составлены доменные архитектуры для каждой последовательности (скрипт). Результат был добавлен в ту же таблицу Excel на страницу 2 (Architecture), где для каждого домена (столбцы) показана встречаемость в каждой последовательности (строки). Колонка, соответсвующая исходному домену, выделена желтым. По данным работы первого скрипта также можно посчитать длину выбранного домена в каждой последовательности (скрипт), длины представлены в таблице на третьей странице (Len), а также с помощью функции ВПР были перенесены на вторую страницу. Из базы данных Uniprot по идентификаторам были получены все последовательности, включающие домен D-ser_dehydrat. С помощью второго из данных скриптов (uniprot-to-taxonomy.py) были получены данные о таксономии этих последовательностей. Они приведены на той же странице (Architecture) таблицы Excel. Однако не для всех из них таксономия была получена, при проверке оказалось, что большинство (я проверила не для всех) неопознанных идентификаторов относится к удаленным записям Uniprot. Выравнивание всех этих последовательностей доменов можно увидеть в проекте Jalview (использовалась раскраска Clustalx). Последовательности доменов, относящихся к выбранным таксонам, фильтровались по следующим критериям: соответствие выбранной доменной архитектуре и таксону, примерное равенство длины домена в последовательности (100+-10 аминокислотных остатков). Исключение составили только несколько эукариотических последовательностей, так как их было довольно мало изначально. Выбранные по этим принципам последовательности отмечены во второй колонке (Взятые для рассмотрения) страницы Architecture основной таблицы Excel. С помощью следующего скрипта из общего выравнивания были выбраны соответствующие последовательности. С помощью Jalview были удалены наихудшие по выравниванию последовательности и с помощью MEGA было построено дерево (методом Neighbor joining). На дереве хорошо видны одинаковые последовательности, так как они объединены в неразрешенные клады. Такие дубликаты последовательностей были удалены из рассмотрения. Итоговый проект Jalview можно посмотреть здесь. В нем последовательности сгруппированы по доменным архитектурам, которые они представляют. Названия последовательностей переименованы в соответствии с архитектурой (1 для однодоменной и 2 для двудоменной) и таксоном (B - Bacteria, E - Eukaryota). Также были удалены плоховыравленные N- и C-концевые участки последовательностей. Полученное дерево представлено на рисунке 2, а скобочную форму дерева можно посмотреть здесь. 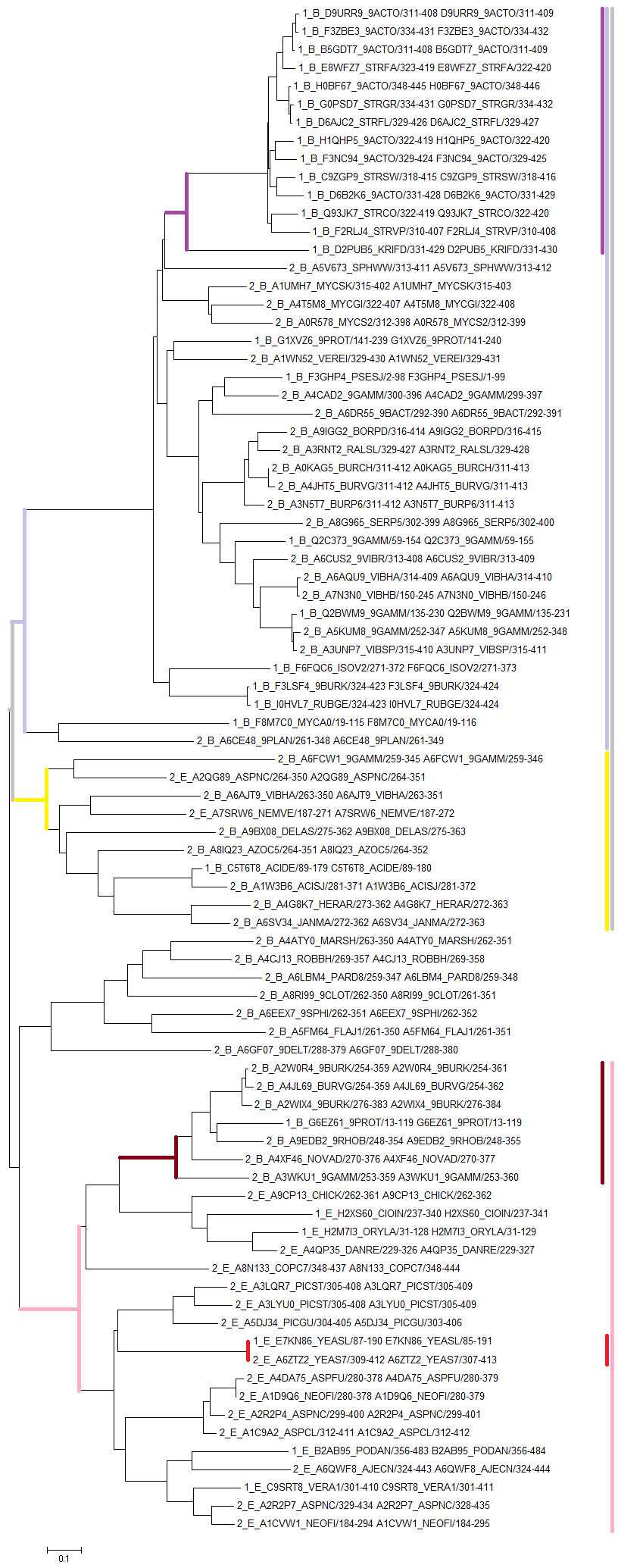
Рисунок 2. Дерево для выбранных последовательностей доменов. Получено с помощью программы MEGA методом Neighbor joining, названия последовательностей: 1 - однодоменные, 2 - двудоменные архитектуры, B - бактерии, E - эукариоты Я оставила одну неразрешенную ветвь (выделена красным цветом), в которой представлены полностью совпадающие эукариотические последовательности. Они относятся к разным штаммам пекарских дрожжей, и, кроме того, определены Pfam как относящиеся к разным доменным архитектурам. Клады полученного дерева (рисунок 2) можно разделить по таксономической принадлежности. Например, есть крупная клада (показана темно-фиолетовым цветом), в которой представлены только бактериальные последовательности. Ее можно расширить (показано серым цветом) до ветви, в которой подавляющее большинство последовательностей принадлежит бактериям, однако есть 2 эукариотические. Интересно что в этой ветви (желтый цвет) кроме одной последовательности все имеют двудоменную структуру. Розовым показана ветвь, в которой преимущественно располагаются домены эукариот, за исключением одной ветви (выделенной бордовым цветом). Не смотря на то, что последовательности разделены по ветвям преимущественно по таксономическому признаку, есть клада, показанная фиолетовым цветом, включающая только однодоменные архитектуры. Получается, что внутри этой группы существовала и эволюционно менялась только однодоменная форма белков. В качестве подсемейства последовательностей я выбрала эукариотические, так как они практически все входят в одну ветвь (розовая линия на рисунке 2). Выравнивание всех этих последовательностей ( всего 21) можно посмотреть здесь. По ним с помощью программы hmm2biuld был построен профиль последовательностей и затем он был откалиброван (программа hmm2calibrate). Результат работы этих двух программ можно увидеть здесь. По этому профилю был проведен поиск по всем белкам, включающим семейство доменов D-ser_dehydrat с помощью программы hmm2search без каких-либо дополнительных параметров. Всего было найдено 603 последовательности с E_value от 9.6 до 1.3e-13. Для определения порога, по которому стоит относить последовательности к подсемейству, была построена ROC-кривая (рисунок 3) с помощью Python и Excel. При пороге чувствительности 80% порог E_value составляет 7.5e-07, специфичность - 85%, характеристики представлены в таблице 1. 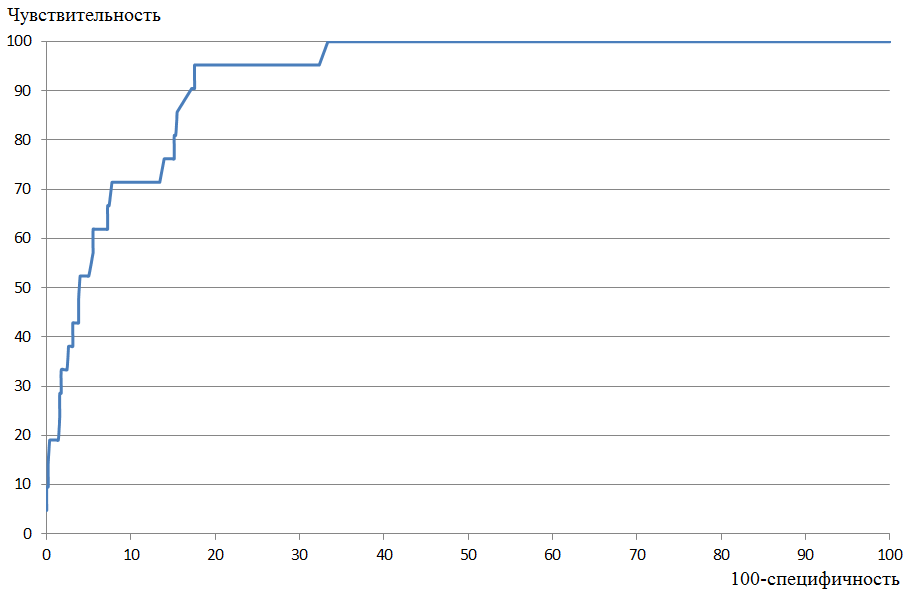
Рисунок 3. ROC-кривая. Получена с помощью Python и Excel Таблица 1.Результаты поиска по профилю при выбранном пороге 7.5e-07
|
|||||||||||||||||||||||||||
| © Pogorelskaya Sasha | Last modification date: 09.05.15 | ||||||||||||||||||||||||||||