| Учебный сайт Саши Погорельской |

|
|
||||||
| Главная | Семестры | Скрипты | Обо мне | Ссылки | ||
|
|
Поиск сигналов Заданием было найти ргуляторный мотив в промоторной области белков E. coli. Файл с этими последовательностями можно посмотреть тут. Также были даны экспериментально установленные сайты связывания белка PurR. Результат работы программы MEME представлен в этом файле. Экспериментально установленные сайты связывания белка PurR выделены синим шрифтом; курсивом выделены сайты, установленные программой MEME при запуске с параметром 1 мотив на последовательность”; жирным – с параметром “0 или 1 мотив на последовательность” (результаты запусков полностью совпадают). Максимальная и минимальная длины мотивов задавались равными 16. В отчете знаком “-” помечены последовательности, для которых мотив был найден на комплментарной цепи. Для всех промоторных областей, кроме трех, наденные MEME сайты заначительно перекрываются с экспериментальными данными (смещение на один нуклеотид из 16). Для двух из исходных последовательностей был найден мотив, но начальных данных нет. Для одной последовательности найденный мотив никак не перекрывается с экспериментом. Профиль для последовательности Шайн-Дальгарно Для работы я скачала полную последовательность ДНК бактерии Syntrophus aciditrophicus SB из геномного бразуреза NCBI. По таблице я координаты начала первых 150 генов и составила координаты последовательностей, предположительно включающих последовательности Шайн-Дальгарно (от -34 до 1). Затем, с помощью программы seqret пакета Emboss были получены сами последовательности:
Для поиска мотива использовалась программа meme, построенный лого можно посмотреть на рисунке 1. Вывод в формате .html можно посмотреть здесь для meme и здесь - для mast. 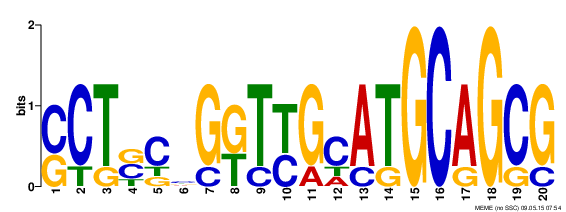
Рисунок 1. Лого, простроенное программой meme по последовательностям 35 нуклеотиддов перед 100 генами |
|||||||||||||
| © Pogorelskaya Sasha | Last modification date: 09.05.15 | ||||||||||||||