
Рисунок 1. Выдача PDBeFOLD.
В данном задании был дан файл PDB с приблизительными координатами какого-то белка. Попробуем определить, что это за белок. Для этого воспользуемся PDBeFOLD. Первые 13 наиболее близких находок PDBeFOLD представляют фосфатазы PTEN в различных состояниях. Исходя из этого можно предположить, что и в нашем файле находятся координаты фосфатазы PTEN.

Рисунок 1. Выдача PDBeFOLD.
Фосфатаза представляет собой продукт гена PTEN. Она обладает двойной специфичностью, субстратами данной фосфатазы могут выступать и фосфорилированные тирозины/серины в белке, и фосфорилированные формы фосфатидилинозитола(Рис 2.1, Рис 2.2). PTEN является важным онкосупрессором. При развитии многих раковых опухолей ген PTEN оказывается мутирован.[1]
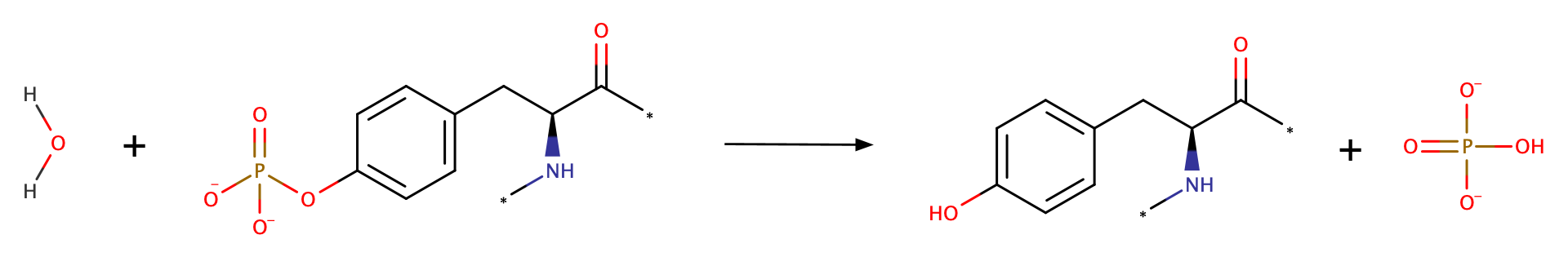
Рисунок 2.1 Катализируемая PTEN реакция отщепления фосфатной группы от фосфорилированного тирозина в белке.
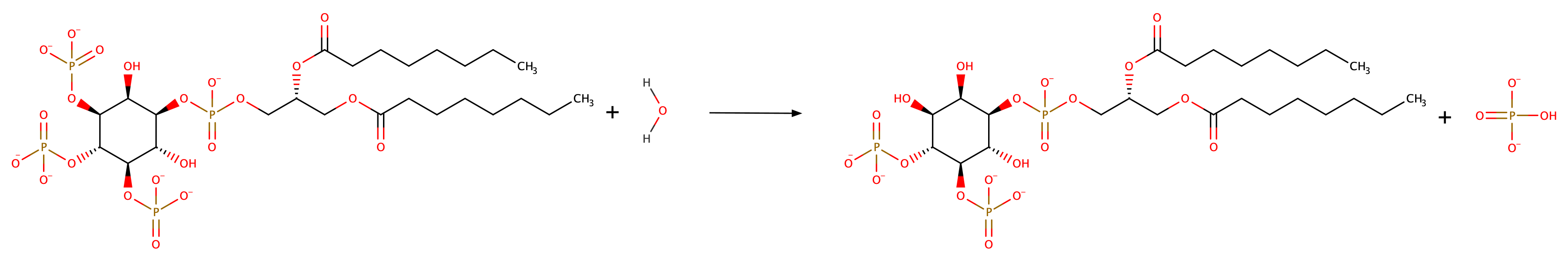
Рисунок 2.2 Катализируемая PTEN реакция отщепления фосфатной группы от фосфатидилинозитол-3,4,5-трифосфата(PI(3,4,5)P3).
Рассмотрим строение белка и расположение активного центра.
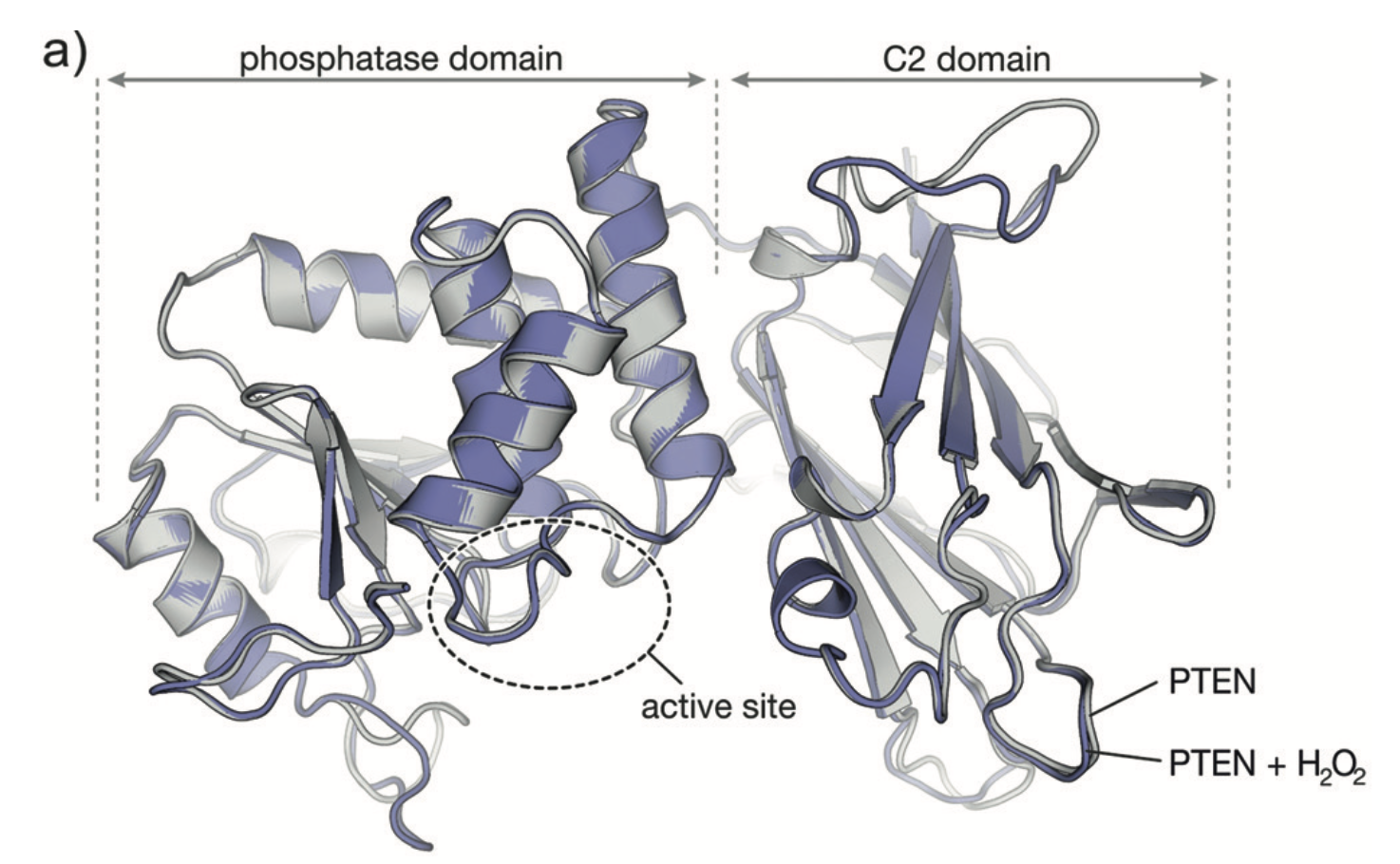
Рисунок 3. Общее устройство фосфатазы PTEN. Выделен фосфатазный домен(слева) и C2 домен(справа). Пунктиром обведен активный центр.[2]
PTEN состоит из двух доменов, которые достаточно легко визуально выделяются: фосфатазного домена и C2 домена(Рис. 3).
C2 домен участвует в образовании контакта с мембраной. Однако, в отличие от аналогичных C2 доменов в других белках, он не требует Ca2+. Вместо этого, как предполагается, в образовании контакта могут участвовать направленные в раствор и способные нести положительный заряд лизины(260, 263, 266, 267, 269), а так же гидрофобные 264-й метионин и 265-й лейцин.
Фосфатазный домен содержит мотив HCXXGXXR(остатки 123-130), образующий P-петлю(Рис. 4). В данной фосфатазе мотив несколько больше, чем в других аналогичых фосфатазах, обладающих двойной специфичностью(напрмер в VHR). Как предполагается, карман большего размера необходим, чтобы связвать достаточно большой субстрат(фосфорилированные формы фосфатидилинозитола).
Два домена соединены консервативными остатками, мутации которых часто происходят при развитии раковых опухолей.[1]
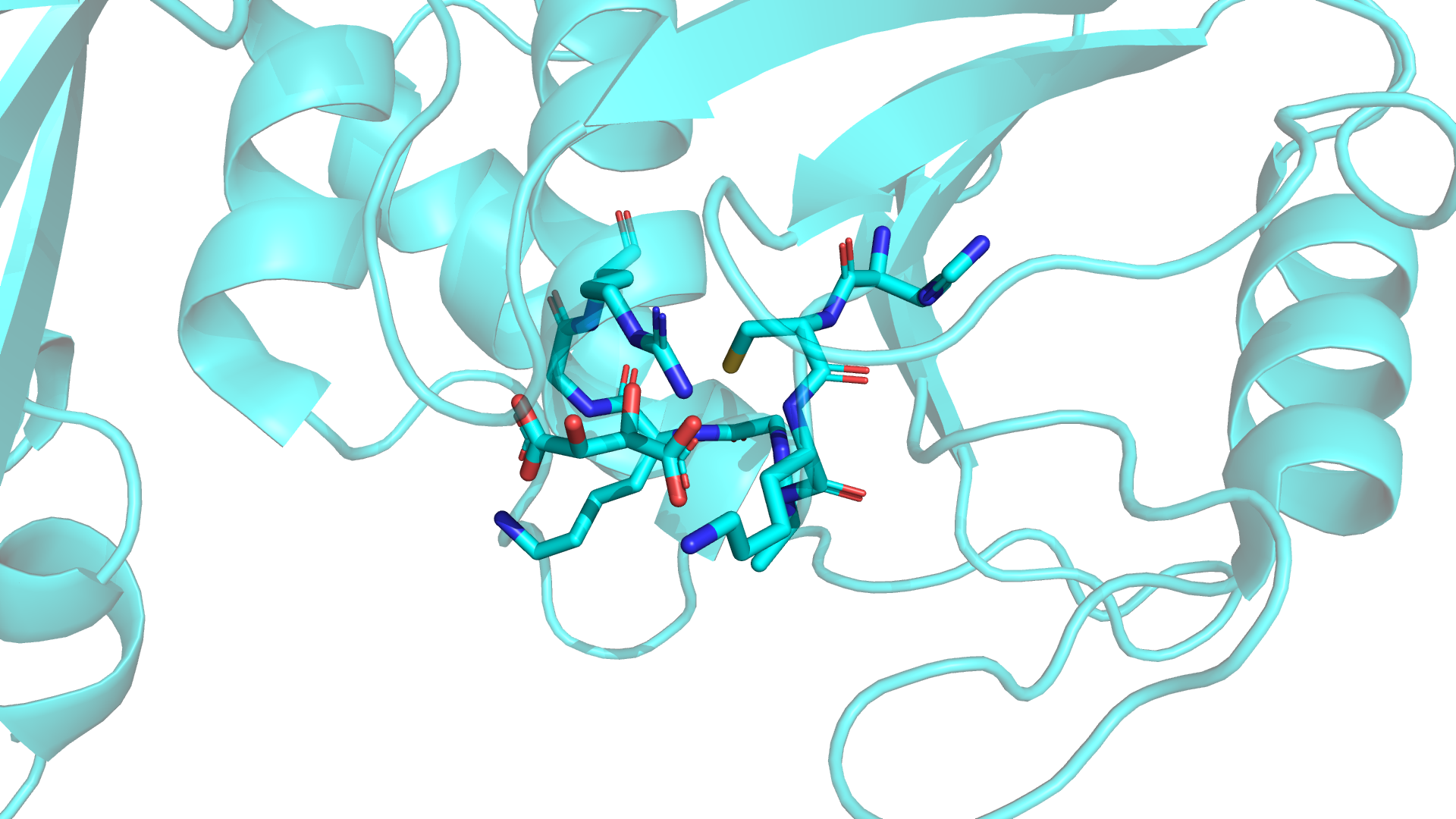
Рисунок 4. P-петля в структуре PTEN(показа sticks). L-винная кислота показана с помощью sticks. PDBID=1D5R.
Во многих структурах фосфатазы PTEN присутствует L-винная кислота. Это связано с тем, что винная кислота досточно хорошо воспроизводит положение субстрата в активном центре. Расстояние между углеродами карбоксильных групп довольно близко расстоянию между атомами фосфора в фосфатах субстрата(4.3 Å и 4.1 Å соответсвенно).
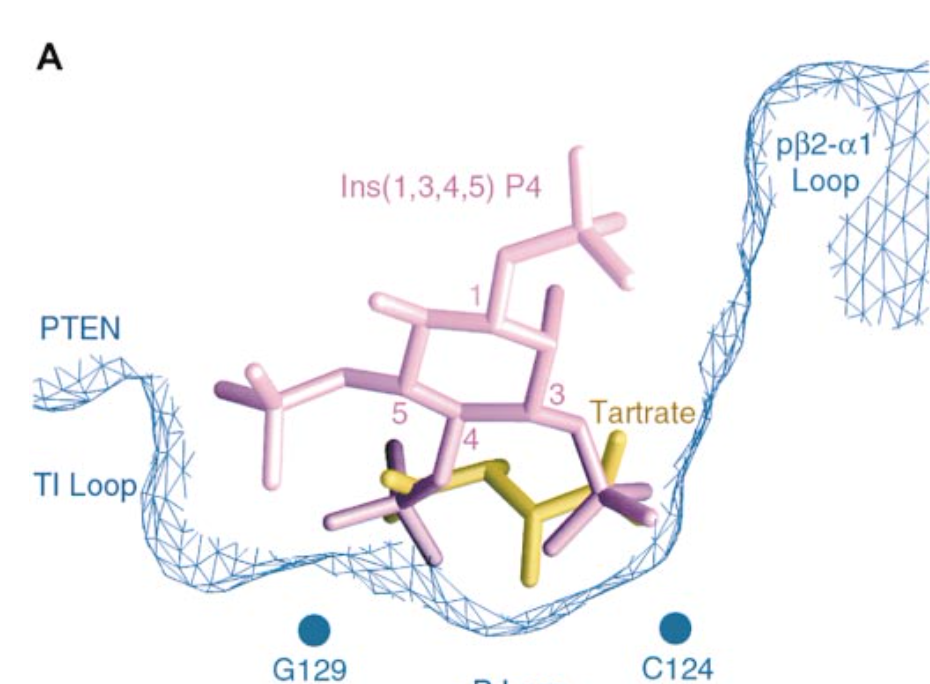
Рисунок 5. Сопоставление PI(1,3,4,5)P4 и L-винной кислоты в активном центре PTEN[1].
Кроме того, данный лиганд образует водородные связи с остовом и с боковыми радикалами Cys-124 и Arg-130 активного сайта.
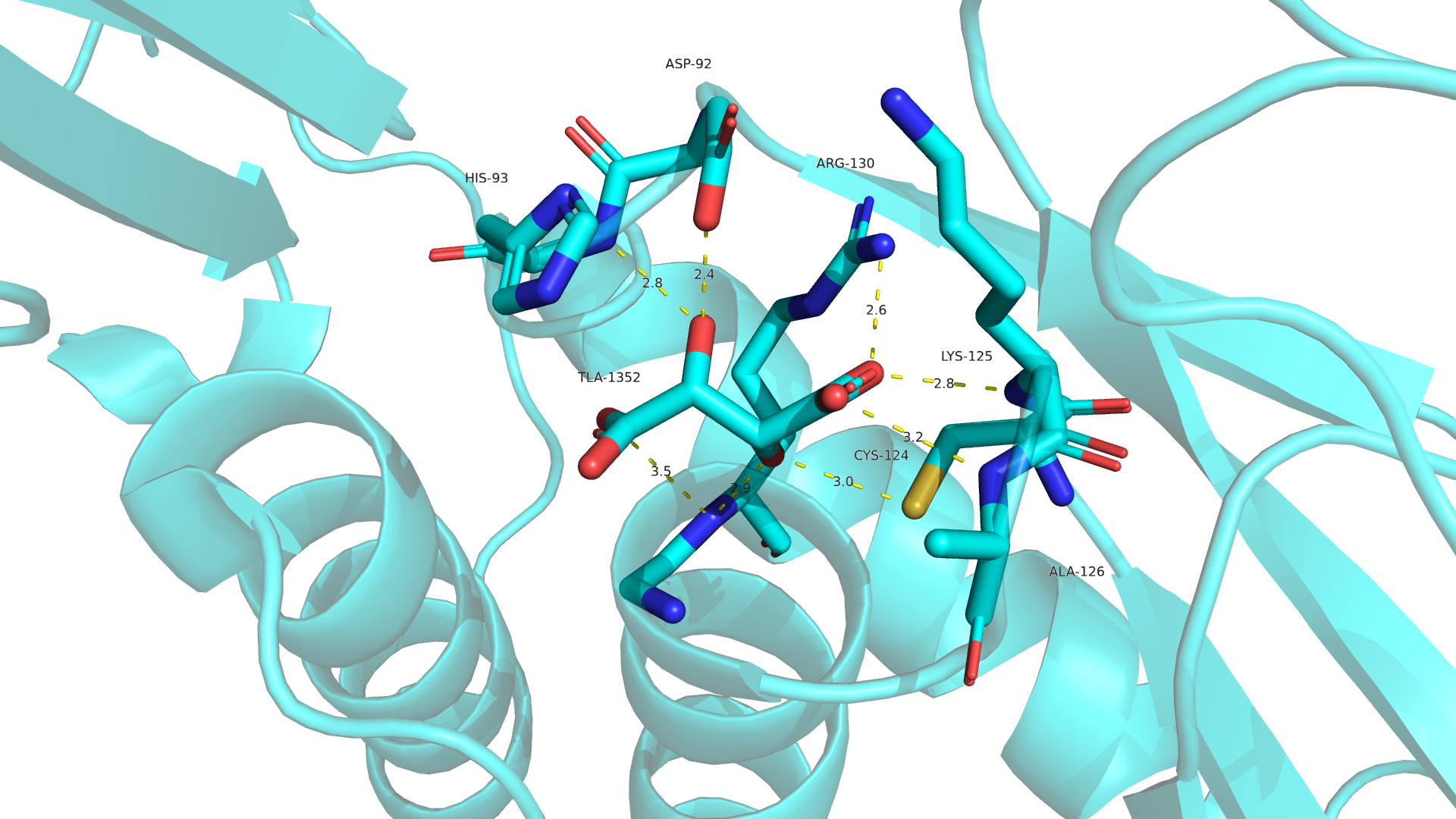
Рисунок 6. Взаимодействия L-винной кислоты с ферментом. PDBID=1D5R
Рассмотрим механизм действия фермента.
Механизм PTEN напоминает таковой у белковой тирозин фосфатазы 1B. Cys124 выступает нуклеофилом, принимая на себя фосфатную группу субстрата. Asp92 отдает протон на кислород в гидроксиле уходящей группы(субстрата). Arg130 стабилизирует переходное состояние. Затем фосфатная группа переносится на воду, Asp92 возврашает себе протон, и фермент возвращется в исходное состояние.[1]

Рисунок 7. Остатки, непосредственно вовлеченные в катализируемую реакцию.PDBID=1D5R
В выдаче PDBeFOLD помимо восстановленных структур PTEN встречаются структуры, окисленные перекисью водорода, и структуры после взаимодействия с биспероксованадием. Рассмотрим их подробнее.
В качестве окисленной формы будем рассматривать структуру с PDBID=5BUG, в качестве структуры после взаимодействия с биспероксованадием возьмем PDBID=5BZX.
Взаимодействие и с перекисью, и с биспероксованадием инактивирует фосфатазу. Как оказалось, в каждом из случаев важный для работы фермента Cys124 образует дисульфидную связь с Cys71(Рис 8.1, Рис 8.2)[2].
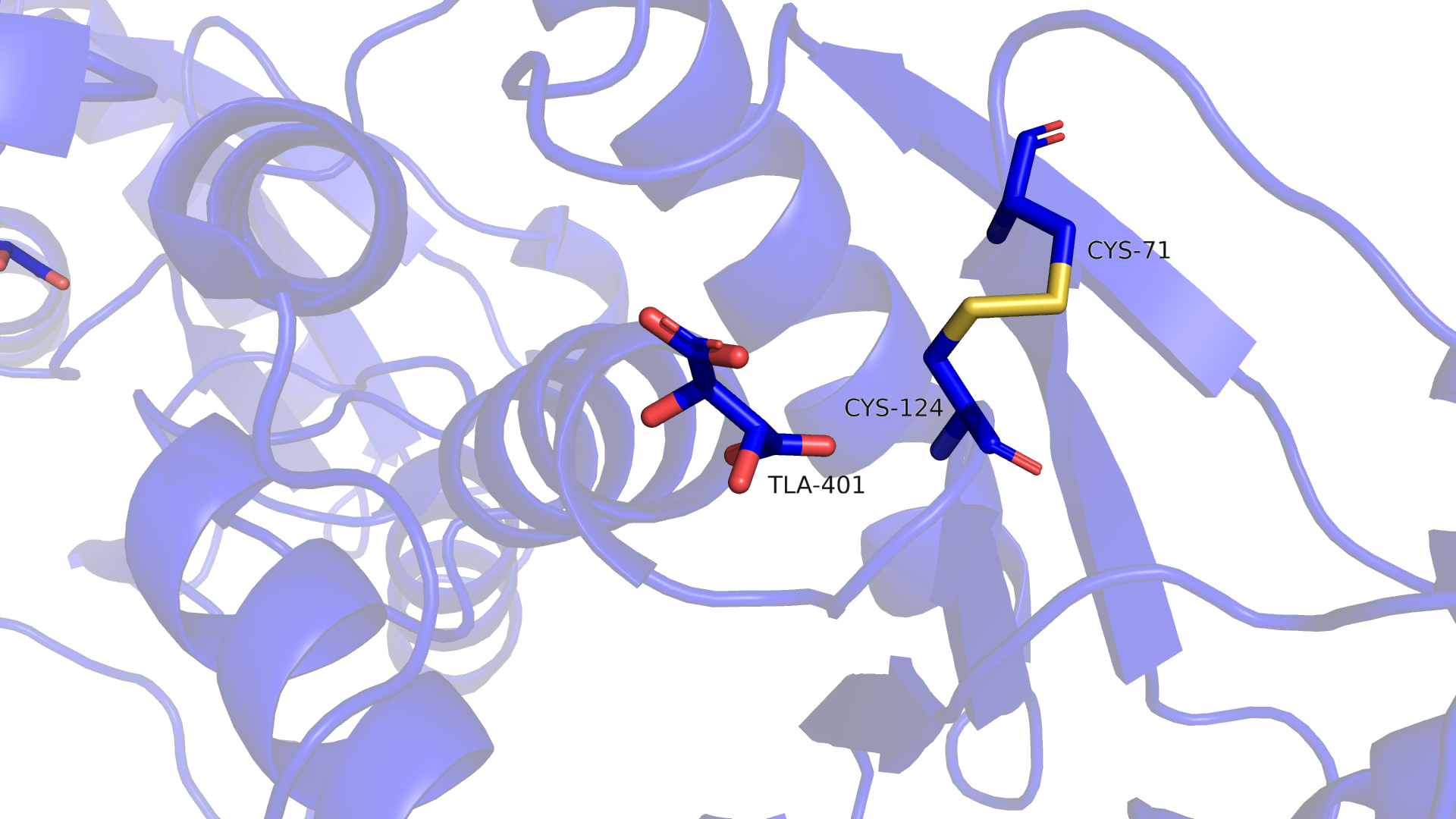
Рисунок 8.1 Дисульфидная связь Cys124-Cys71 в структуре PTEN, окисленной перекисью водорода.PDBID=5BUG
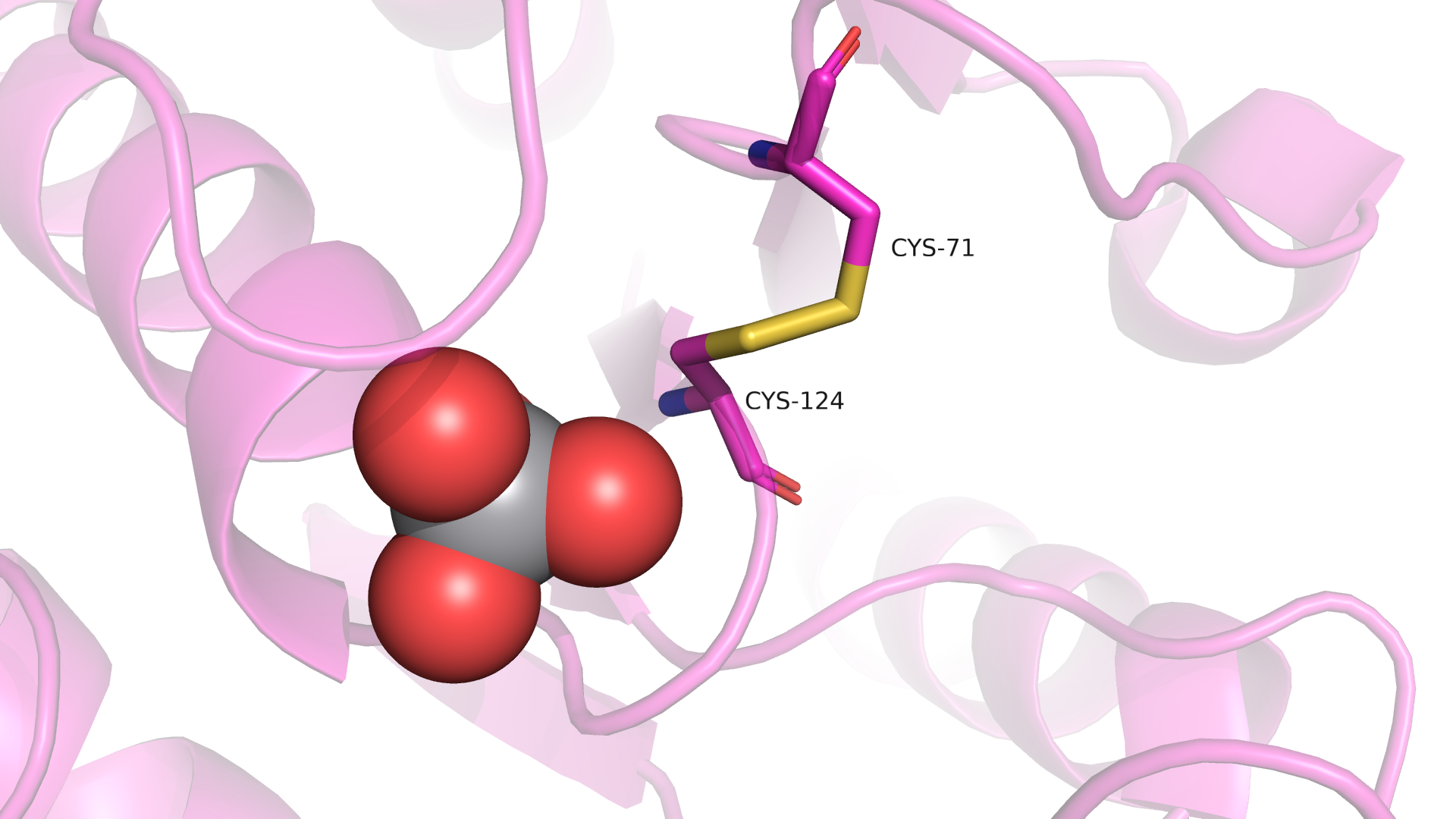
Рисунок 8.2 Дисульфидная связь Cys124-Cys71 в структуре PTEN после обработки биспероксованадием. PDBID=5BZX
Стоит отметить, что в каждом из приведенных случаев происходит обратимое ингибирование. Благодаря этому биспероксованадий может быть рассмотрен в качестве нейропротектора и кардиопротектора, поскольку необратимое ингибирование важного онкосупрессора PTEN крайне нежелательно[2].
Теперь обратимся к CATH, чтобы еще раз уточнить доменную организацию фермента и узнать место доменов в иерархии CATH. Будем рассматривать структуру с PDBID=1D5R.
CATH выделяет в структуре два домена, рассмотрим их. Первый домен называется 1d5rA01(Рис. 9.2), он входит в функциональное семейство фосфатидилинозитол-3,4,5-трифосфат 3-фосфатазы и фосфатазы белков двойной специфичности PTEN, суперсемейство(группа гомологии) тирозин фосфатазы белков(CATH: 3.90.190.10). Полная иерархия(класс, архитектура, топология) представлена на Рис. 9.1.

Рисунок 9.1 Место домена 1d5rA01 в иерархии CATH.
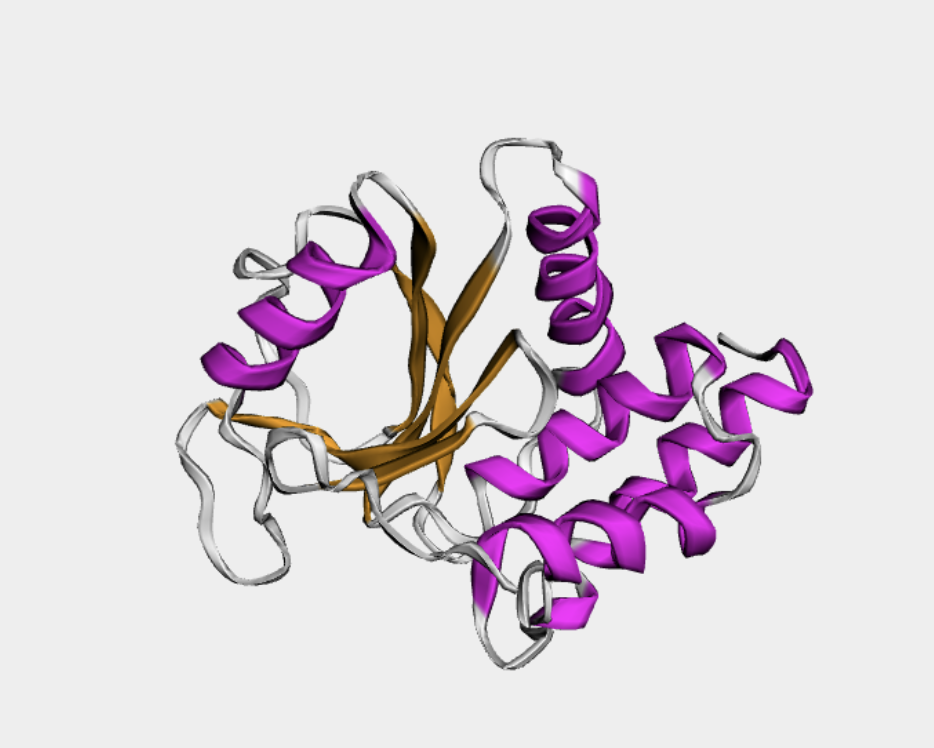
Рисунок 9.2 Структура домена 1d5rA01, цветами отображены типы вторичной структуры.
Далее рассмотрим второй домен. Он называется 1d5rA02(Рис. 10.2). Он входит в то же функциональное семейство, что и первый домен. Однако его суперсемейство(группа гомологии) CATH 2.60.40.1110. Место домена в полной иерархии CATH можно увидеть на Рис. 10.2.

Рисунок 10.1 Место домена 1d5rA02 в иерархии CATH.

Рисунок 10.2 Структура домена 1d5rA02, цветами отображены типы вторичной структуры.
В целом, в начале было произведено правильное разбиение фермента на два домена: фосфатазный домен(определенный CATH, как 1d5rA01) и домен C2(d5rA02). Функции данных доменов, указанные в начале, тоже соотносятся с информацией из CATH.
В данном задании найдем в PDBeFOLD два белка, имеющих не более 35% идентичности по последовательности с 1D5R и выполняющих другую функцию.
Lowest acceptable match был выставлен на 55/55.
Были обнаружены белки, имеющие менее 35% идентичности по последовательности: ауксилин(PDBID=3N0A) и растительная фосфатаза(PDBID=3NME). Процент идентичности по последовательности для них составил 28% и 13% соответственно.
Далее рассмотрим каждый из найденных белков.
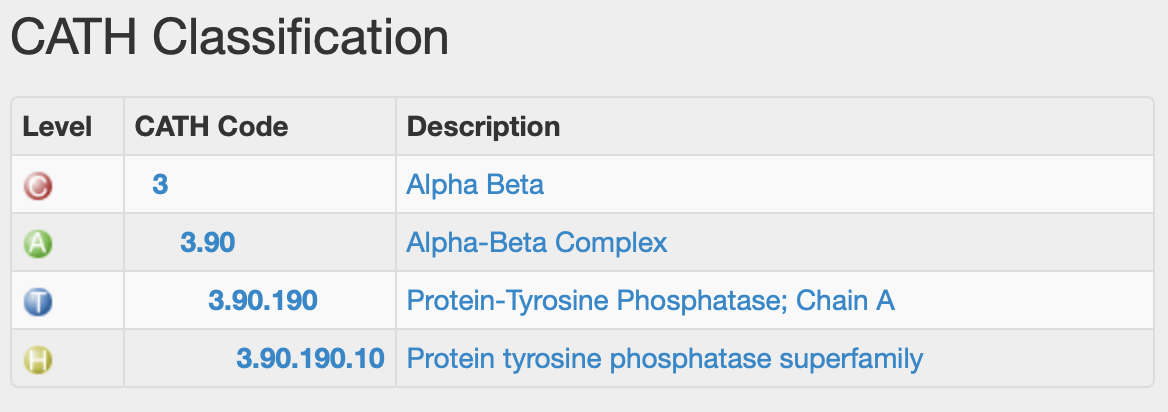
Рисунок 11.1 Место домена 3n0aA01 в иерархии CATH.
Теперь рассмотрим второй домен.
3n0aA02 входит в функциональное семейство "изоформа X2-ассоциированной киназы циклин-G", суперсемейство(группа гомологии) CATH: 2.60.40.1110 . Полная иерархия(класс, архитектура, топология) представлена на Рис. 11.2. Класс, архитектура, топология и группа гомологии совпадают с C2 доменом PTEN(PDBID=1D5R).
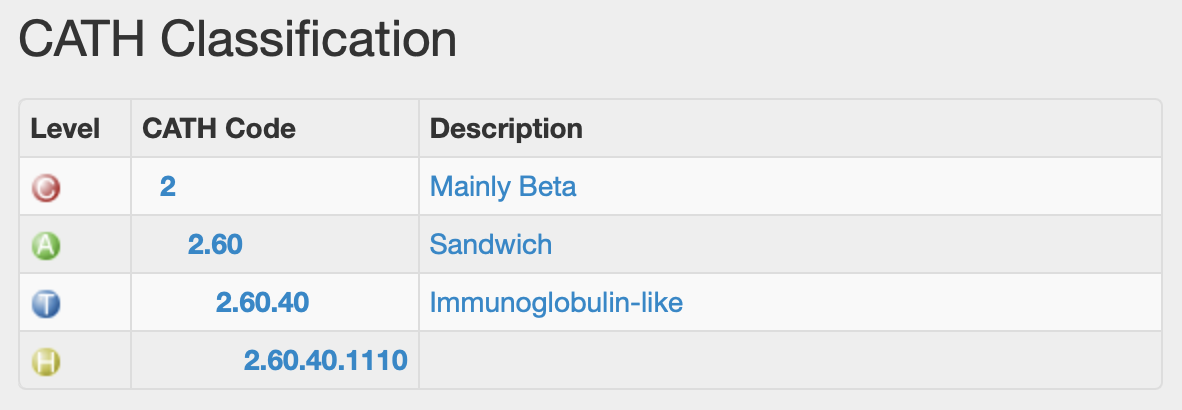
Рисунок 11.2 Место домена 3n0aA02 в иерархии CATH.
Сравним структуры 1D5R и 3N0A. Воспользуемся функцией super в PyMOL.

Рисунок 12. Сравнение структур 1D5R(розовая) и 3N0A(голубая).
Прежде всего, ауксилин 3N0A несколько длиннее PTEN 1DR5. Укладка альфа-спиралей и бета-листов в целом оказывается достаточно похожей, однако некоторые отличия наблюдаются и в ней(некоторые участки на месте петель PTEN в ауксилине свернуты в бета-листы и альфа-спирали).
Известно, что ауксилин хорошо связывает PI(1,3,4,5)P4, однако лишен фосфатазной активности. Рассмотрим отличия в остатках активного центра.
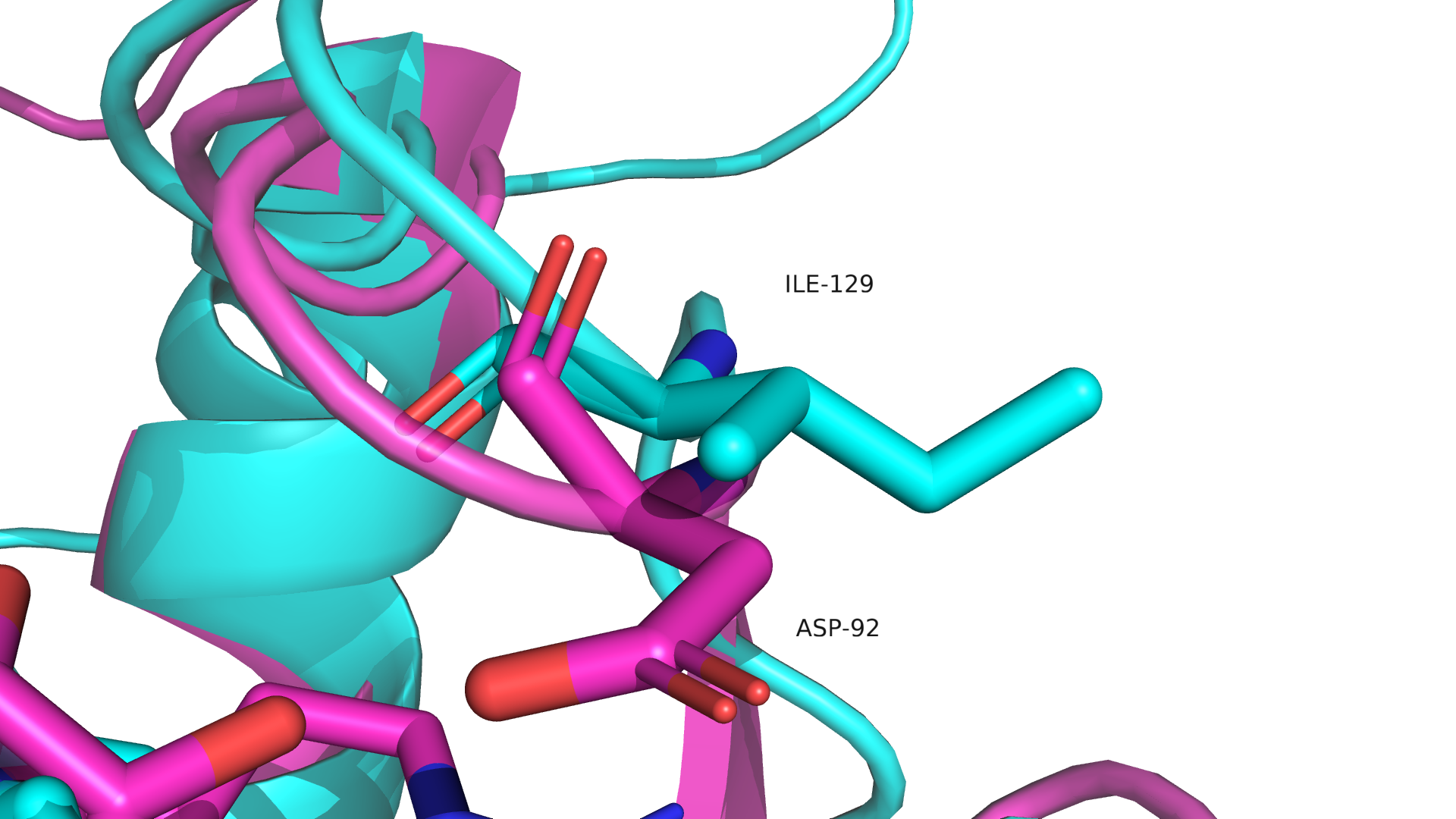
Рисунок 13.1 Сравнение фрагмента активного структур 1D5R(розовая) и 3N0A(голубая).
На месте Asp92, одного из трех главных остатков, отвечающих за фосфатазную активность, в структуре 3N0A находится Ile129(Рис 13.1). Как мне кажется, это отличие играет одну из главных ролей в утрате ауксилином фосфатазной активности. Однако данный белок все таки может связывать субстрат PTEN. Рассмотрим P-петлю подробнее.
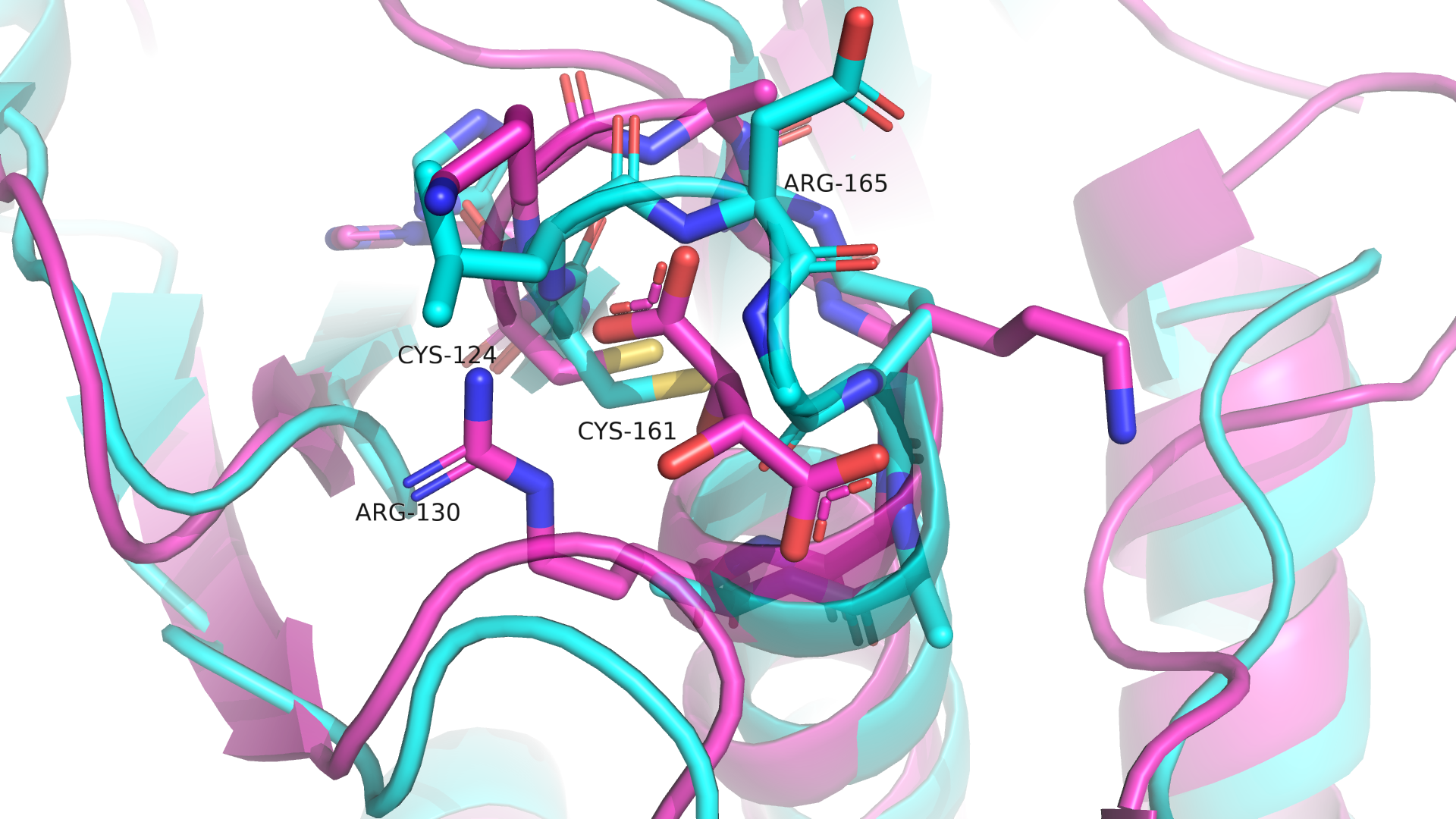
Рисунок 13.2 Сравнение P-петли структур 1D5R(розовая) и 3N0A(голубая).
На Рис. 13.2 видно, что петля в структуре 3N0A уложена гораздо плотнее, чем в PTEN. В образуемом ей кармане недостаточно места, чтобы аналог субстрата расположился так же, как и в структуре 1D5R. Важные для фосфатазной активности цистеины Cys124(1D5R) и Cys161(3N0A) расположены в одном месте и ориентированы одинаково. Однако аргинины(необходимые для поддержания переходного состояния)в этих двух структурах находятся в разных местах петли и ориентированы в разные стороны. Кроме того, аминокислотный состав петель в этих структурах значительно отличается.
Крахмал представляет собой нерастворимый в воде полимер, требующий согласованной активности киназ и фосфатаз для солюбилизации внешней поверхности глюкана и катаболизма. Все известные геномы растений содержат ген глюкановой фосфатазы Starch Excess4 (SEX4). SEX4 может дефосфорилировать как поверхность гранул крахмала, так и растворимые фосфоглюканы. SEX4 имеет домен DSP, обладающий тирозин/серин/треонин белковой и углеводной фосфатазной активностью. Активный сайт представляет собой PTP-петлю(остатки 197-205) с последоваетльностью HCTAGMGRA.[5]
Стоит отметить, что структура 3NME была сделана каталитически неактивной путем замены Cys198 на Ser198. В активном центре структуры связан фосфат.
Обратимся к CATH. Для структуры с PDBID=3NME обнаруживаются по два домена для каждой из цепей(здесь и в дальнейшем будет рассматривать цепь B, поскольку именно она оказалась в выдаче PDBeFOLD): 3nmeB01 и 3nmeB02.
3nmeB01 входит в функциональное семейство "Фосфоглюканфосфатаза DSP4 хлоропласта", суперсемейство(группа гомологии) тирозин фосфатазы белков(CATH: 3.90.190.10). Полная иерархия(класс, архитектура, топология) представлена на Рис. 14.1. Класс, архитектура, топология и группа гомологии совпадают с фосфатазным доменом PTEN(PDBID=1D5R).3

Рисунок 14.1 Место домена 3nmeB01 в иерархии CATH.
Рассмотрим второй домен.
3nme02 входит в функциональное семейство "Фосфоглюканфосфатаза DSP4 хлоропласта", суперсемейство(группа гомологии) иммуноглобулины(CATH:2.60.40.10) . Полная иерархия(класс, архитектура, топология) представлена на Рис. 14.2. Класс, архитектура, топология с C2 доменом PTEN(PDBID=1D5R).

Рисунок 14.2 Место домена 3nmeB02 в иерархии CATH.
Рассмотрим отличия структур 1D5R и 3NME.
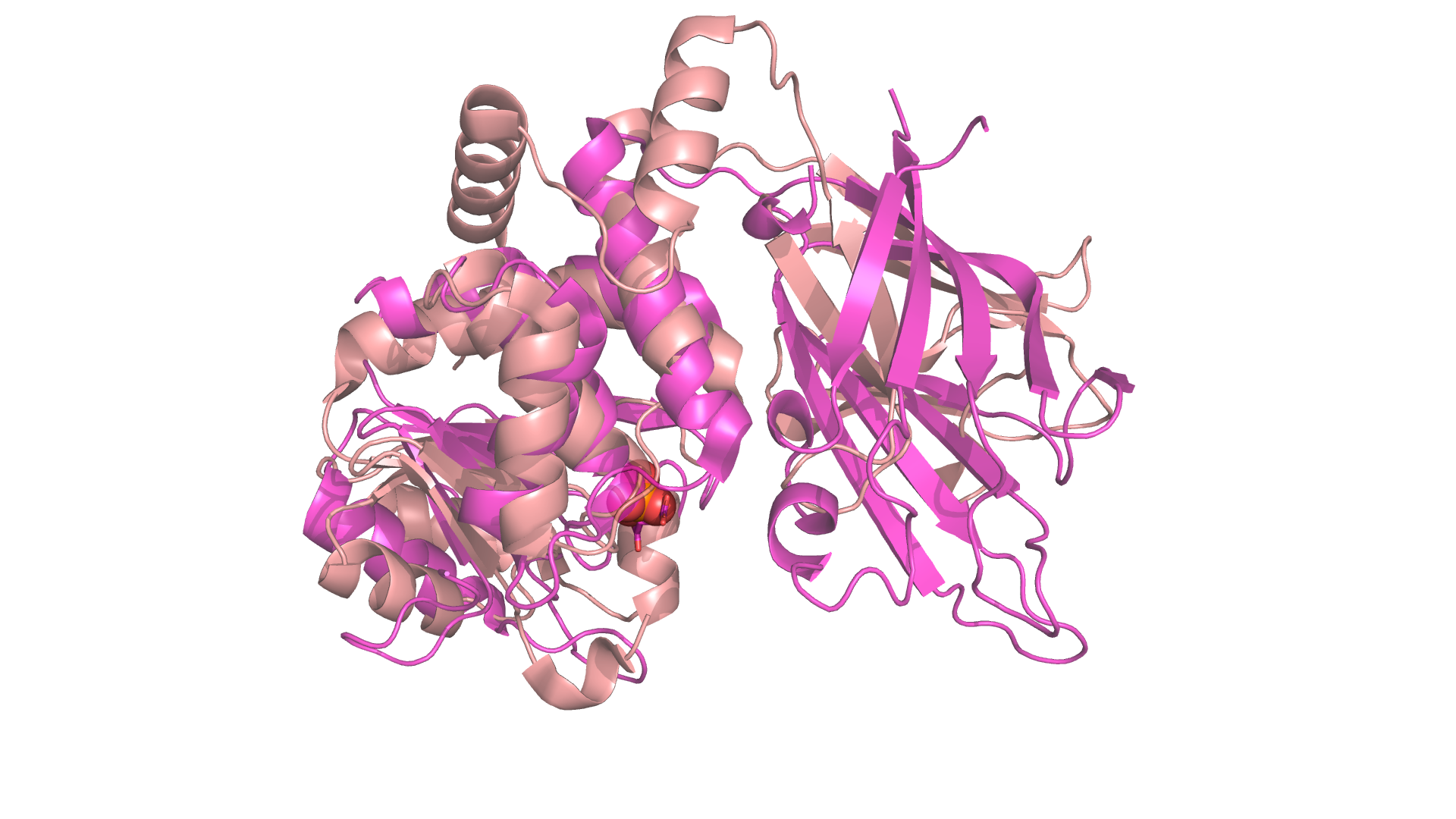
Рисунок 15. Сравнение структур 1D5R(розовая) и 3NME(бежевая).
Видно, что второй домен 3NME лег на структуру 1D5R хуже, чем DSP(на Рис. 15 слева). В целом структуры оказались менее похожи, чем 1D5R и 3N0A, что вполне можно объяснить низким процентом идентичности по последовательности(13%). Рассмотрим более детально активный центр.
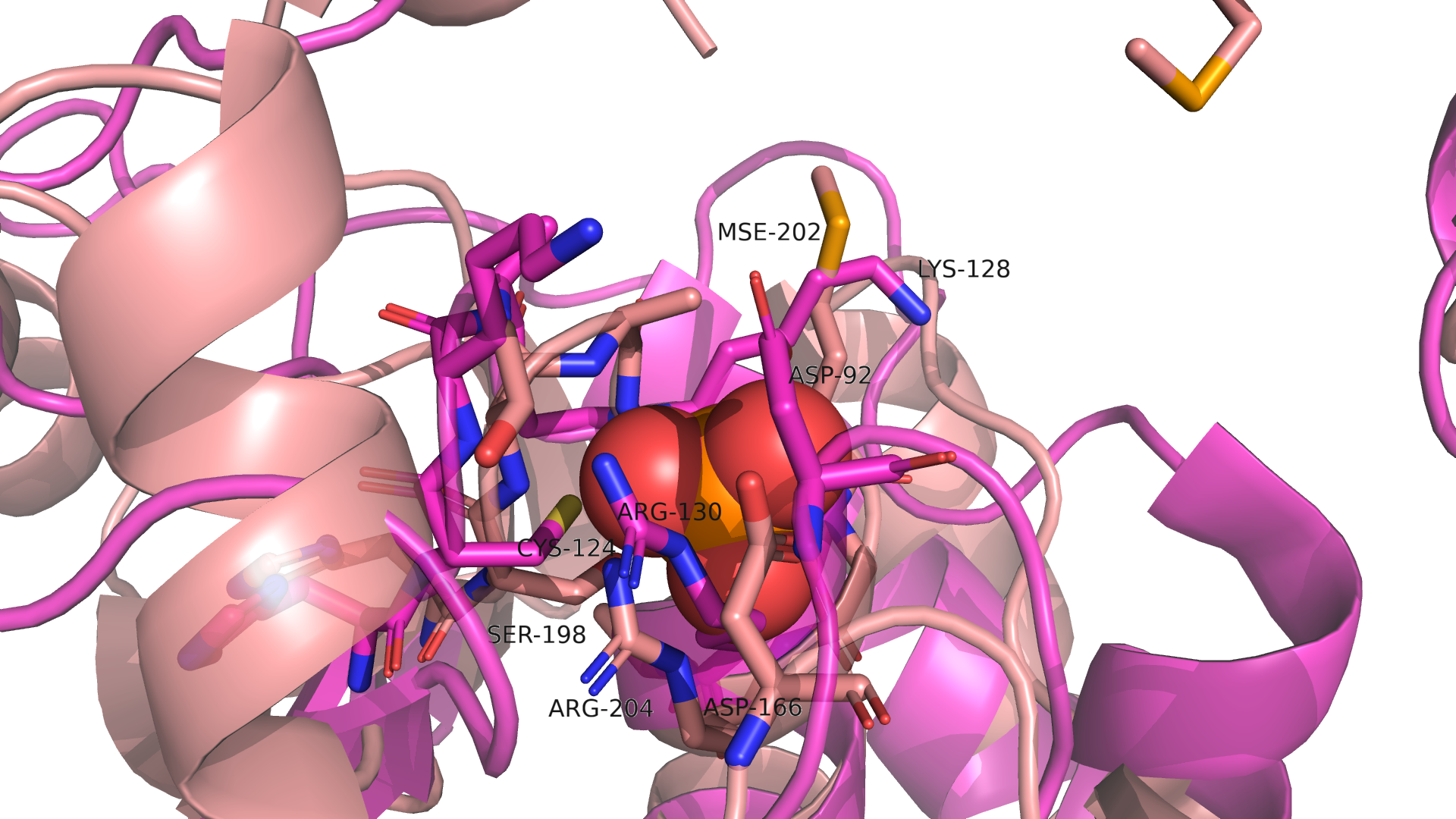
Рисунок 16. Сравнение P-петли структур 1D5R(розовая) и PTP-петли 3NME(бежевая).
PTP-петля 3NME, как и в случае с петлей 3N0A, образует карман значительно меньшего размера, чем P-петля PTEN. Каталитические Cys, Asp, Arg присутствуют в структуре 3N0A. Более того, они ориентированы крайне похожим образом. Однако есть и различия: в структуре 3MNE на месте Lys128 находится Mse202(в природном белке там метионин, селенометионин был использован для решения фазовой проблемы), на месте Lys125 находится Thr199.
Попробуем выделить функционалный мотив, отвечающий за функцию, общую для всех белков.
Как мне кажется, общим для трех белков является распознование и связывание с фосфорилированным субстратом. Для этого используется P или же PTP петля в фосфатазном домене и ее окружение. Наблюдается общность укладки фосфатазного домена у всех трех белков: бета-лист, окруженный альфа-спиралями(Рис 12, Рис 15). Каталитический центр(центр связывания) всех трех белков располагается в одинаковом месте в фосфатазном домене(отмечен пунктиром на Рис. 3).
Помимо этого, у фосфатазы PTEN и ауксилина присутствуют сходные домены C2, необходимые для контакта с мембраной(Рис 12).
Рассмотрим различия в функциях белков.
Фосфатаза PTEN единственная из трех способна и связывать, и отщеплять фосфатную группу от PI(1,3,4,5)P4. Это связано в первую очередь с наличием всех трех аминокислотных остатков(Cys, Asp, Arg), необходимых для катализа. Помимо это, сайт связывания фосфатазы PTEN имеет наибольшие размеры среди трех рассмотренных белков, что может оказаться принципиально при работе с достаточно крупным субстратом. Наконец, субстрат в активном центре поддерживается взаимодействиями с окружением(Рис. 6).
Ауксилин способен связывать PI(1,3,4,5)P4, но не способен отщеплять от него фосфатную группу. Основной причиной этого является то, что на месте Asp92, одного из трех главных остатков, отвечающих за фосфатазную активность, в структуре 3N0A находится Ile129(Рис 13.1).
Растительная фосфатаза обладает тирозин/серин/треонин белковой и углеводной фосфатазной активностью, поскольку имеет в реакционном центре остатки Cys, Asp, Arg, расположенные сходным образом с фосфатазой PTEN. Как мне кажется, меньшие размеры кармана связывания субстрата и отсутствие некоторых важных для удержания субстрата в кармане остатков не позволяет проявлять растительной фосфатазе активность по отношению к PI(1,3,4,5)P4.
1.Lee J. O. et al. Crystal structure of the PTEN tumor suppressor: implications for its phosphoinositide phosphatase activity and membrane association //Cell. – 1999. – Т. 99. – №. 3. – С. 323-334.
2.Lee C. U. et al. Redox modulation of PTEN phosphatase activity by hydrogen peroxide and bisperoxidovanadium complexes //Angewandte Chemie International Edition. – 2015. – Т. 54. – №. 46. – С. 13796-13800.
3.Ungewickell E. et al. Role of auxilin in uncoating clathrin-coated vesicles //Nature. – 1995. – Т. 378. – №. 6557. – С. 632-635.
4.Guan R. et al. Structure of the PTEN-like region of auxilin, a detector of clathrin-coated vesicle budding //Structure. – 2010. – Т. 18. – №. 9. – С. 1191-1198.
5.Vander Kooi C. W. et al. Structural basis for the glucan phosphatase activity of Starch Excess4 //Proceedings of the National Academy of Sciences. – 2010. – Т. 107. – №. 35. – С. 15379-15384.
© Simon Konnov 2017-2021