Предсказание генов прокариот
Задание 1
Исходная плазмида принадлежит бактерии Lactobacillus plantarum CP015858 .
L. plantarum относится к грамположительным анаэробными неспорообразующим молочнокислым бактериям. Особенность этих бактерий - превращение углеводов в молочную кислоту. L. plantarum часто встречается в таких продуктах, как квашеная капуста, соленые огурцы, оливки в рассоле, в некоторых сырах. [1]
L. plantarum вырабатывают антимикробные вещества и экзополисахариды, благодаря чему может существовать в желудочно-кишечном тракте человека. Этот факт позволяет использовать L. plantarum в качестве доставщика лекарственных препаратов в желудочно-кишечный тракт. [2]
Получение предсказания генов с помощью Prodigal
Сначала последовательность плазмиды была получена в форматах gb и gff с помощью средств пакета EMBOSS. Кроме того, был получен файл с кодирующими последовательностями:
seqret genbank::genbank:CP015858 seq.gb featcopy seq.gb seq1.gff extractfeat seq.gb -type CDS -describe product СDS.fasta
Далее была использована прогграммма Prodigal (Prokaryotic Dynamic Programming Genefinding Algorithm), которая предназначена для предсказания генов прокариот. Были использованы следующие параметры:
prodigal.windows.exe -i seq.gb -c -f sco -o seq_out.sco
В результате был получен файл seq_out.sco. Результаты программы Prodigal оказались немного отличными от данных о генах, указанных в Genbank.
Обработка результатов
Для сравнения полученных данных были написаны несколько скриптов на Python: 1.py, 2.py.
Первый скрипт переводит файл из формалта .gff в .sco для удобства в дальнейшей работе. С помощью него был получен файл seq_inp.sco.
Второй скрипт оценивает показатели, приведенные ниже. В нем создается класс gene с основными параметрами из файла .sco( номер, координата начала, координата конца, ориентация).
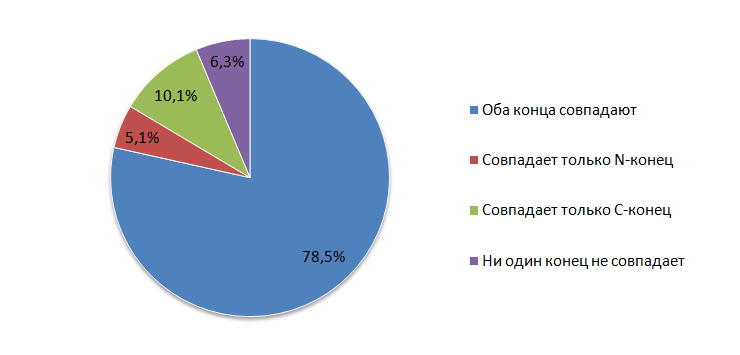
Кроме того, создается несколько функций:Далее на вход подаются два файда в формате .sco - анноотации генов из Genbank и Prodigal. Последовательно проверяется выполнение функций isf, isn, isс. Причем если функция выполняется то к соотстветствующей переменной прибавляется единица. Таким образом были подсчитаны параметры, представленные в табл.1 и рис.2:
| Параметр сравнения исходного файла (Genbank) с результатами Prodigal | Количество (процент об общего чила генов) |
| Начало и конец гена совпадает | 62 (78,5%) |
| Только N-конец белка совпадает | 4 (6,3%) |
| Только C-конец белка совпадает | 8 (10,1%) |
| Оба конца не совпадают | 5(6,3%) |
Рассмотрение конкретных случаев
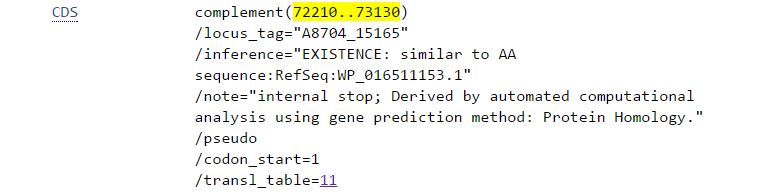
Рассмотрим ген, координаты которого в Genbankе - 72210:73130(-). По результатам Prodigal в данной позиции находятся 2 гена с координатами 72210:72515(-) и 72573:73130(-). Если в первом предсказанном гене с аннотацией Genbank совпадает C-конец белка, то во втором - N-конец.
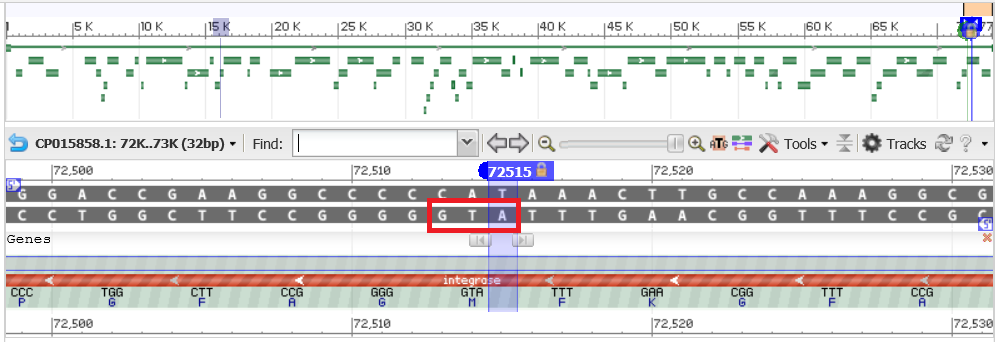
Рассмотрим подробнее первый предсказанный ген(координаты 72210:72515(-)). В геномном браузере на сайте NCBI в позициях 72523-72515 расположен кодон TGA, который является старт-кодоном на обратной цепи (рис.3). Однако в аннтации Genbank этот кодон не является началом трансляции белка.
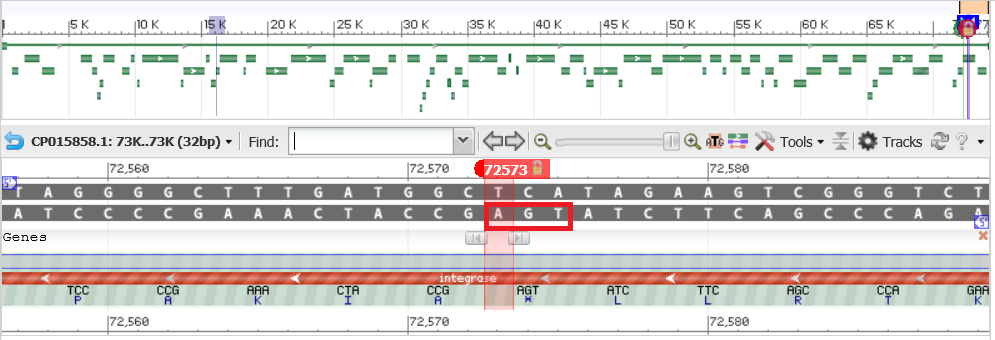
Теперь рассмотрим второй предсказанный ген(координаты 72573:73130(-). В геномном браузере на сайте NCBI в позициях 72573-72574 расположен кодон AGT, который является стjg-кодоном на обратной цепи (рис.4). Несмотря на то, что в геномном браузере он помечен "*", трансляция белка на нем не заканчивается.
На рис. 5 представлен фрагмент записи Genbank о гене с координатами 72210:73130(-). Здесь указано, что на данном участке имеется внутренний стоп-кодон, и что этот ген является псевдогеном. То есть он является нефункциональным из-за мутации, которая привела к появлению стоп-кодона. Также здесь указано, что это ген был предсказан на основе гомологии белков. Таким образом, возможно, в действительности результатом трансляции данного участка являются 2 белка, как и предсказал Prodigal.
Задание 2
Аналогичные действия были проведены для геномов Methanosarcina acetivorans (NC_003552.1) и Escherichia coli (NC_000913).
Краткие сведения об их геномах и предсказанных генах представлена в табл.2.
| Параметр | M. acetivorans | E. coli |
| Размер генома, bp | 5751492 | 4641652 |
| Количество генов(Genbank) | 4856 | 4518 |
| Количество генов(Prodigal) | 4885 | 4318 |
На рис. 6, 7 представлены показатели сравнения генов, аннотированных в Genbank и предсказанных Progital для M. acetivorans и E. coli, соответственно.
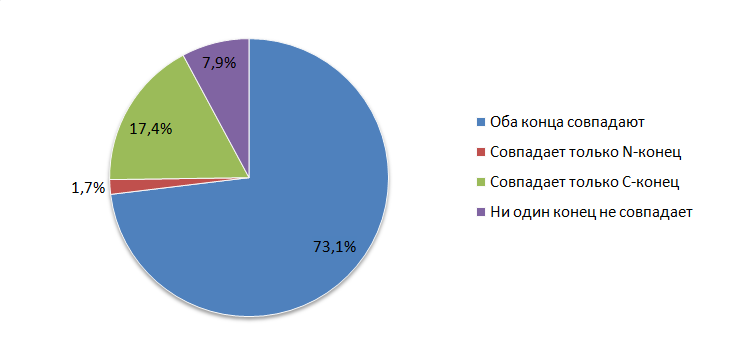
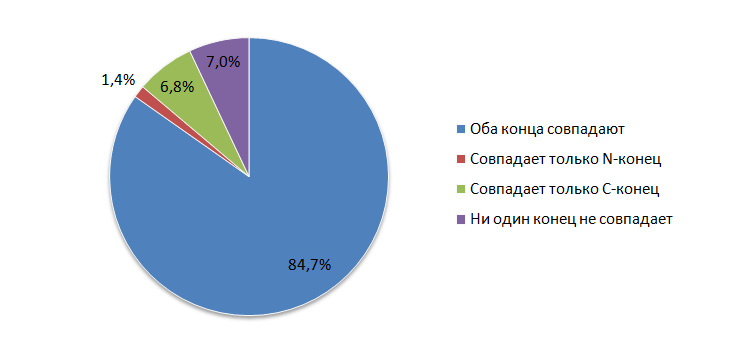
Исходя из полученных данных можно сделать следующие выводы:
Ссылки
1.Wikipedia: Lactobacillus_plantarum
2. Degradation of Raw Starch by a Wild Amylolytic Strain of Lactobacillus plantarum APPLIED AND ENVIRONMENTAL MICROBIOLOGY, Dec. 1994, p. 4319-4323.